Mapping of QTLs for root morphology and nitrogen uptake of maize under different nitrogen conditions
-
摘要:目的玉米的根系形态与氮素吸收能力关系密切,利用单片段代换群体对玉米苗期根系形态相关性状和植株氮吸收量进行 QTL 定位,可为进一步精细定位并克隆控制玉米低氮下优异根系形态和氮吸收的主效 QTL 奠定基础。方法以氮效率差异显著的两亲本许 178 和综 3 构建的 150 个玉米单片段代换系 (SSSL) 群体作为研究材料,进行水培试验。以 Ca (NO3)2 作为氮源,设置高氮 (4 mmol/L NO3– )和低氮 (0.05 mmol/L NO3– ) 两个处理,培养 20 d 后分根、冠收获植株,测定生物量和氮含量。通过 WinRHIZO 根系分析系统获得根系的总根长、根表面积、根体积、根直径和根尖数等指标。根据代换系与亲本许 178 表型值的 T-test 结果,利用该群体 SSR 遗传连锁图谱,在 P ≤ 0.001 条件下定位所调查性状的 QTL。结果高氮条件下 SSSL 群体除了根直径与总根长和根尖数没有显著相关性以外,其它各性状之间均显著或极显著正相关,并且植株氮吸收量也与根系各性状呈显著或极显著正相关;低氮条件下,除了根直径以外,植株氮吸收量与其他根系性状均呈极显著正相关,并且地上部和根部氮累积量均与根表面积的相关性最大。在高氮条件下共检测到 102 个 QTL 位点,包括 40 个根形态相关 QTL、34 个植株生物量 QTL 和 28 个氮吸收量 QTL;在低氮条件下共检测到 85 个 QTL 位点,包括 47 个根形态 QTL、22 个植株生物量 QTL 和 16 个氮吸收量 QTL。所检测到的根形态相关 QTL 与生物量和氮积累量 QTL 成簇存在,同一 QTL 区间多同时检测到根形态 QTL 和氮吸收量 QTL。高氮条件下,在代换系 1428、1376、1282、1266 和 1473 的代换区间上检测到高氮特异的 QTL 簇,同时包括多个根形态和氮吸收量 QTL,贡献率从–43% 到 84%。低氮下,在代换系 1419 和 1314 的代换区间上同时检测到低氮特异的多个根形态和氮吸收量 QTL,贡献率从–32% 到 55%。结论单片段代换系 1419 和 1314 所包含的代换片段 bnlg182—bnlg2295 和 umc1013—umc2047 检测到多个低氮特异的 QTL,并且这两个区间在前人的研究中均有玉米氮效率相关 QTL 检测到,说明该区间包含有玉米根系形态和氮吸收量的主效 QTL,在玉米氮高效吸收中可能起重要作用。Abstract:ObjectivesThere is a strong relationship between maize root morphology and nitrogen uptake capacity. In this study, QTLs for maize root morphology and plant nitrogen uptake were identified using single segment substitution lines (SSSLs) to provide support for fine mapping and cloning of major QTLs controlling maize root morphology and nitrogen uptake.Methods150 maize SSSLs derived from a cross between a N-efficient inbred line Xu178 and a N-inefficient inbred line Zong 3 were used for solution culture. Ca(NO3)2 was used as nitrogen source and high nitrogen level (4 mmol/L NO3–) and low nitrogen level (0.05 mmol/L NO3–) were supplied for each line, and each N level had six seedlings. After 20 days culture, seedlings were harvested, and the biomass and nitrogen contents of shoots and roots were analyzed respectively. Total root length (TRL), root surface area (RSA), root volume (RV), root average diameter (RAD) and root tip numbers (RTN) were determined from the root images using WinRHIZO. According to the results of the T-test of the phenotype values of SSSL and Xu178, QTLs for each trait were mapped in the SSR genetic linkage map when P value was less than 0.001.ResultsUnder the high N level, all root traits were significantly correlated with each other except that between RAD and either TRL or RTN, and the plant nitrogen uptake was significantly correlated with all the root morphology related traits. Under the low N level, all the root morphology traits were strongly correlated with plant nitrogen uptake except for RAD, and the RSA showed the most significant correlation. Under the high N level, 102 QTLs were detected including 40 QTLs for root morphology traits, 34 QTLs for plant biomass and 28 QTLs for plant nitrogen uptake. Under the low N level, 85 QTLs were detected including 47 QTLs for root morphology traits, 22 QTLs for plant biomass and 16 QTLs for plant nitrogen uptake. Most of the QTLs for N uptake coincided in cluster with those for root morphology. Several QTLs for root morphology and nitrogen uptake were mapped in the same substituted segment region. Under the high N condition, five high N-specific QTLs clusters containing several root morphology traits and nitrogen uptake were detected in SSSL lines of 1428, 1376, 1282, 1266 and 1473. The single QTL additive effect contribution was from –43% to 84%. Moreover, several QTLs for root morphology and nitrogen uptake were identified in the SSSL lines of 1419 and 1314 under the LN condition, with the additive effect contribution from –32% to 55%.ConclusionsIn the present study, several LN-specific QTLs were mapped in substituted segment of bnlg182–bnlg2295 in line 1419 and umc1013–umc2047 in line 1314, in which some QTLs related to nitrogen use efficiency of maize were detected in previous researches. It was indicated that there were some major QTLs for maize root morphology and plant nitrogen uptake located in the two regions which would play important role in maize nitrogen uptake efficiency. The present research serves as a basis for the major QTLs fine-mapping and candidate genes cloning.
-
Keywords:
- maize /
- SSSLs /
- root morphology /
- QTL /
- nitrogen deficiency
-
玉米是全球最重要的多用途作物之一,广泛应用于粮食、饲料、化工、生物能源等行业。为保证国家粮食安全,我国在《国家粮食安全中长期规划纲要》中明确提出到 2020 年需要新增粮食 1000 亿斤,其中需要新增玉米 650 亿斤,占全国新增粮食的 65%,因此玉米在我国未来粮食生产中的地位举足轻重。我国玉米高产育种已取得了重大成就,超高产记录不断突破。不容忽视的是,目前的玉米高产记录都是高肥高水条件下获得,同样为提高玉米产量,我国农田的氮肥投入量大、氮肥利用率低,不但严重制约我国玉米产量和农业经济效益的进一步提高,而且成为导致河流和湖泊富营养化的重要因素之一[1]。因此,提高玉米的氮素利用率、培育氮高效品种,从而在适当降低施氮量的条件下维持较高产量水平,对实现农业可持续发展和环境保护意义重大[2]。
研究表明,在玉米籽粒形成中 54.5%~60.6% 的氮来自营养体的再运转[3]。因此,植株前期氮吸收效率对于后期提高玉米籽粒产量至关重要。植物主要通过根系从外界吸收矿质营养,有效的根系形态对培育氮吸收高效的玉米基因型、减少氮淋失非常重要[4]。许多研究表明,增加根系体积,包括根干重、根长和根密度,可以提高玉米氮吸收能力和籽粒产量[5–6]。玉米的根系形态受土壤有效氮含量的强烈影响,植株通过改变根系形态更有效地从土壤中获取氮素营养。在低氮条件下,玉米的节根数目减少、总根长增加,而在氮充足的土壤侧根易发生和伸长[7–8]。因此,可以利用玉米根系形态对土壤氮水平响应的可塑性来提高玉米的氮吸收效率。目前数量性状位点 (QTL) 作图已成为研究复杂性状遗传位点甚至确定候选基因的有力工具。许多与玉米氮效率相关性状的QTL已经被定位,包括氮吸收效率、氮利用效率[9]、花后氮吸收、氮再利用[1],以及氮同化、代谢等性状[9]。同样,诸多控制玉米根系形态的 QTL 也在不同的群体中被定位和分析[10–14]。但是,这些研究多数都是对正常生长条件下玉米根系形态相关性状进行 QTL 定位,对低氮胁迫下玉米根系形态相关性状的 QTL 定位相对缺乏。
刘宗华等[15]前期研究结果表明,在大田不施氮肥条件下,综 3 的产量降低了 54%,而许 178 的产量仅降低了 1.2%,表明许 178 对低氮胁迫不敏感,而综 3 对低氮胁迫表现敏感。本研究以综 3 为供体亲本,许 178 为轮回亲本构建的染色体单片段代换系 (single segment substitution lines, SSSLs)[16]作为研究材料,通过营养液培养对群体在高氮和低氮条件下根系形态相关性状,以及氮吸收量的 QTL 进行了定位。研究分析了低氮胁迫下玉米苗期根形态与氮吸收效率之间的遗传机制,为通过分子标记辅助选择培育低氮下具有优异根系形态的玉米基因型提供参考。
1. 材料与分析
1.1 供试材料
以综 3 为供体亲本、许 178 为受体亲本,经过 4 个世代回交和 3 个世代自交,结合 SSR 分子标记辅助选择,共获得了 239 份纯合的单片段代换系材料,SSSL 平均代换片段长度为 47.67 cM,导入片段总长 11394.22 cM,覆盖玉米基因组 67.6%[15]。利用其中的 150 个单片段代换系进行玉米苗期不同氮水平下根系形态 QTL 检测,同时结合各株系表型值及片段所在位置,对相应根系形态 QTL 进行初步定位。
1.2 幼苗培养
取 150 个单片段代换系及亲本各 30 粒饱满完整的种子,以 10% H2O2 浸泡消毒 30 min,蒸馏水冲洗 3~4 次,置于湿润的纱布上催芽,然后移入干净的石英砂中育苗,光照培养室日间温度为 25℃,夜间温度为 20℃。用蒸馏水培养至两叶,挑取发育健壮、长势一致的幼苗共 12 株,分别移入低氮和高氮营养液中培养,每个氮水平 6 株。幼苗定植于穿孔直径为 2 cm、厚 0.5 cm 的塑料板上,用海绵固定幼苗,培养容器采用黑色方形塑料盒 (38 cm × 28 cm × 12 cm),每盆培养幼苗 12 株,内盛营养液 10 L,3 天更换一次营养液。营养液配方参考文献[17],具体如下:Ca (NO3)2·4H2O 2 mmol/L;K2SO4 0.75 mmol/L;MgSO4·7H2O 0.65 mmol/L;KCl:0.1 mmol/L;KH2PO4 0.25 mmol/L;H3BO3 0.001 mmol/L;MnSO4·H2O 0.001 mmol/L;CuSO4·5H2O 0.0001 mmol/L;ZnSO4·7H2O 0.001 mmol/L;(NH4)6MoO24·4H2O 5 × 10–6mmol/L;FeSO4·7H2O 0.1 mmol/L;Na2EDTA 0.1mmol/L;CaCl2 2 mmol/L (低氮处理时加入,用以补平 Ca 离子浓度)。其中低氮处理氮浓度为 0.05 mmol/L,高氮水平含氮 4 mmol/L,共培养 20 天。
1.3 根系性状的测定
培养 20 天后,每个重复取生长一致的 4 株幼苗,蒸馏水冲洗干净,用剪刀剪下根系,WinRHIZO 根系分析系统分别测定单株玉米的总根长 (TRL, cm)、根尖数 (RTN)、根体积 (RV, cm3)、根表面积 (RSA, cm2)、根直径 (RAD, mm)。将新鲜根和茎叶分别置于 105℃ 烘箱杀青 30 min,然后 80℃ 烘干,测定地上部干重 (SDW)、根干重 (RDW)。
1.4 氮含量的测定
植株烘干粉碎后,采用 H2SO4–H2O2 进行消化,通过流动注射仪测定氮含量。植株氮累积量计算:
地上部氮累积量 (SNU,mg) = 地上部干重 (g) × 地上部氮含量 (mg/g);
根氮累积量 (RNU,mg) = 根部干重 (g) × 根部氮含量 (mg/g)。
1.5 QTL 定位
利用 SPSS19.0 软件对各染色体片段代换系和双亲本根系相关数据进行统计分析。将轮回亲本的观察值作为对照,通过方差分析和 T 检验比较各 SSSL 与许 178 之间的差异,以 P ≤ 0.001 时,认为该代换片段上有相关 QTL 存在;当 P > 0.001 时,认为在该代换片段上没有相关 QTL 的存在[18]。参照 Eshed 和 Zamir 的方法[19]估算各 QTL 的加性效应值和加性效应贡献率,加性效应值 = (SSSL 表型值 – 许 178 表型值)/2,加性效应贡献率 = 加性效应值/许 178 表型值 × 100%。
QTL 命名遵循 Mc Couch 等[20]制定的原则,QTL 名称中前面的字母为鉴定性状的英文词首的缩写,第 1 个数字为 QTL 所在染色体的编号,第 2 个数字为该性状在该染色体上的 QTL 序号。
1.6 QTL 代换作图
参照 Paterson 等[20]的方法,如果在含有重叠代换片段的不同染色体片段代换系中同时检测到同一性状的 QTL,且遗传效应方向一致,则认为该 QTL 存在于代换片段的重叠区段上;如果在一个染色体片段代换系中检测到 QTL 的存在,而在代换片段具有重叠关系的另一个染色体片段代换系中未检测到,则认为该 QTL 位于这 2 个代换系代换片段的非重叠区段上。
1.7 数据处理
采用 Microsoft Excel 2010 和 SPSS 19.0 软件对数据进行整理和统计分析。
2. 结果与分析
2.1 不同氮水平下亲本和 SSSL 群体各表型性状值分析
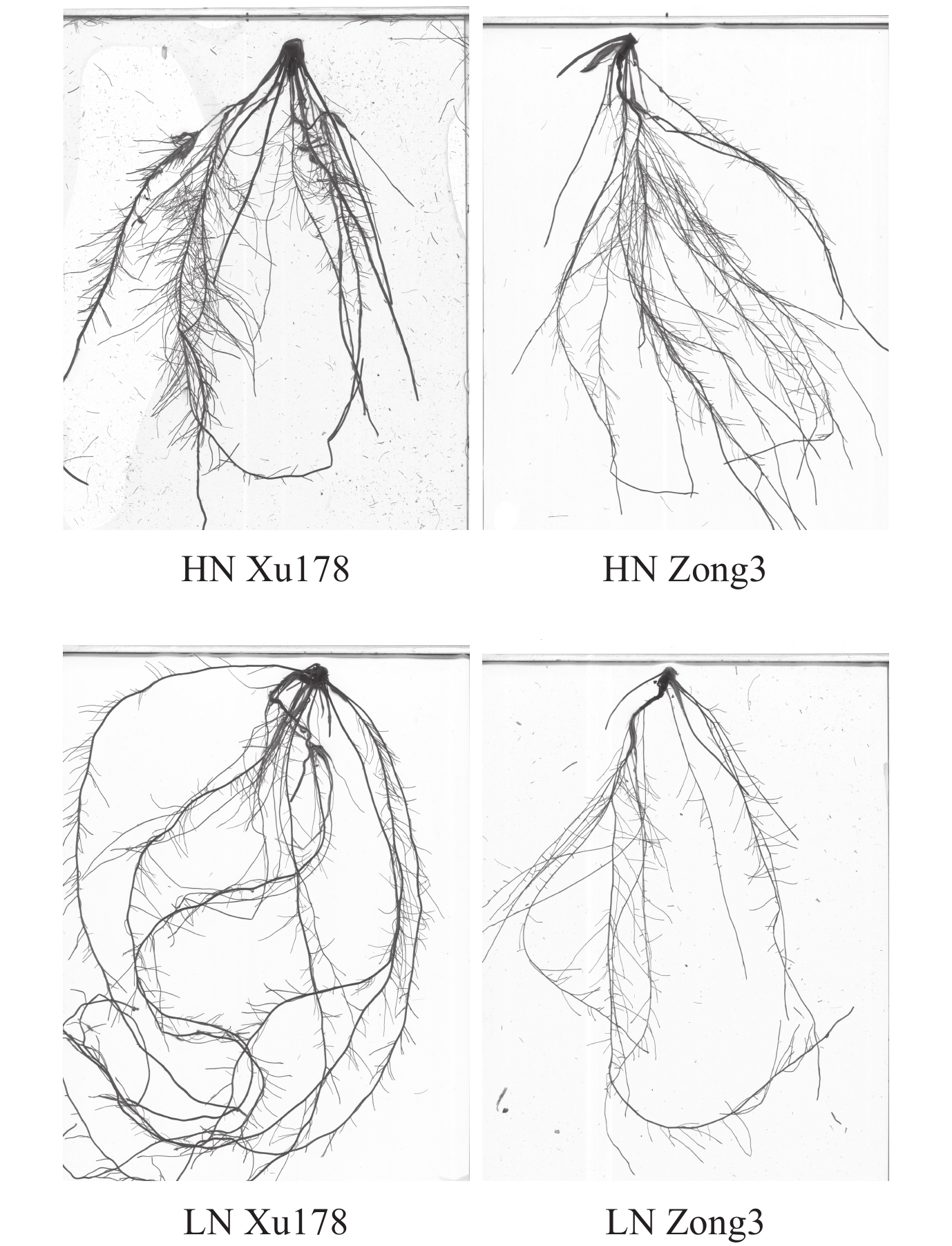
高氮条件下,亲本许 178 和综 3 的根系形态并没有显著差异,但是低氮胁迫显著增加了许 178 的根长,其根系远大于综 3 (图 1),表明在低氮条件下许 178 具有明显的根系优势。
数据统计结果显示,高氮下两亲本除了根尖数有显著差异外,其他根系性状均无显著差异 (表 1)。低氮条件下,氮高效亲本许 178 的生物量显著高于综 3,并且许 178 的根系性状 (总根长、根表面积、根体积、根直径和根尖数) 大于综 3,最终导致许 178 植株总氮吸收量是综 3 的 2.3 倍。对亲本 9 个性状进行双因素主效应分析发现,除了根直径外,其他 8 个性状氮水平的主效应差异均达到显著或极显著差异,而两亲本的基因型差异在总根长、根表面积和根体积上没有达到显著水平,但是除了根直径外,氮水平和基因型的交互作用在两亲本间的差异均达到显著或极显著水平 (表 1)。
表 1 不同氮水平下亲本各表型值和双因素显著性分析Table 1. Mean values and significance of two-factor ANOVA analysis for the nine traits in the parents性状
Trait高氮 High-N 低氮 Low-N 显著性 Significance 许 178
Xu178综 3
Zong3许 178
Xu178综 3
Zong3氮水平 (N)
N level基因型 (G)
Genotype交互作用
N × G总根长 TRL 828 1082 784 378 ** ns ** 根表面积 RSA 126 145 121 64.9 ** ns ** 根直径 RAD 0.51 0.43 0.50 0.40 ns *** ns 根体积 RV 1.23 1.50 1.37 0.64 * ns ** 根尖数 RTN 2190 1245 717 641 *** ** ** 地上部干重 SDW 0.81 0.77 0.23 0.15 *** *** *** 根干重 RDW 0.26 0.23 0.16 0.09 *** *** *** 地上部氮累积量 SNU 28.58 23.88 2.38 1.13 *** ** * 根氮累积量 RNU 4.54 4.85 1.38 0.52 *** * *** 注(Note):TRL—总根长 Total root length; RSA—根表面积 Root surface area; RAD—根直径 Root average diameter; RV—根体积 Root volume; RTN—根尖数 Root tip number; SDW—地上部干重 Shoot dry weight; RDW—根干重 Root dry weight; SNU—地上部氮累积量 Shoot nitrogen uptake; RNU—根部氮累积量 Root nitrogen uptake; G—基因型 Genotype; N × G—氮水平 × 基因型 N level × genotype; ns—无显著性差异 Not significant; ***—P ≤ 0.001; **—0.001 < P ≤ 0.01; *—0.01 < P ≤ 0.05. 在两个氮水平下,SSSL 群体的 9 个性状均表现连续变异,接近正态分布 (表 2)。这些性状的变异系数从 5.6%~33.9%。根直径的变异系数较小,其他性状的变异系数均大于 20%。在两个氮水平下,每个性状均观察到超亲现象。在 SSSL 群体中,基因型对 9 个性状的主效应作用均达到显著或极显著差异。除了根干重外,氮水平及其与基因型的交互作用对其他 8 个性状的作用均达到显著水平。这些结果表明,研究所选用的 SSSL 群体对氮胁迫敏感,并且群体基因型差异性显著,有利于我们利用该群体对根性状和氮吸收相关 QTL 进行定位和进一步深入分析。
表 2 不同氮水平下 SSSL 群体各表型值和双因素显著性分析Table 2. Mean values and significance of two-factor ANOVA analysis for the nine traits in the SSSL population性状
Trait高氮 High-N 低氮 Low-N 显著性 Significance 均值
Mean范围
Range变异 (%)
CV均值
Mean范围
Range变异 (%)
CV氮水平 (N)
N level基因型 (G)
Genotype交互作用
N × G总根长 TRL 1224 604~2558 22.1 986 297~1439 21.5 *** *** *** 根表面积 RSA 219 103~338 21.8 150 47.83~266 23.9 *** *** *** 根直径 RAD 0.57 0.45~0.69 8.4 0.49 0.41~0.56 5.6 *** *** *** 根体积 RV 3.09 0.91~4.57 26.6 1.87 0.61~3.19 25.7 *** *** *** 根尖数 RTN 1758 738~3748 33.9 919 466~1725 26.9 *** *** *** 地上部干重 SDW 1.26 0.55~1.88 21.4 0.26 0.09~0.42 24.1 *** *** *** 根干重 RDW 0.32 0.11~0.52 27.2 0.2 0.07~0.32 21.3 ns ** ns 地上部氮累积量 SNU 38.25 13.57~64.11 24.8 2.83 1.08~4.78 21.8 *** *** *** 根氮累积量 RNU 6.62 2.38~10.23 27.0 1.34 0.63~2.31 26.9 *** * * 注(Note):TRL—总根长 Total root length; RSA—根表面积 Root surface area; RAD—根直径 Root average diameter; RV—根体积 Root volume; RTN—根尖数 Root tip number; SDW—地上部干重 Shoot dry weight; RDW—根干重 Root dry weight; SNU—地上部氮累积量 Shoot nitrogen uptake; RNU—根部氮累积量 Root nitrogen uptake; ns—无显著性差异 not significant; ***—P ≤ 0.001; **—0.001 < P ≤ 0.01; *—0.01 < P ≤ 0.05. 2.2 不同氮水平下 SSSL 群体各表型性状的相关性分析
高氮条件下 SSSL 群体除了根直径与总根长和根尖数没有显著相关性以外,其它各性状之间均呈显著或极显著正相关,并且各根系性状与植株生物量均呈极显著正相关,植株氮吸收量也与根系各性状呈显著或极显著正相关 (表 3)。在低氮条件下,根直径与总根长、根表面积、地上部生物量之间均没有显著相关性,其他根系性状之间均显著相关。此外,低氮条件下,除了根直径,植株氮吸收量与其他根系性状均呈极显著正相关性。在两个氮水平下,均是根体积与根表面积的相关性最大 (高氮条件 r = 0.913,P < 0.001;低氮条件 r = 0.927,P < 0.001)。高氮条件下,地上部氮累积量与根表面积相关性最高,根系氮累积量则与根体积的相关性最高,而在低氮条件下,地上部和根系氮累积量均与根表面积的相关性最大。
表 3 群体各表型性状在高氮水平 (对角线上方) 和低氮水平 (对角线下方) 的 Pearson 相关系数Table 3. Pearson’s correlation coefficients between the traits under high N (above diagonal) and low N conditions (below diagonal) in the SSSL populationTRL RSA RAD RV RTN SDW RDW SNU RNU TRL 0.913*** –0.049 0.780*** 0.454*** 0.767*** 0.738*** 0.616*** 0.585*** RSA 0.872*** 0.228** 0.934*** 0.392*** 0.840*** 0.838*** 0.661*** 0.674*** RAD –0.129 0.118 0.434*** –0.148 0.299** 0.393*** 0.174* 0.330*** RV 0.795*** 0.927*** 0.301*** 0.271** 0.827*** 0.854*** 0.633*** 0.704*** RTN 0.476*** 0.417*** –0.179* 0.352*** 0.279** 0.289** 0.203* 0.246** SDW 0.698*** 0.752*** 0.093 0.726*** 0.458*** 0.880*** 0.682*** 0.725*** RDW 0.792*** 0.840*** 0.169* 0.835*** 0.425*** 0.831*** 0.676*** 0.760*** SNU 0.521*** 0.542*** 0.054 0.536*** 0.387*** 0.707*** 0.599*** 0.701*** RNU 0.388** 0.456*** 0.033 0.446*** 0.328*** 0.430*** 0.422*** 0.394*** 注(Note):TRL—总根长 Total root length; RSA—根表面积 Root surface area; RAD—根直径 Root average diameter; RV—根体积 Root volume; RTN—根尖数 Root tip number; SDW—地上部干重 Shoot dry weight; RDW—根干重 Root dry weight; SNU—地上部氮累积量 shoot nitrogen uptake; RNU—根氮累积量 Root nitrogen uptake. ***—P ≤ 0.001; **—0.001 < P ≤ 0.01; *—0.01 < P ≤ 0.05. 2.3 不同氮条件下玉米根系形态 QTL 分析
通过 QTL 代换作图,对 SSSL 群体检测到的根系形态 QTL 进行分析。高氮水平下,在 P ≤ 0.001 条件下共检测到 40 个根系形态 QTL,其中 8 个总根长 QTL,7 个根表面积 QTL,4 个根直径 QTL,10 个根体积 QTL 和 11 个根尖数 QTL (表 4)。总根长 QTL 主要分布在第 1、3、5、7、9 和 10 号染色体,第 1 染色体最多,其中 qhnTRL1b 表现为负向加性效应,其余均表现为正向加性效应。代换系 1306 包含的 qhnTRL1c 加性效应贡献率最大 (59.8%)。检测到根表面积 QTL 与总根长类似,仅有一个 QTL 表现负向加性效应,其余均为正向,加性效应贡献率为–23.5%~83.7%。检测到的根直径 QTL 的数量最少,均为正向加性效应,贡献率从 14.1%~17.4%。检测到的根体积的 QTL 均为正向加性效应,且加性效应贡献率很高,介于 79.4%~125.1%,这表明在高氮水平下综 3 片段的导入可能有利于代换系获得更大的根系体积。与其他根系性状不同,检测到的 10 个根尖 QTL 中有 9 个表现负向加性效应,贡献率为–23.2%~–36.5%,只有 qhnRTN7a 为正向加性效应。
表 4 高氮下玉米 SSSL 群体的根系形态 QTL 及其效应值Table 4. QTL location of the maize root morphology in the SSSL population under the high nitrogen condition性状
TraitQTL 代换系
SSSLBin 代换片段
Substituted segment片段长度 (cM)
Segment length加性效应
AE加性效应贡献率
AEC (%)总根长
TRLqhnTRL1a 1339 1.01 end—umc1269 81.53 392.54 47.4 qhnTRL1b 1468 1.03 phi001—bnlg182 7.60 –220.68 –26.6 qhnTRL1c 1306 1.05 umc1461—umc1124 9.10 495.78 59.8 qhnTRL3a 1481 3.02 bnlg1144—umc1425 44.00 351.43 42.4 qhnTRL5 1293 5.02 umc1587—bnlg118 139.10 360.79 43.6 qhnTRL7 1343 7.00 umc1642—umc1929 110.95 455.78 55.0 qhnTRL9 1327 9.04 umc1771—umc1519 2.26 297.66 35.9 qhnTRL10 1255-1 10.03 phi050—umc1506 48.50 326.49 39.4 根表面积
RSAqhnRSA1a 1468 1.03 phi001—bnlg182 7.60 –29.73 –23.5 qhnRSA1b 1376 1.06 umc1122—umc1013 61.26 105.87 83.7 qhnRSA4 1350 4.08 bnlg1444—umc1109 176.54 65.78 52.0 qhnRSA6 1282 6.00 bnlg1043—umc1883 20.09 73.70 58.3 qhnRSA7 1365 7.00 umc1241—umc1585 236.47 66.07 52.3 qhnRSA8 1485 8.04 bnlg2046—umc1724 93.52 67.33 53.3 qhnRSA9 1266 9.01 umc1867—umc1170 38.40 75.27 59.5 根直径
RADqhnRAD1 1461 1.05 umc1703—bnlg1598 37.90 0.09 17.4 qhnRAD6 1469 6.00 bnlg238—umc1883 16.29 0.08 16.2 qhnRAD8 1485 8.04 bnlg2046—umc1724 93.52 0.07 14.1 qhnRAD10 1473 10.04 umc1077—umc2350 15.10 0.08 16.2 根体积
RVqhnRV1b 1306 1.05 umc1461—umc1124 9.10 1.38 112.7 qhnRV2 1325 2.04 umc1541—phi083 4.90 0.97 79.4 qhnRV4 1349 4.08 bnlg1444—umc2041 9.35 1.24 100.8 qhnRV5 1321 5.02 umc1587—umc2072 138.95 1.21 98.9 qhnRV7a 1365 7.00 umc1241—umc1585 236.47 1.33 108.2 qhnRV7b 1267 7.05 bnlg1805—phi069 76.55 1.53 125.1 qhnRV8 1485 8.04 bnlg2046—umc1724 93.52 1.51 123.3 qhnRV9 1266 9.01 umc1867—umc1170 38.40 1.51 123.6 低氮水平下,通过 QTL 代换作图,在 P ≤ 0.001 条件下共检测到 47 个根系形态 QTL,其中 10 个总根长 QTL,5 个根表面积 QTL,5 个根直径 QTL,12 个根体积 QTL 和 15 个根尖数 QTL (表 5)。其中检测到的总根长和根表面积 QTL 与高氮条件类似,多为正向加性效应,各有一个 QTL 为负向加性效应。与高氮条件不同,低氮下检测到的根直径 QTL 均为负向加性效应,加性效应贡献率为–5.0%~–8.9%。低氮下检测到的根尖数 QTL 多为正向加性效应,仅 qlnRTN4b 表现为负向加性效应,并且加性效应的贡献率低于高氮条件,这可能与低氮胁迫下植物根系的适应性反应有关。检测到的 12 个根体积 QTL 中有 3 个为负向加性效应,其中在第 1 染色体检测到 5 个 QTL。
表 5 低氮下玉米 SSSL 群体的根系形态 QTL 及其效应值Table 5. QTL location of the root morphology in the SSSL population under the low nitrogen condition性状
TraitQTL 代换系
SSSLBin 代换片段
Substituted segment片段长度 (cM)
Segment length加性效应
AE加性效应贡献率
AEC (%)总根长
TRLqlnTRL1a 1339 1.01 end—umc1269 81.53 281 35.8 qlnTRL1b 1394 1.02 bnlg1007—phi095 50.50 328 41.8 qlnTRL1c 1314 1.08 umc1013—umc2047 59.95 –250 –31.9 qlnTRL4a 1374 4.05 umc1662—bnlg1444 141.14 242 30.8 qlnTRL4b 1296 4.11 phi076—end 25.10 160 20.5 qlnTRL5 1328 5.02 umc1587—umc1680 213.20 493 62.9 qlnTRL6 1448 6.04 umc1614—umc1462 43.35 226 28.9 qlnTRL7 1277 7.00 umc1241—umc1642 13.60 305 39.0 qlnTRL9 1241 9.05 umc1078—phi236654 28.66 150 19.2 qlnTRL10 1240 10.06 bnlg1028—bnlg2190 15.90 247 31.5 根表面积
RSAqlnRSA1a 1394 1.02 bnlg1007—phi095 50.50 51.19 42.2 qlnRSA1b 1419 1.03 bnlg182—bnlg2295 55.50 44.53 36.7 qlnRSA1c 1314 1.08 umc1013—umc2047 59.95 –34.52 –28.5 qlnRSA5 1328 5.02 umc1587—umc1680 213.20 72.45 59.7 qlnRSA7 1382 7.01 umc2160—umc1585 73.17 42.70 35.2 根直径
RADqlnRAD1a 1339 1.01 end—umc1269 81.53 –0.04 –7.3 qlnRAD1b 1415 1.03 bnlg182—bnlg2295 55.50 –0.02 –5.0 qlnRAD3b 1423 3.02 bnlg1144—umc1425 44.00 –0.04 –7.3 qlnRAD3a 1341 3.09 umc1052—end 8.56 –0.04 –8.9 qlnRAD7a 1382 7.01 umc2160—umc1585 73.17 –0.03 –5.4 根体积
RVqlnRV1a 1437 1.01 umc1269—bnlg1007 52.12 –0.37 –26.7 qlnRV1b 1394 1.02 bnlg1007—phi095 50.50 0.70 51.1 qlnRV1c 1419 1.03 bnlg182—bnlg2295 55.50 0.65 47.7 qlnRV1d 1403 1.05 umc2025—umc1689 11.06 0.50 36.4 qlnRV1e 1314 1.08 umc1013—umc2047 59.95 –0.35 –25.6 qlnRV3 1423 3.02 bnlg1144—umc1425 44.00 –0.20 –14.3 qlnRV4 1313 4.08 bnlg1444—umc2041 9.35 0.57 41.7 qlnRV5 1328 5.02 umc1587—umc1680 213.20 0.91 66.3 qlnRV6 1276 6.00 umc1883—umc1257 47.48 0.53 38.4 qlnRV7 1239 7.05 bnlg1805—phi069 76.55 0.60 44.1 qlnRV9 1462 9.02 umc1170—bnlg1401 77.90 0.61 44.3 qlnRV10 1388 10.06 umc1993—umc1569 47.45 0.77 55.9 根尖数
RTNqlnRTN1a 1419 1.03 bnlg182—bnlg2295 55.50 395 55.0 qlnRTN1b 1479 1.06 bnlg1598—umc2396 35.80 384 53.6 qlnRTN2 1366 2.06 bnlg1396—umc2129 18.86 275 38.3 qlnRTN3a 1457 3.02 bnlg1144—umc1425 44.00 205 28.6 qlnRTN3b 1486 3.08 umc1844—bnlg1182 60.24 203 28.3 qlnRTN4a 1313 4.08 bnlg1444—umc2041 9.35 426 59.4 qlnRTN4b 1292 4.09 umc1989—phi076 90.99 –125 –17.5 qlnRTN4c 1296 4.11 phi076—end 25.10 240 33.5 qlnRTN5 1328 5.02 umc1587—umc1680 213.20 222 31.0 qlnRTN6 1469 6.00 bnlg238—umc1883 16.29 217 30.3 qlnRTN7a 1277 7.00 umc1241—umc1642 13.60 349 48.7 qlnRTN7b 1382 7.01 umc2160—umc1585 73.17 504 70.3 qlnRTN8 1480 8.06 umc1724—phi015 57.58 433 60.4 qlnRTN10a 1324 10.03 umc1739—umc1053 25.80 230 32.0 qlnRTN10b 1395 10.06 bnlg2190—end 46.60 234 32.7 注(Note):TRL—total root length; RSA—Root surface area; RAD—Root average diameter; RV—Root volume; RTN—Root tip number; QTL—数量性状位点 Quantitative trait locus; AE—Additive effect; AEC—Additive effect contribution; AEC (%) = (SSSL 表型值 – 许 178 表型值)/(2 × 许 178 表型值) × 100, AEC (%) = (SSSLs – Xu178)/(2 × Xu178) × 100. 2.4 不同氮条件下生物量 QTL 分析
高氮条件下,通过 QTL 代换作图在 P ≤ 0.001 时,SSSL 群体共检测到 20 个地上部干重 QTL 和 14 个根干重 QTL (表 6)。20 个地上部干重 QTL 分布在除第 8 染色体的其余 9 条染色体上。其中 qhnSDW1b 表现为负向加性效应,其余 QTL 均表现为正向加性效应,单个 QTL 的加性效应贡献率为–40.1%~88.3%。14 个根干重 QTL 分布在除第 2、8 染色体外的其他 8 条染色体上,其中有 4 个表现负向加性效应,其他 10 个 QTL 为正向加性效应。单个 QTL 的加性效应贡献率为–10.2%~42.8%。
表 6 高氮条件下玉米苗期生物量 QTL 及其效应Table 6. QTL location of plant biomass in the SSSL population under the high nitrogen condition性状
TraitQTL 代换系
SSSLBin 代换片段
Substituted segment片段长度 (cM)
Segment length加性效应
AE加性效应贡献率
AEC (%)地上部干重
SDWqhnSDW1a 1279 1.01 umc1106—phi427913 29.58 0.28 35.0 qhnSDW1b 1428 1.03 phi001—bnlg182 7.60 –0.32 –40.1 qhnSDW1c 1306 1.05 umc1461—umc1124 9.10 0.35 43.8 qhnSDW1d 1459 1.06 umc2396—umc2047 148.92 0.51 63.8 qhnSDW2 1366 2.06 bnlg1396—umc2129 18.86 0.30 37.4 qhnSDW3a 1481 3.02 bnlg1144—umc1425 44.00 0.38 47.2 qhnSDW3c 1335 3.08 umc1844—bnlg1182 60.24 0.51 63.8 qhnSDW4a 1349 4.08 bnlg1444—umc2041 9.35 0.47 57.9 qhnSDW4b 1296 4.11 phi076—end 25.10 0.31 38.8 qhnSDW5a 1421 5.00 end—bnlg1006 50.10 0.53 65.7 qhnSDW5b 1293 5.02 umc1587—bnlg118 139.10 0.26 32.6 qhnSDW5c 1286 5.06 umc1680—bnlg118 39.10 0.26 32.7 qhnSDW6 1282 6.00 bnlg1043—umc1883 20.09 0.49 60.2 qhnSDW7a 1277 7.00 umc1241—umc1642 13.60 0.28 34.6 qhnSDW7b 1382 7.01 umc2160—umc1585 73.17 0.23 28.3 qhnSDW7c 1239 7.05 bnlg1805—phi069 76.55 0.26 31.9 qhnSDW9a 1266 9.01 umc1867—umc1170 38.40 0.28 35.0 qhnSDW9b 1241 9.05 umc1078—phi236654 28.66 0.38 47.5 qhnSDW10a 1436 10.04 umc2350—bnlg1028 48.50 0.54 66.8 qhnSDW10b 1388 10.06 umc1993—umc1569 47.45 0.71 88.3 根干重
RDWqhnRDW1a 1428 1.03 phi001—bnlg182 7.60 –0.04 –29.4 qhnRDW1b 1297 1.05 umc1461—umc1124 9.10 0.10 38.2 qhnRDW1c 1376 1.06 umc1122—umc1013 61.26 0.11 41.4 qhnRDW3a 1455 3.01 umc2377—bnlg1647 27.43 –0.01 –10.2 qhnRDW3b 1481 3.02 bnlg1144—umc1425 44.00 0.09 32.7 qhnRDW4 1349 4.08 bnlg1444—umc2041 9.35 0.11 42.8 qhnRDW5a 1421 5.00 end—bnlg1006 50.10 0.09 33.2 qhnRDW5b 1328 5.06 umc1587—umc1680 213.20 –0.07 –24.8 qhnRDW6 1282 6.00 bnlg1043—umc1883 20.09 0.09 33.4 qhnRDW7 1267 7.05 bnlg1805—phi069 76.55 0.08 29.0 qhnRDW9a 1462 9.02 umc1170—bnlg1401 77.90 –0.05 –18.7 qhnRDW9b 1241 9.05 umc1078—phi236654 28.66 0.09 33.2 qhnRDW10a 1473 10.04 umc1077—umc2350 15.10 0.07 26.9 qhnRDW10b 1395 10.06 bnlg2190—end 46.60 0.11 40.3 注(Note):SDW—Shoot dry weight; RDW—Root dry weight; QTL—数量性状位点 Quantitative trait locus; AE—Additive effect; AEC—Additive effect contribution; AEC (%) = (SSSL 表型值 – 许 178 表型值)/(2 × 许 178 表型值) × 100, AEC (%) = (SSSLs – Xu178)/(2 × Xu178) × 100. 低氮条件下,通过 QTL 代换作图,在 P ≤ 0.001 条件下共检测到 22 个生物量 QTL,地上部干重 QTL 和根干重 QTL 均为 11 个 (表 7)。其中地上部干重 QTL 有 6 个分布在第 1 染色体,其余分布在第 3、4、5、7 和 10 号染色体上,加性效应贡献率为–24.5%~59.9%,qlnSDW5 的加性效应贡献率最高。与地上部干重 QTL 分布情况类似,检测到的根干重 QTL 也有 5 个分布于第 1 染色体,其余分布在第 2、3、4、5 和 7 号染色体上,单个 QTL 的加性效应贡献率为–19.2%~48.4%。
表 7 低氮条件下玉米苗期生物量 QTL 及其效应Table 7. QTL location of plant biomass in the SSSL population under the low nitrogen condition性状
TraitQTL 代换系
SSSLBin 代换片段
Substituted segment片段长度 (cM)
Segment length加性效应
AE加性效应贡献率
AEC (%)地上部干重 SDW qlnSDW1a 1437 1.01 umc1269—bnlg1007 52.12 –0.06 –24.5 qlnSDW1b 1394 1.02 bnlg1007—phi095 50.50 0.09 37.3 qlnSDW1c 1419 1.03 bnlg182—bnlg2295 55.50 0.08 35.2 qlnSDW1d 1383 1.05 umc1689—umc1703 8.94 0.08 35.9 qlnSDW1e 1314 1.08 umc1013—umc2047 59.95 –0.05 –20.1 qlnSDW3 1420 3.02 bnlg1144—umc1425 44.00 –0.02 –10.3 qlnSDW4a 1313 4.08 bnlg1444—umc2041 9.35 0.06 26.2 qlnSDW4b 1292 4.09 umc1989—phi076 90.99 –0.02 –9.3 qlnSDW5 1328 5.02 umc1587—umc1680 213.20 0.14 59.9 qlnSDW7 1277 7.00 umc1241—umc1642 13.60 0.08 34.4 qlnSDW10 1255-1 10.03 phi050—umc1506 48.50 0.09 39.0 根干重
RDWqlnRDW1a 1437 1.01 umc1269—bnlg1007 52.12 –0.03 –19.2 qlnRDW1b 1394 1.02 bnlg1007—phi095 50.50 0.07 41.8 qlnRDW1c 1419 1.03 bnlg182—bnlg2295 55.50 0.06 39.7 qlnRDW1d 1403 1.05 umc2025—umc1689 11.06 0.05 32.4 qlnRDW1e 1314 1.08 umc1013—umc2047 59.95 –0.03 –18.0 qlnRDW2 1361 2.07 umc2129—phi090 67.10 0.04 27.7 qlnRDW3a 1455 3.01 umc2377—bnlg1647 27.43 –0.02 –13.5 qlnRDW3b 1335 3.08 umc1844—bnlg1182 60.24 0.04 25.8 qlnRDW4 1301 4.08 bnlg1444—umc2041 9.35 0.04 23.3 qlnRDW5 1328 5.02 umc1587—umc1680 213.20 0.08 48.4 qlnRDW7 1382 7.01 umc2160—umc1585 73.17 0.04 27.7 注(Note):SDW—Shoot dry weight; RDW—Root dry weight; QTL—数量性状位点 Quantitative trait locus; AE—Additive effect; AEC—Additive effect contribution; AEC (%) = (SSSL 表型值 – 许 178 表型值)/(2 × 许 178 表型值) × 100, AEC(%) = (SSSLs – Xu178)/(2 × Xu178) × 100. 2.5 不同氮条件下植株氮累积量 QTL 分析
高氮条件下,通过 QTL 代换作图,在 P ≤ 0.001 条件下共检测到 13 个地上部氮累积量 QTL 和 15 个根部氮累积量 QTL (表 8)。这些 QTL 中只有 qhnSNU1b 和 qhnRNU1a 表现为负向加性效应。除第 3 和第 8 染色体外,其余染色体均检测到地上部氮累积量 QTL,单个 QTL 的加性效应贡献率为–42.7%~60.9%。根部氮累积量的 QTL 则在 10 条染色体上均有分布,其中第 1 和 9 染色体分别检测到 3 个 QTL,这些 QTL 的加性效应贡献率为–35.5%~71.7%。
表 8 高氮条件下苗期玉米植株氮累积量 QTL 分析Table 8. QTL location of the nitrogen accumulation in the SSSL population under the high nitrogen condition性状
TraitQTL 代换系
SSSLBin 代换片段
Substituted segment片段长度 (cM)
Segment length加性效应
AE加性效应贡献率
AEC (%)地上部氮累积量
SNUqhnSNU1a 1279 1.01 umc1106—phi427913 29.58 9.00 31.5 qhnSNU1b 1428 1.03 phi001—bnlg182 7.60 –12.25 –42.7 qhnSNU2a 1335 2.04 umc1541—umc2079 60.24 12.88 45.1 qhnSNU2b 1366 2.06 bnlg1396—umc2129 18.86 8.43 29.5 qhnSNU4 1349 4.08 bnlg1444—umc2041 9.35 10.91 38.2 qhnSNU5a 1421 5.00 end—bnlg1006 50.10 17.41 60.9 qhnSNU5b 1321 5.02 umc1587—umc2072 138.95 8.21 28.7 qhnSNU5c 1286 5.06 umc1680—bnlg1711 39.10 4.61 16.1 qhnSNU6 1282 6.00 bnlg1043—umc1883 20.09 12.55 43.9 qhnSNU7 1382 7.01 umc2160—umc1585 73.17 7.66 26.8 qhnSNU9 1238 9.02 umc2078—umc1636 34.20 7.09 24.8 qhnSNU10a 1255-1 10.03 phi050—umc1506 48.50 11.35 39.7 qhnSNU10b 1395 10.06 bnlg2190—end 46.60 11.49 40.2 根部氮累积量
RNUqhnRNU1a 1428 1.03 phi001—bnlg182 7.60 –1.61 –35.5 qhnRNU1b 1297 1.05 umc1461—umc1124 9.10 2.85 62.8 qhnRNU1c 1376 1.06 umc1122—umc1013 61.26 3.25 71.7 qhnRNU2 1366 2.06 bnlg1396—umc2129 18.86 1.85 40.7 qhnRNU3 1486 3.08 umc1844—bnlg1182 60.24 1.76 38.9 qhnRNU4 1349 4.08 bnlg1444—umc2041 9.35 1.91 42.2 qhnRNU5a 1283 5.00 umc1491—umc1478 90.00 1.36 30.0 qhnRNU5b 1321 5.02 umc1587—umc2072 138.95 1.47 32.5 qhnRNU6 1282 6.00 bnlg1043—umc1883 20.09 2.64 58.1 qhnRNU7 1239 7.04 bnlg1805—phi069 76.55 2.39 52.6 qhnRNU8 1427 8.06 umc1724—phi015 57.58 2.44 53.9 qhnRNU9a 1238 9.02 umc2078—umc1636 34.20 1.81 39.9 qhnRNU9b 1327 9.04 umc1771—umc1519 2.26 1.30 28.7 qhnRNU9c 1241 9.05 umc1078—phi236654 28.66 2.05 45.1 qhnRNU10 1308 10.04 umc1053—umc1506 36.30 2.23 49.3 注(Note):SNU—Shoot nitrogen upt;padding-top:3pt;line-height:10pt;height:12.7ptake; RNU—Root nitrogen upt;padding-top:3pt;line-height:10pt;height:12.7ptake; AE—Additive effect; QTL—数量性状位点 Quantitative trait locus; AEC—Additive effect contribution; AEC (%) = (SSSL 表型值 – 许 178 表型值)/(2 × 许 178 表型值) × 100, AEC (%) = (SSSLs – Xu178)/(2 × Xu178) × 100. 低氮条件下,共检测到 6 个地上部氮累积量 QTL 和 10 个根部氮累积量 QTL (P ≤ 0.001) (表 9)。6 个地上部氮累积量 QTL 均为正向加性效应,并且 3 个位于第 1 染色体上,单个 QTL 的贡献率为 33.2%~55.2%。检测到的 10 个根部氮累积量 QTL 中有 6 个表现负向加性效应,4 个为正向加性效应,单个 QTL 的贡献率为–27.3%~33.8%。
表 9 低氮条件下苗期玉米植株氮累积量 QTL 分析Table 9. QTL location of the nitrogen contents in the SSSL population under the low nitrogen condition性状
TraitQTL 代换系
SSSLBin 代换片段
Substituted segment片段长度 (cM)
Segment length加性效应
AE加性效应贡献率
AEC (%)地上部氮累积量 SNU qlnSNU1a 1394 1.02 bnlg1007—phi095 50.50 1.20 50.3 qlnSNU1b 1419 1.03 bnlg182—bnlg2295 55.50 1.31 55.2 qlnSNU1c 1479 1.06 bnlg1598—umc2396 35.80 1.08 45.5 qlnSNU8 1412 8.06 umc1724—phi015 57.58 0.78 32.7 qlnSNU9 1241 9.05 umc1078—phi236654 28.66 0.79 33.2 qlnSNU10 1324 10.03 umc1739—umc1053 25.80 0.97 40.7 根部氮累积量
RNUqlnRNU1a 1419 1.03 bnlg182—bnlg2295 55.50 0.40 29.0 qlnRNU1b 1297 1.05 umc1461—umc1124 9.10 –0.31 –22.6 qlnRNU1c 1413 1.06 umc2396—umc2047 59.95 0.37 27.0 qlnRNU4a 1374 4.05 umc1662—bnlg1444 141.14 0.31 22.7 qlnRNU4b 1292 4.09 umc1989—phi076 90.99 –0.31 –22.4 qlnRNU5 1416 5.00 umc1491—umc1478 90.00 –0.30 –21.9 qlnRNU6 1282 6.00 bnlg1043—umc1883 20.09 –0.30 –21.5 qlnRNU8 1494 8.06 umc2356—phi080 79.30 –0.20 –14.5 qlnRNU9 1259 9.04 umc1492—umc2207 20.30 –0.38 –27.3 qlnRNU10 1324 10.03 umc1739—umc1053 25.80 0.47 33.8 注(Note):SNU—Shoot nitrogen upt;padding-top:3pt;line-height:10pt;height:12.7ptake; RNU—Root nitrogen upt;padding-top:3pt;line-height:10pt;height:12.7ptake; AE—Additive effect; QTL—数量性状位点 Quantitative trait locus; AEC—Additive effect contribution; AEC (%) = (SSSL 表型值 – 许 178 表型值)/(2 × 许 178 表型值) × 100, AEC (%) = (SSSLs – Xu178)/(2 × Xu178) × 100. 2.6 QTL 在染色体上的分布
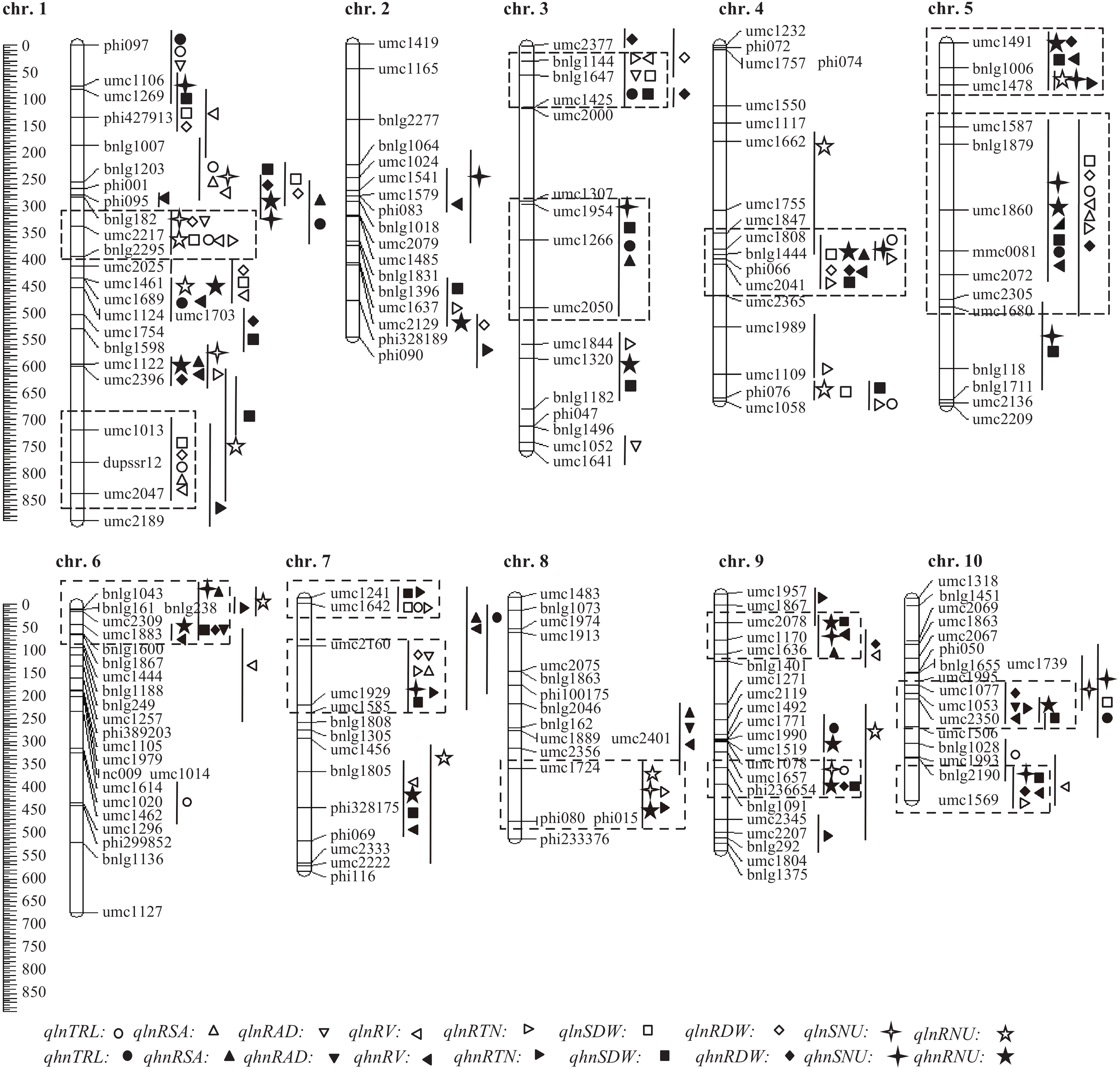
两个氮水平下检测到的 QTL 在染色体上的分布如图 2所示。结果显示,所检测到的根系性状和氮吸收相关 QTL 在玉米的 10 条染色体上均有分布,其中第 1 染色体上的 QTL 数目最多,第 2 和第 8 染色体上的 QTL 数目最少。对低氮和高氮下分别检测到的 QTL 进行分析可以得到不同氮条件下的特异性 QTL。有些染色体区间仅能在低氮水平下检测到 QTL,例如第 1 染色体 bnlg182—bnlg2295 (代换系 1419 的代换区间) 和 umc1013—umc2047 (代换系 1314 的代换区间) 区间。包含这些染色体片段的代换系很有可能只控制低氮条件下的根系形态,有利于我们发掘低氮特异的 QTL。同样,图 2结果也显示在有些染色体区段仅检测到高氮条件下 QTL,例如,第 1 染色体 phi001—bnlg182 (bin1.03,代换系 1428 的代换区间),umc1122—umc2396 (bin1.06,代换系 1376 的代换区间),第 6 染色体 bnlg1043—umc1883 (代换系 1282 的代换区间),第 9 染色体的 umc2078—umc1636 (代换系 1238 的代换区间),以及第 10 染色体的 umc1077—umc2350 (代换系 1473 的代换区间)。这些代换系在低氮条件下未检测到 QTL,我们将其定义为高氮特异的 QTL。这两类 QTL 为氮环境特异性 QTL,说明在不同氮条件下玉米有不同的代谢机制。其余的 QTL 区间均同时检测到高氮和低氮条件下的 QTL。此外,从 QTL 在染色体上的分布可以看出,玉米氮吸收相关的 QTL 区间均能检测到根系形态相关的 QTL,说明玉米根系形态与其氮吸收能力密切相关 (图 2)。
![]() 图 2 不同氮水平下玉米苗期根系形态及氮累积量 QTL 在染色体上的分布[注(Note):图中黑色线条表示代换系的代换片段所在的位置,线条右边的图形是检测到的各个性状的 QTL;虚线方框为 QTL 簇区间;A black line represents each maize chromosome with the marker intervals where the substituted segment was located. The detected QTLs were represented as different graphs in the right side of the black lines. Each dotted box represents the QTL cluster across chromosomes.]Figure 2. Position of the QTLs for maize root morphology and N uptake at the seedling stage under low and high N levels
图 2 不同氮水平下玉米苗期根系形态及氮累积量 QTL 在染色体上的分布[注(Note):图中黑色线条表示代换系的代换片段所在的位置,线条右边的图形是检测到的各个性状的 QTL;虚线方框为 QTL 簇区间;A black line represents each maize chromosome with the marker intervals where the substituted segment was located. The detected QTLs were represented as different graphs in the right side of the black lines. Each dotted box represents the QTL cluster across chromosomes.]Figure 2. Position of the QTLs for maize root morphology and N uptake at the seedling stage under low and high N levels3. 讨论
玉米已成为我国种植面积最大的农作物,但是目前其高产和超高产依赖大量的氮肥施用。如何提高玉米的氮肥利用率、减少肥料投入、保护环境已成为玉米高产高效的研究热点和亟待解决的问题。植物对土壤中氮素的获取很大程度上取决于其根系形态[22]。研究者通过对不同基因型玉米进行研究提出了玉米氮高效吸收的理想根系形态,即通过增加根体积,包括根干重、根长和根密度,提高玉米氮吸收能力和籽粒产量[8, 23]。研究表明玉米的根系形态受到土壤中有效氮含量影响,在缺氮条件下玉米节根数目降低,但是总根长增加[7]。玉米通过这种根系形态的改变从土壤中获取更多的氮素,用于植株的生长发育。本研究结果也显示,在低氮条件下,代换系玉米幼苗茎叶的生物量与总根长、根表面积、根体积、根尖数呈极显著相关性,而与根直径之间的相关性未达到显著水平。并且,低氮下植株氮吸收量与总根长、根表面积、根体积和根尖数均呈极显著正相关 (表 3)。这些结果说明,通过对玉米根性状相关 QTL 的研究有利于深入认识玉米氮吸收的遗传机制。
氮吸收效率和根系形态都是复杂的性状,受基因型和环境的共同影响。目前我们对玉米氮高效和根系形态的遗传基础仍知之甚少,对于两者之间的关系认识不足,这也限制了基于根系形态来筛选氮高效材料或品种的效率。近来 QTL 作图已经成为确定复杂性状的遗传位点甚至候选基因的有效工具。很多氮高效相关的农艺性状已经被定位,包括生物量[25]、籽粒氮吸收量[26]、吐丝后氮吸收效率以及氮再利用效率等[5]。同时,很多调控根系形态的 QTL 也在不同的玉米连锁群中被定位[10–11, 27–28]。例如,Burton 等[27]利用三个玉米重组自交系对 21 个根系性状进行 QTL 定位,共定位到 15 个 QTL,分布在 8 条染色体上 (第 6 和 10 染色体上没有检测到 QTL),表型变异为 0.44%~13.5%。Burton 等[27]对 Zhu 等[13]、Lebreton 等[12]研究中所定位到的 QTL 进行元分析,发现在 bin 1.03、1.04 和 7.03 位置分别有控制初生胚根上侧根长度、节根数目和次生胚根数目的 QTL。此外,Song 等[11]利用营养液培养方法,对含有 204 个材料的重组自交系根系性状进行 QTL 定位,在 bin1.06 检测到平均侧根长度 (SLL) 和根冠比的 QTL。本研究利用氮效率差异显著的两个玉米自交系构建的单片段代换系群体对玉米苗期的根系形态和氮吸收量进行了 QTL 定位分析。在高氮条件下,共检测到 40 个根系性状 QTL,34 个生物量 QTL,以及 28 个氮吸收量 QTL。对 HN 下的 QTL 进行分析发现,有些染色体区段仅特异检测到 HN 下的 QTL,并且不同性状的 QLT 成簇排列 (图 2),这些区段检测到 HN 下 QTL 不少于 5 个,表明这些区段可能包含有高氮条件下控制玉米根系形态的主效 QTL。本研究也在 bin1.03 (第 1 染色体 phi001—bnlg182 区间) 和 bin1.06 (umc1122—umc2396 区间) 检测到正常氮条件下根形态 QTL,与 Burton 等[27]和 Song 等[11]定位到的根形态相关 QTL 区间有重叠。这些结果表明在这两个 QTL 区间可能存在控制氮充足条件下玉米根系形态的主效 QTL。目前就我国农业生产现状而言,氮肥的施用量短期内很难显著降低[29],因此,培育在正常或高肥力条件下高效吸收的品种也是提高氮效率的途径之一[30]。在高产栽培条件下,随着产量水平的提高,作物需氮量增加,施氮总量也会提高。但是,我国目前玉米农田生产中多数农民采用一次性基肥,不仅增加了硝酸盐淋失的可能性,而且过高的硝酸盐浓度也限制了根系的生长发育,进而降低氮肥的吸收效率。因此,米国华等[31]提出玉米氮高效吸收的理想根构型具有在高产氮肥投入条件下仍能维持正常的侧根生长、总根长密度高,提高整体剖面氮素有效性的特点。本研究中所定位到的高氮特异的 QTL 簇对研究玉米高氮条件下氮高效吸收的遗传机制和基因定位具有一定意义。
本研究在低氮条件下共检测到 85 个根性状相关 QTL,在第 1 染色体 bnlg182—bnlg2295 (bin1.04) 和 umc1013—umc2047 (bin1.08) 区间不仅检测到低氮下根系形态相关 QTL,而且同时检测到低氮下生物量和氮吸收量的 QTL,说明低氮胁迫下植株氮吸收量与根系形态紧密联系。其中,第 1 染色体 bnlg182—bnlg2295 (bin1.04) 区间在 Burton 等[27]、Song 等[11]的研究中均检测到根系形态相关 QTL。Li 等[14]利用重组自交系,设置高氮和低氮水平,通过大田试验和水培实验对氮利用效率 (NUE) 和根系构型 (RSA) 相关性状的 QTL 进行定位,共检测到 331 个 QTL。该研究发现氮吸收效率与根系构型之间存在显著的表型相关性。研究表明大约 70% 的 NUE 相关 QTL 簇与根系构型 QTL 簇重叠,这表明根系构型与氮利用率相关性状之间有显著的遗传学关系,可以通过分子标记筛选优异的根系构型来提高玉米的氮利用效率[14]。本研究结果也显示,除了极个别的代换区间仅单独检测到氮累积量的 QTL,其他绝大部分植株氮吸收量 QTL 都与根形态相关 QTL 成簇存在 (图 2)。此外,Li 等[14]的研究发现有 5 个重要的 QTL 簇对玉米获得大的根系和更高的氮吸收效率有利,其中一个 QTL 簇位于染色体区间 bin1.04。这一结果与我们发现的 QTL 簇区间 bnlg182—bnlg2295 (bin1.04) 有重叠,并且在这个 QTL 区间同时检测到低氮下植株氮吸收量的 QTL,表明该区间很有可能包含有控制玉米氮高效的主效 QTL 位点,进一步对包含该区段的代换系 1419 进行精细定位可能获得氮高效候选基因。Gallais 等[9]通过大田和水培相结合的方法对玉米花后氮素吸收、氮再利用、籽粒产量、叶片硝酸还原酶、谷氨酰胺合成酶活性等性状进行了 QTL 定位,发现三个染色体位置上检测到产量 QTL、氮再利用 QTL、GS 活性 QTL 和一个编码胞质 GS 的基因具有一致性,并且位于第 5 染色体的 GS 位点能够部分解释玉米氮利用效率的表型变异。同时,该研究定位的 QTL 簇有些与本研究重要 QTL 簇位置接近,例如,Gallais 等[9]在第 1 染色体的顶端区间检测到茎秆氮再利用 QTL、GDH 和 GS 酶活性相关 QTL,而我们的结果显示在同一区段检测到总根长、植株生物量等 QTL;在位于第 5 染色体 umc1587—umc1680 (代换系 1328 的代换区间) 的 QTL 簇接近的位置,Gallais 等[9]定位到叶片硝酸盐含量、GS 和 NR 活性 QTL;在 bin1.08 位置 (umc1013—umc2047 区间,代换系 1314) Gallais 等检测到控制整株氮再利用的 QTL。此外,齐欢欢等[32]研究发现第 1 染色体的 umc2217—umc1770 区段与穗长、穗位高和穗行数有关,这一区间与我们的 QTL 簇 bnlg182—bnlg2295 (代换系 1419) 部分重叠。这说明本研究所定位到的低氮特异 QTL 簇可能在玉米低氮条件下氮素高效吸收中起重要作用,与氮高效吸收密切相关。此外,在一些 QTL 区间同时检测到多个高氮和低氮 QTL,且成簇排列,这些 QTL 不受氮水平特异诱导。例如第 3 染色体 bnlg144—umc1425 区间,第 7 染色体 umc1241—umc1642 区间,第 8 染色体 umc1724—phi080 区间 (图 2)。这些不同环境下检测到的 QTL 簇表明玉米在不同的氮环境下具有不同的代谢机制。高、低氮特异的 QTL 簇对我们研究玉米适应氮胁迫具有重要意义,有利于挖掘不同氮水平下的氮高效基因,更有针对性地开展玉米氮高效育种。
4. 结论
不同氮水平下,玉米的根系形态存在显著差异,对高、低氮条件下检测到的特异 QTL 进行分析有助于解析不同氮水平下玉米高效吸收氮素的遗传机制。本研究在染色体区段 bnlg182—bnlg2295 (bin1.04,代换系 1419)、umc1013—umc2047 (bin1.08,代换系 1314) 区间检测到多个低氮特异的 QTL,在 phi001—bnlg182 (bin1.03,代换系 1428) 和 umc1122—umc2396 (bin1.06,代换系 1376) 等区间检测到高氮条件下特异的 QTL。这些 QTL 区间同时包含根系形态 QTL 和氮吸收量 QTL,并且在前人的研究中均有玉米氮效率或根系形态相关 QTL 检测到,说明这些区间在玉米氮高效吸收中可能起重要作用,后期会对包含该区段的代换系进行精细定位,获得候选基因。
-
图 2 不同氮水平下玉米苗期根系形态及氮累积量 QTL 在染色体上的分布
[注(Note):图中黑色线条表示代换系的代换片段所在的位置,线条右边的图形是检测到的各个性状的 QTL;虚线方框为 QTL 簇区间;A black line represents each maize chromosome with the marker intervals where the substituted segment was located. The detected QTLs were represented as different graphs in the right side of the black lines. Each dotted box represents the QTL cluster across chromosomes.]
Figure 2. Position of the QTLs for maize root morphology and N uptake at the seedling stage under low and high N levels
表 1 不同氮水平下亲本各表型值和双因素显著性分析
Table 1 Mean values and significance of two-factor ANOVA analysis for the nine traits in the parents
性状
Trait高氮 High-N 低氮 Low-N 显著性 Significance 许 178
Xu178综 3
Zong3许 178
Xu178综 3
Zong3氮水平 (N)
N level基因型 (G)
Genotype交互作用
N × G总根长 TRL 828 1082 784 378 ** ns ** 根表面积 RSA 126 145 121 64.9 ** ns ** 根直径 RAD 0.51 0.43 0.50 0.40 ns *** ns 根体积 RV 1.23 1.50 1.37 0.64 * ns ** 根尖数 RTN 2190 1245 717 641 *** ** ** 地上部干重 SDW 0.81 0.77 0.23 0.15 *** *** *** 根干重 RDW 0.26 0.23 0.16 0.09 *** *** *** 地上部氮累积量 SNU 28.58 23.88 2.38 1.13 *** ** * 根氮累积量 RNU 4.54 4.85 1.38 0.52 *** * *** 注(Note):TRL—总根长 Total root length; RSA—根表面积 Root surface area; RAD—根直径 Root average diameter; RV—根体积 Root volume; RTN—根尖数 Root tip number; SDW—地上部干重 Shoot dry weight; RDW—根干重 Root dry weight; SNU—地上部氮累积量 Shoot nitrogen uptake; RNU—根部氮累积量 Root nitrogen uptake; G—基因型 Genotype; N × G—氮水平 × 基因型 N level × genotype; ns—无显著性差异 Not significant; ***—P ≤ 0.001; **—0.001 < P ≤ 0.01; *—0.01 < P ≤ 0.05. 表 2 不同氮水平下 SSSL 群体各表型值和双因素显著性分析
Table 2 Mean values and significance of two-factor ANOVA analysis for the nine traits in the SSSL population
性状
Trait高氮 High-N 低氮 Low-N 显著性 Significance 均值
Mean范围
Range变异 (%)
CV均值
Mean范围
Range变异 (%)
CV氮水平 (N)
N level基因型 (G)
Genotype交互作用
N × G总根长 TRL 1224 604~2558 22.1 986 297~1439 21.5 *** *** *** 根表面积 RSA 219 103~338 21.8 150 47.83~266 23.9 *** *** *** 根直径 RAD 0.57 0.45~0.69 8.4 0.49 0.41~0.56 5.6 *** *** *** 根体积 RV 3.09 0.91~4.57 26.6 1.87 0.61~3.19 25.7 *** *** *** 根尖数 RTN 1758 738~3748 33.9 919 466~1725 26.9 *** *** *** 地上部干重 SDW 1.26 0.55~1.88 21.4 0.26 0.09~0.42 24.1 *** *** *** 根干重 RDW 0.32 0.11~0.52 27.2 0.2 0.07~0.32 21.3 ns ** ns 地上部氮累积量 SNU 38.25 13.57~64.11 24.8 2.83 1.08~4.78 21.8 *** *** *** 根氮累积量 RNU 6.62 2.38~10.23 27.0 1.34 0.63~2.31 26.9 *** * * 注(Note):TRL—总根长 Total root length; RSA—根表面积 Root surface area; RAD—根直径 Root average diameter; RV—根体积 Root volume; RTN—根尖数 Root tip number; SDW—地上部干重 Shoot dry weight; RDW—根干重 Root dry weight; SNU—地上部氮累积量 Shoot nitrogen uptake; RNU—根部氮累积量 Root nitrogen uptake; ns—无显著性差异 not significant; ***—P ≤ 0.001; **—0.001 < P ≤ 0.01; *—0.01 < P ≤ 0.05. 表 3 群体各表型性状在高氮水平 (对角线上方) 和低氮水平 (对角线下方) 的 Pearson 相关系数
Table 3 Pearson’s correlation coefficients between the traits under high N (above diagonal) and low N conditions (below diagonal) in the SSSL population
TRL RSA RAD RV RTN SDW RDW SNU RNU TRL 0.913*** –0.049 0.780*** 0.454*** 0.767*** 0.738*** 0.616*** 0.585*** RSA 0.872*** 0.228** 0.934*** 0.392*** 0.840*** 0.838*** 0.661*** 0.674*** RAD –0.129 0.118 0.434*** –0.148 0.299** 0.393*** 0.174* 0.330*** RV 0.795*** 0.927*** 0.301*** 0.271** 0.827*** 0.854*** 0.633*** 0.704*** RTN 0.476*** 0.417*** –0.179* 0.352*** 0.279** 0.289** 0.203* 0.246** SDW 0.698*** 0.752*** 0.093 0.726*** 0.458*** 0.880*** 0.682*** 0.725*** RDW 0.792*** 0.840*** 0.169* 0.835*** 0.425*** 0.831*** 0.676*** 0.760*** SNU 0.521*** 0.542*** 0.054 0.536*** 0.387*** 0.707*** 0.599*** 0.701*** RNU 0.388** 0.456*** 0.033 0.446*** 0.328*** 0.430*** 0.422*** 0.394*** 注(Note):TRL—总根长 Total root length; RSA—根表面积 Root surface area; RAD—根直径 Root average diameter; RV—根体积 Root volume; RTN—根尖数 Root tip number; SDW—地上部干重 Shoot dry weight; RDW—根干重 Root dry weight; SNU—地上部氮累积量 shoot nitrogen uptake; RNU—根氮累积量 Root nitrogen uptake. ***—P ≤ 0.001; **—0.001 < P ≤ 0.01; *—0.01 < P ≤ 0.05. 表 4 高氮下玉米 SSSL 群体的根系形态 QTL 及其效应值
Table 4 QTL location of the maize root morphology in the SSSL population under the high nitrogen condition
性状
TraitQTL 代换系
SSSLBin 代换片段
Substituted segment片段长度 (cM)
Segment length加性效应
AE加性效应贡献率
AEC (%)总根长
TRLqhnTRL1a 1339 1.01 end—umc1269 81.53 392.54 47.4 qhnTRL1b 1468 1.03 phi001—bnlg182 7.60 –220.68 –26.6 qhnTRL1c 1306 1.05 umc1461—umc1124 9.10 495.78 59.8 qhnTRL3a 1481 3.02 bnlg1144—umc1425 44.00 351.43 42.4 qhnTRL5 1293 5.02 umc1587—bnlg118 139.10 360.79 43.6 qhnTRL7 1343 7.00 umc1642—umc1929 110.95 455.78 55.0 qhnTRL9 1327 9.04 umc1771—umc1519 2.26 297.66 35.9 qhnTRL10 1255-1 10.03 phi050—umc1506 48.50 326.49 39.4 根表面积
RSAqhnRSA1a 1468 1.03 phi001—bnlg182 7.60 –29.73 –23.5 qhnRSA1b 1376 1.06 umc1122—umc1013 61.26 105.87 83.7 qhnRSA4 1350 4.08 bnlg1444—umc1109 176.54 65.78 52.0 qhnRSA6 1282 6.00 bnlg1043—umc1883 20.09 73.70 58.3 qhnRSA7 1365 7.00 umc1241—umc1585 236.47 66.07 52.3 qhnRSA8 1485 8.04 bnlg2046—umc1724 93.52 67.33 53.3 qhnRSA9 1266 9.01 umc1867—umc1170 38.40 75.27 59.5 根直径
RADqhnRAD1 1461 1.05 umc1703—bnlg1598 37.90 0.09 17.4 qhnRAD6 1469 6.00 bnlg238—umc1883 16.29 0.08 16.2 qhnRAD8 1485 8.04 bnlg2046—umc1724 93.52 0.07 14.1 qhnRAD10 1473 10.04 umc1077—umc2350 15.10 0.08 16.2 根体积
RVqhnRV1b 1306 1.05 umc1461—umc1124 9.10 1.38 112.7 qhnRV2 1325 2.04 umc1541—phi083 4.90 0.97 79.4 qhnRV4 1349 4.08 bnlg1444—umc2041 9.35 1.24 100.8 qhnRV5 1321 5.02 umc1587—umc2072 138.95 1.21 98.9 qhnRV7a 1365 7.00 umc1241—umc1585 236.47 1.33 108.2 qhnRV7b 1267 7.05 bnlg1805—phi069 76.55 1.53 125.1 qhnRV8 1485 8.04 bnlg2046—umc1724 93.52 1.51 123.3 qhnRV9 1266 9.01 umc1867—umc1170 38.40 1.51 123.6 表 5 低氮下玉米 SSSL 群体的根系形态 QTL 及其效应值
Table 5 QTL location of the root morphology in the SSSL population under the low nitrogen condition
性状
TraitQTL 代换系
SSSLBin 代换片段
Substituted segment片段长度 (cM)
Segment length加性效应
AE加性效应贡献率
AEC (%)总根长
TRLqlnTRL1a 1339 1.01 end—umc1269 81.53 281 35.8 qlnTRL1b 1394 1.02 bnlg1007—phi095 50.50 328 41.8 qlnTRL1c 1314 1.08 umc1013—umc2047 59.95 –250 –31.9 qlnTRL4a 1374 4.05 umc1662—bnlg1444 141.14 242 30.8 qlnTRL4b 1296 4.11 phi076—end 25.10 160 20.5 qlnTRL5 1328 5.02 umc1587—umc1680 213.20 493 62.9 qlnTRL6 1448 6.04 umc1614—umc1462 43.35 226 28.9 qlnTRL7 1277 7.00 umc1241—umc1642 13.60 305 39.0 qlnTRL9 1241 9.05 umc1078—phi236654 28.66 150 19.2 qlnTRL10 1240 10.06 bnlg1028—bnlg2190 15.90 247 31.5 根表面积
RSAqlnRSA1a 1394 1.02 bnlg1007—phi095 50.50 51.19 42.2 qlnRSA1b 1419 1.03 bnlg182—bnlg2295 55.50 44.53 36.7 qlnRSA1c 1314 1.08 umc1013—umc2047 59.95 –34.52 –28.5 qlnRSA5 1328 5.02 umc1587—umc1680 213.20 72.45 59.7 qlnRSA7 1382 7.01 umc2160—umc1585 73.17 42.70 35.2 根直径
RADqlnRAD1a 1339 1.01 end—umc1269 81.53 –0.04 –7.3 qlnRAD1b 1415 1.03 bnlg182—bnlg2295 55.50 –0.02 –5.0 qlnRAD3b 1423 3.02 bnlg1144—umc1425 44.00 –0.04 –7.3 qlnRAD3a 1341 3.09 umc1052—end 8.56 –0.04 –8.9 qlnRAD7a 1382 7.01 umc2160—umc1585 73.17 –0.03 –5.4 根体积
RVqlnRV1a 1437 1.01 umc1269—bnlg1007 52.12 –0.37 –26.7 qlnRV1b 1394 1.02 bnlg1007—phi095 50.50 0.70 51.1 qlnRV1c 1419 1.03 bnlg182—bnlg2295 55.50 0.65 47.7 qlnRV1d 1403 1.05 umc2025—umc1689 11.06 0.50 36.4 qlnRV1e 1314 1.08 umc1013—umc2047 59.95 –0.35 –25.6 qlnRV3 1423 3.02 bnlg1144—umc1425 44.00 –0.20 –14.3 qlnRV4 1313 4.08 bnlg1444—umc2041 9.35 0.57 41.7 qlnRV5 1328 5.02 umc1587—umc1680 213.20 0.91 66.3 qlnRV6 1276 6.00 umc1883—umc1257 47.48 0.53 38.4 qlnRV7 1239 7.05 bnlg1805—phi069 76.55 0.60 44.1 qlnRV9 1462 9.02 umc1170—bnlg1401 77.90 0.61 44.3 qlnRV10 1388 10.06 umc1993—umc1569 47.45 0.77 55.9 根尖数
RTNqlnRTN1a 1419 1.03 bnlg182—bnlg2295 55.50 395 55.0 qlnRTN1b 1479 1.06 bnlg1598—umc2396 35.80 384 53.6 qlnRTN2 1366 2.06 bnlg1396—umc2129 18.86 275 38.3 qlnRTN3a 1457 3.02 bnlg1144—umc1425 44.00 205 28.6 qlnRTN3b 1486 3.08 umc1844—bnlg1182 60.24 203 28.3 qlnRTN4a 1313 4.08 bnlg1444—umc2041 9.35 426 59.4 qlnRTN4b 1292 4.09 umc1989—phi076 90.99 –125 –17.5 qlnRTN4c 1296 4.11 phi076—end 25.10 240 33.5 qlnRTN5 1328 5.02 umc1587—umc1680 213.20 222 31.0 qlnRTN6 1469 6.00 bnlg238—umc1883 16.29 217 30.3 qlnRTN7a 1277 7.00 umc1241—umc1642 13.60 349 48.7 qlnRTN7b 1382 7.01 umc2160—umc1585 73.17 504 70.3 qlnRTN8 1480 8.06 umc1724—phi015 57.58 433 60.4 qlnRTN10a 1324 10.03 umc1739—umc1053 25.80 230 32.0 qlnRTN10b 1395 10.06 bnlg2190—end 46.60 234 32.7 注(Note):TRL—total root length; RSA—Root surface area; RAD—Root average diameter; RV—Root volume; RTN—Root tip number; QTL—数量性状位点 Quantitative trait locus; AE—Additive effect; AEC—Additive effect contribution; AEC (%) = (SSSL 表型值 – 许 178 表型值)/(2 × 许 178 表型值) × 100, AEC (%) = (SSSLs – Xu178)/(2 × Xu178) × 100. 表 6 高氮条件下玉米苗期生物量 QTL 及其效应
Table 6 QTL location of plant biomass in the SSSL population under the high nitrogen condition
性状
TraitQTL 代换系
SSSLBin 代换片段
Substituted segment片段长度 (cM)
Segment length加性效应
AE加性效应贡献率
AEC (%)地上部干重
SDWqhnSDW1a 1279 1.01 umc1106—phi427913 29.58 0.28 35.0 qhnSDW1b 1428 1.03 phi001—bnlg182 7.60 –0.32 –40.1 qhnSDW1c 1306 1.05 umc1461—umc1124 9.10 0.35 43.8 qhnSDW1d 1459 1.06 umc2396—umc2047 148.92 0.51 63.8 qhnSDW2 1366 2.06 bnlg1396—umc2129 18.86 0.30 37.4 qhnSDW3a 1481 3.02 bnlg1144—umc1425 44.00 0.38 47.2 qhnSDW3c 1335 3.08 umc1844—bnlg1182 60.24 0.51 63.8 qhnSDW4a 1349 4.08 bnlg1444—umc2041 9.35 0.47 57.9 qhnSDW4b 1296 4.11 phi076—end 25.10 0.31 38.8 qhnSDW5a 1421 5.00 end—bnlg1006 50.10 0.53 65.7 qhnSDW5b 1293 5.02 umc1587—bnlg118 139.10 0.26 32.6 qhnSDW5c 1286 5.06 umc1680—bnlg118 39.10 0.26 32.7 qhnSDW6 1282 6.00 bnlg1043—umc1883 20.09 0.49 60.2 qhnSDW7a 1277 7.00 umc1241—umc1642 13.60 0.28 34.6 qhnSDW7b 1382 7.01 umc2160—umc1585 73.17 0.23 28.3 qhnSDW7c 1239 7.05 bnlg1805—phi069 76.55 0.26 31.9 qhnSDW9a 1266 9.01 umc1867—umc1170 38.40 0.28 35.0 qhnSDW9b 1241 9.05 umc1078—phi236654 28.66 0.38 47.5 qhnSDW10a 1436 10.04 umc2350—bnlg1028 48.50 0.54 66.8 qhnSDW10b 1388 10.06 umc1993—umc1569 47.45 0.71 88.3 根干重
RDWqhnRDW1a 1428 1.03 phi001—bnlg182 7.60 –0.04 –29.4 qhnRDW1b 1297 1.05 umc1461—umc1124 9.10 0.10 38.2 qhnRDW1c 1376 1.06 umc1122—umc1013 61.26 0.11 41.4 qhnRDW3a 1455 3.01 umc2377—bnlg1647 27.43 –0.01 –10.2 qhnRDW3b 1481 3.02 bnlg1144—umc1425 44.00 0.09 32.7 qhnRDW4 1349 4.08 bnlg1444—umc2041 9.35 0.11 42.8 qhnRDW5a 1421 5.00 end—bnlg1006 50.10 0.09 33.2 qhnRDW5b 1328 5.06 umc1587—umc1680 213.20 –0.07 –24.8 qhnRDW6 1282 6.00 bnlg1043—umc1883 20.09 0.09 33.4 qhnRDW7 1267 7.05 bnlg1805—phi069 76.55 0.08 29.0 qhnRDW9a 1462 9.02 umc1170—bnlg1401 77.90 –0.05 –18.7 qhnRDW9b 1241 9.05 umc1078—phi236654 28.66 0.09 33.2 qhnRDW10a 1473 10.04 umc1077—umc2350 15.10 0.07 26.9 qhnRDW10b 1395 10.06 bnlg2190—end 46.60 0.11 40.3 注(Note):SDW—Shoot dry weight; RDW—Root dry weight; QTL—数量性状位点 Quantitative trait locus; AE—Additive effect; AEC—Additive effect contribution; AEC (%) = (SSSL 表型值 – 许 178 表型值)/(2 × 许 178 表型值) × 100, AEC (%) = (SSSLs – Xu178)/(2 × Xu178) × 100. 表 7 低氮条件下玉米苗期生物量 QTL 及其效应
Table 7 QTL location of plant biomass in the SSSL population under the low nitrogen condition
性状
TraitQTL 代换系
SSSLBin 代换片段
Substituted segment片段长度 (cM)
Segment length加性效应
AE加性效应贡献率
AEC (%)地上部干重 SDW qlnSDW1a 1437 1.01 umc1269—bnlg1007 52.12 –0.06 –24.5 qlnSDW1b 1394 1.02 bnlg1007—phi095 50.50 0.09 37.3 qlnSDW1c 1419 1.03 bnlg182—bnlg2295 55.50 0.08 35.2 qlnSDW1d 1383 1.05 umc1689—umc1703 8.94 0.08 35.9 qlnSDW1e 1314 1.08 umc1013—umc2047 59.95 –0.05 –20.1 qlnSDW3 1420 3.02 bnlg1144—umc1425 44.00 –0.02 –10.3 qlnSDW4a 1313 4.08 bnlg1444—umc2041 9.35 0.06 26.2 qlnSDW4b 1292 4.09 umc1989—phi076 90.99 –0.02 –9.3 qlnSDW5 1328 5.02 umc1587—umc1680 213.20 0.14 59.9 qlnSDW7 1277 7.00 umc1241—umc1642 13.60 0.08 34.4 qlnSDW10 1255-1 10.03 phi050—umc1506 48.50 0.09 39.0 根干重
RDWqlnRDW1a 1437 1.01 umc1269—bnlg1007 52.12 –0.03 –19.2 qlnRDW1b 1394 1.02 bnlg1007—phi095 50.50 0.07 41.8 qlnRDW1c 1419 1.03 bnlg182—bnlg2295 55.50 0.06 39.7 qlnRDW1d 1403 1.05 umc2025—umc1689 11.06 0.05 32.4 qlnRDW1e 1314 1.08 umc1013—umc2047 59.95 –0.03 –18.0 qlnRDW2 1361 2.07 umc2129—phi090 67.10 0.04 27.7 qlnRDW3a 1455 3.01 umc2377—bnlg1647 27.43 –0.02 –13.5 qlnRDW3b 1335 3.08 umc1844—bnlg1182 60.24 0.04 25.8 qlnRDW4 1301 4.08 bnlg1444—umc2041 9.35 0.04 23.3 qlnRDW5 1328 5.02 umc1587—umc1680 213.20 0.08 48.4 qlnRDW7 1382 7.01 umc2160—umc1585 73.17 0.04 27.7 注(Note):SDW—Shoot dry weight; RDW—Root dry weight; QTL—数量性状位点 Quantitative trait locus; AE—Additive effect; AEC—Additive effect contribution; AEC (%) = (SSSL 表型值 – 许 178 表型值)/(2 × 许 178 表型值) × 100, AEC(%) = (SSSLs – Xu178)/(2 × Xu178) × 100. 表 8 高氮条件下苗期玉米植株氮累积量 QTL 分析
Table 8 QTL location of the nitrogen accumulation in the SSSL population under the high nitrogen condition
性状
TraitQTL 代换系
SSSLBin 代换片段
Substituted segment片段长度 (cM)
Segment length加性效应
AE加性效应贡献率
AEC (%)地上部氮累积量
SNUqhnSNU1a 1279 1.01 umc1106—phi427913 29.58 9.00 31.5 qhnSNU1b 1428 1.03 phi001—bnlg182 7.60 –12.25 –42.7 qhnSNU2a 1335 2.04 umc1541—umc2079 60.24 12.88 45.1 qhnSNU2b 1366 2.06 bnlg1396—umc2129 18.86 8.43 29.5 qhnSNU4 1349 4.08 bnlg1444—umc2041 9.35 10.91 38.2 qhnSNU5a 1421 5.00 end—bnlg1006 50.10 17.41 60.9 qhnSNU5b 1321 5.02 umc1587—umc2072 138.95 8.21 28.7 qhnSNU5c 1286 5.06 umc1680—bnlg1711 39.10 4.61 16.1 qhnSNU6 1282 6.00 bnlg1043—umc1883 20.09 12.55 43.9 qhnSNU7 1382 7.01 umc2160—umc1585 73.17 7.66 26.8 qhnSNU9 1238 9.02 umc2078—umc1636 34.20 7.09 24.8 qhnSNU10a 1255-1 10.03 phi050—umc1506 48.50 11.35 39.7 qhnSNU10b 1395 10.06 bnlg2190—end 46.60 11.49 40.2 根部氮累积量
RNUqhnRNU1a 1428 1.03 phi001—bnlg182 7.60 –1.61 –35.5 qhnRNU1b 1297 1.05 umc1461—umc1124 9.10 2.85 62.8 qhnRNU1c 1376 1.06 umc1122—umc1013 61.26 3.25 71.7 qhnRNU2 1366 2.06 bnlg1396—umc2129 18.86 1.85 40.7 qhnRNU3 1486 3.08 umc1844—bnlg1182 60.24 1.76 38.9 qhnRNU4 1349 4.08 bnlg1444—umc2041 9.35 1.91 42.2 qhnRNU5a 1283 5.00 umc1491—umc1478 90.00 1.36 30.0 qhnRNU5b 1321 5.02 umc1587—umc2072 138.95 1.47 32.5 qhnRNU6 1282 6.00 bnlg1043—umc1883 20.09 2.64 58.1 qhnRNU7 1239 7.04 bnlg1805—phi069 76.55 2.39 52.6 qhnRNU8 1427 8.06 umc1724—phi015 57.58 2.44 53.9 qhnRNU9a 1238 9.02 umc2078—umc1636 34.20 1.81 39.9 qhnRNU9b 1327 9.04 umc1771—umc1519 2.26 1.30 28.7 qhnRNU9c 1241 9.05 umc1078—phi236654 28.66 2.05 45.1 qhnRNU10 1308 10.04 umc1053—umc1506 36.30 2.23 49.3 注(Note):SNU—Shoot nitrogen upt;padding-top:3pt;line-height:10pt;height:12.7ptake; RNU—Root nitrogen upt;padding-top:3pt;line-height:10pt;height:12.7ptake; AE—Additive effect; QTL—数量性状位点 Quantitative trait locus; AEC—Additive effect contribution; AEC (%) = (SSSL 表型值 – 许 178 表型值)/(2 × 许 178 表型值) × 100, AEC (%) = (SSSLs – Xu178)/(2 × Xu178) × 100. 表 9 低氮条件下苗期玉米植株氮累积量 QTL 分析
Table 9 QTL location of the nitrogen contents in the SSSL population under the low nitrogen condition
性状
TraitQTL 代换系
SSSLBin 代换片段
Substituted segment片段长度 (cM)
Segment length加性效应
AE加性效应贡献率
AEC (%)地上部氮累积量 SNU qlnSNU1a 1394 1.02 bnlg1007—phi095 50.50 1.20 50.3 qlnSNU1b 1419 1.03 bnlg182—bnlg2295 55.50 1.31 55.2 qlnSNU1c 1479 1.06 bnlg1598—umc2396 35.80 1.08 45.5 qlnSNU8 1412 8.06 umc1724—phi015 57.58 0.78 32.7 qlnSNU9 1241 9.05 umc1078—phi236654 28.66 0.79 33.2 qlnSNU10 1324 10.03 umc1739—umc1053 25.80 0.97 40.7 根部氮累积量
RNUqlnRNU1a 1419 1.03 bnlg182—bnlg2295 55.50 0.40 29.0 qlnRNU1b 1297 1.05 umc1461—umc1124 9.10 –0.31 –22.6 qlnRNU1c 1413 1.06 umc2396—umc2047 59.95 0.37 27.0 qlnRNU4a 1374 4.05 umc1662—bnlg1444 141.14 0.31 22.7 qlnRNU4b 1292 4.09 umc1989—phi076 90.99 –0.31 –22.4 qlnRNU5 1416 5.00 umc1491—umc1478 90.00 –0.30 –21.9 qlnRNU6 1282 6.00 bnlg1043—umc1883 20.09 –0.30 –21.5 qlnRNU8 1494 8.06 umc2356—phi080 79.30 –0.20 –14.5 qlnRNU9 1259 9.04 umc1492—umc2207 20.30 –0.38 –27.3 qlnRNU10 1324 10.03 umc1739—umc1053 25.80 0.47 33.8 注(Note):SNU—Shoot nitrogen upt;padding-top:3pt;line-height:10pt;height:12.7ptake; RNU—Root nitrogen upt;padding-top:3pt;line-height:10pt;height:12.7ptake; AE—Additive effect; QTL—数量性状位点 Quantitative trait locus; AEC—Additive effect contribution; AEC (%) = (SSSL 表型值 – 许 178 表型值)/(2 × 许 178 表型值) × 100, AEC (%) = (SSSLs – Xu178)/(2 × Xu178) × 100. -
[1] Mi G, Chen F, Zhang F. Physiological and genetic mechanisms for nitrogen use efficiency in maize[J]. Journal of Crop Science Biotechnology, 2007, 10(2): 57–63.
[2] Xu G, Fan X, Miller A J. Plant nitrogen assimilation and use efficiency[J]. Annual Review of Plant Biology, 2012, 63: 153–182.
[3] DeBruin J, Messina C D, Munaro E, et al. N distribution in maize plant as a marker for grain yield and limits on its remobilization after flowering[J]. Plant Breeding, 2013, 132(5): 500–505.
[4] Mackay A D, Barber S A. Effect of nitrogen on root growth of two corn genotypes in the field[J]. Agronomy Journal, 1986, 78(4): 699–708.
[5] Coque M, Martin A, Veyrieras J B, et al. Genetic variation for N-remobilization and post-silking N-uptake in a set of maize recombinant inbred lines. 3. QTL detection and coincidences[J]. Theoretical and Applied Genetics, 2008, 117(5): 729–747.
[6] Mu X, Chen F, Wu Q, et al. Genetic improvement of root growth increases maize yield via enhanced post-silking nitrogen uptake[J]. European Journal of Agronomy, 2015, 63: 55–61.
[7] Saengwilai P, Tian X, Lynch J P. Low crown root number enhances nitrogen acquisition from low-nitrogen soils in maize[J]. Plant Physiology, 2014, 166(2): 581–589.
[8] Lynch J P. Steep, cheap and deep: an ideotype to optimize water and N acquisition by maize root systems[J]. Annals of Botany, 2013, 112(2): 347–357.
[9] Gallais A, Hirel B. An approach to the genetics of nitrogen use efficiency in maize[J]. Journal of Experimental Botany, 2004, 55(396): 295–306.
[10] Hochholdinger F, Tuberosa R. Genetic and genomic dissection of maize root development and architecture[J]. Current Opinion of Plant Biology, 2009, 12(2): 172–177.
[11] Song W, Wang B, Hauck A L, et al. Genetic dissection of maize seedling root system architecture traits using an ultra-high density bin-map and a recombinant inbred line population[J]. Journal of Integrate Plant Biology, 2016, 58(3): 266–279.
[12] Lebreton C, Lazić-Jančić V, Steed A, et al. Identification of QTL for drought responses in maize and their use in testing causal relationships between traits[J]. Journal of Experimental Botany, 1995, 46: 853–865.
[13] Zhu J, Kaeppler S M, Lynch J P. Mapping of QTLs for lateral root branching and length in maize (Zea mays L.) under differential phosphorus supply[J]. Theoretical and Applied Genetics, 2005, 111(4): 688–695.
[14] Li P, Chen F, Cai H, et al. A genetic relationship between nitrogen use efficiency and seedling root traits in maize as revealed by QTL analysis[J]. Journal of Experimental Botany, 2015, 66(11): 3175–3188.
[15] 毛克举, 李卫华, 付志远, 等. 玉米自交系许178背景的综3染色体单片段代换系的构建[J]. 河南农业大学学报, 2013, 47(1): 6–9, 15. Mao K J, Li W H, Fu Zh Y, et al. Development of a set of signal segment substitution lines of an elite inbred line Zong 3 on the genetic background Xu 178 in maize (Zea mays L.)[J]. Journal of Henan Agriculture University, 2013, 47(1): 6–9, 15. [16] 刘宗华, 王春丽, 汤继华, 等. 氮胁迫对不同玉米自交系若干农艺性状和产量的影响[J]. 河南农业大学学报, 2006, 40(6): 573–577. Liu Z H, Wang C L, Tang J H, et al. Effect of low nitrogen stress on several agronomy characters and grain yield of different maize inbreds[J]. Journal of Henan Agriculture University, 2006, 40(6): 573–577. [17] 田秋英. 根系形态在玉米高效获取氮素中的作用及其生理调节机制[D]. 北京: 中国农业大学博士学位论文, 2005. Tian Q Y. Role of root morphology in efficient nitrogen acquisition in maize and its physiological regulation mechanism[D]. Beijing: PhD Dissertation of China Agricultural University, 2005.
[18] 刘冠明, 李文涛, 曾瑞珍, 等. 水稻单片段代换系代换片段的QTL鉴定[J]. 遗传学报, 2004, 12: 1395–1400. Liu G M, Li W T, Zeng R Z, et al. Identification of QTLs on substituted segments in signal segment substitution lines of rice[J]. Acta Genetica Sinica, 2004, 12: 1395–1400. [19] Eshed Y, Zamir D. An introgression line population of Lycopersicon Pennellii in the cultivated tomato enables the identification and fine mapping of yield-associated QTL[J]. Genetics, 1995, 141(3): 1147–1162.
[20] McCouch S R, Cho Y G, Yano M. Report on QTL nomenclature[J]. Rice Genetics Newsletter, 1997, 14: 11–13.
[21] Paterson A H, DeVerna J W, Lanini B, et al. Fine mapping of quantitative trait loci using selected overlapping recombinant chromosomes, in an interspecies cross of tomato[J]. Genetics, 1990, 124(3): 735–742.
[22] Forde B G. Nitrogen signaling pathways shaping root system architecture: an update[J]. Current Opinion of Plant Biology, 2014, 21: 30–36.
[23] Mi G, Chen F, Wu Q, et al. Ideotype root architecture for efficient nitrogen acquisition by maize in intensive cropping systems[J]. Science China (Life Sciences), 2010, 53(12): 1369–1373.
[24] 李卓坤, 彭涛, 张卫东, 等. 利用" 永久F_2”群体进行小麦幼苗根系性状QTL分析[J]. 作物学报, 2010, 36(3): 442–448. Li Z K, Peng T, Zhang W D, et al. Analysis of QTLs for root traits at seedling stage using an "Immortalized F2" population of wheat[J]. Acta Agronomic Sinica, 2010, 36(3): 442–448. [25] Cai H, Chu Q, Yuan L, et al. Identification of quantitative trait loci for leaf area and chlorophyll content in maize (Zea mays) under low nitrogen and low phosphorus supply[J]. Molecular Breeding, 2012, 30(7): 251–266.
[26] Liu Q, Qin J, Li T, et al. Fine mapping and candidate gene analysis of qSTL3, a stigma length-conditioning locus in rice (Oryza sativa L.)[J]. PloS One, 2015, 10: e0127938.
[27] Burton A L, Johnson J M, Foerster J M, et al. QTL mapping and phenotypic variation for root architectural traits in maize (Zea mays L.)[J]. Theoretical and Applied Genetics, 2014, 127(11): 2293–2311.
[28] Salvi S, Giuliani S, Ricciolini C, et al. Two major quantitative trait loci controlling the number of seminal roots in maize co-map with the root developmental genes rtcs and rum1[J]. Journal of Experimental Botany, 2016, 67(4): 1149–1159.
[29] 武良, 张卫峰, 陈新平, 等. 中国农田氮肥投入和生产效率[J]. 中国土壤与肥料, 2016, (4): 76–83. Wu L, Zhang W F, Chen X P, et al. Nitrogen fertilizer input and nitrogen use efficiency in Chinese farmland[J]. Soils and Fertilizers Sciences in China, 2016(4): 76–83. [30] 米国华, 陈范骏, 春亮, 等. 玉米氮高效品种的生物学特征[J]. 植物营养与肥料学报, 2007, 13(1): 155–159. Mi G H, Chen F J, Chun L, et al. Biological characteristics of nitrogen efficient maize genotypes[J]. Plant Nutrition and Fertilizer Science, 2007, 13(1): 155–159. [31] 米国华, 陈范骏, 吴秋平, 等. 玉米高效吸收氮素的理想根构型[J]. 中国科学(生命科学), 2010, 40(12): 1112–1116. Mi G H, Chen F J, Wu Q P, et al. Ideotype root architecture for efficient nitrogen acquisition by maize in intensive cropping systems[J]. Science China (Life Sciences), 2010, 40(12): 1112–1116. [32] 齐欢欢, 段利超, 胡伟, 等. 利用导入系群体对玉米产量及产量相关性状进行定位分析[J]. 玉米科学, 2013, 21(4): 24–27, 32. Qi H H, Duan L C, Hu W, et al. Identification of signification loci for yield and yield-related traits in maize with introgression lines[J]. Journal of Maize Science, 2013, 21(4): 24–27, 32. -
期刊类型引用(9)
1. 武阳春,高婷婷,侯宗运,杜金洹,代玉仙,任军,靳一男,刘岩峰,徐国良,李穆. 玉米自交系在苗期耐低氮能力的鉴定与筛选. 江苏农业科学. 2024(22): 62-68 .  百度学术
百度学术
2. 孙浩楠,曹霞,朱贵爽,李佳锴,周磊,吕叶,邱红梅,李志刚. 耐低氮大豆资源的苗期筛选与评价. 大豆科学. 2023(05): 545-553 .  百度学术
百度学术
3. 杨朔,胡仁,侯俊,李锦涛,黄飞,聂玺斌,胡洋,李炫,何文祥,孔小雁. 根区施肥条件下氮肥品种及施氮量对玉米苗期根系特征及生长的影响. 中国土壤与肥料. 2023(09): 111-119 .  百度学术
百度学术
4. 远月丽,易媛媛,战勇,陈李淼,袁松丽,黄毅,肖之源,张婵娟,周新安. 大豆氮高效种质苗期筛选与鉴定. 中国油料作物学报. 2022(03): 539-547 .  百度学术
百度学术
5. 宋岩,张锐,鱼尚奇,刘春花,高山,王雨,罗立新,张建良. 不同施氮水平对核桃砧木苗形态特征及生理特性的影响. 福建农业学报. 2020(03): 309-316 .  百度学术
百度学术
6. 梁媛媛,李东亚,许恒,王祎,汤继华,韩燕来. 高低氮条件下玉米SSSL群体生物量、氮浓度及氮累积量的QTL定位. 植物营养与肥料学报. 2020(07): 1226-1238 .  本站查看
本站查看
7. 李东亚,王祎,汤继华,许恒,谭金芳,韩燕来. 高氮和低氮条件下玉米穗位叶持绿性状的QTL定位. 植物营养与肥料学报. 2019(01): 115-122 .  本站查看
本站查看
8. 王峻,宋科,潘剑君,薛永,孙丽娟,蔡银美. 氮磷养分胁迫下小麦幼苗期生物学响应研究. 中国土壤与肥料. 2019(02): 152-158+164 .  百度学术
百度学术
9. 杨硕,金文俊,黄海蒙,王军,周得宝,赵阳阳,董召荣,宋贺. 耕层土层交换对土壤氮素关键转化过程和玉米氮素利用的影响. 中国生态农业学报(中英文). 2019(10): 1515-1527 .  百度学术
百度学术
其他类型引用(11)



 下载:
下载: