Response of nirS-type denitrifier community and network structures to long-term application of chemical fertilizers in a black soil of northeast China
-
摘要:目的
探明长期施用氮磷钾化肥对东北黑土农田土壤nirS型反硝化细菌群落和网络结构的影响,为更加合理的肥料配施提供理论依据。
方法基于农业农村部黑龙江耕地保育与农业环境科学观测实验站平台,选取不施肥 (NoF)、氮肥 (N)、磷肥 (P)、钾肥 (K)、氮钾肥 (NK)、氮磷肥 (NP)、磷钾肥 (PK)、氮磷钾肥 (NPK) 8个施肥处理,借助荧光定量PCR、Illumina MiSeq高通量测序和分子生态网络技术,分析东北黑土nirS型反硝化细菌丰度、群落及网络结构,及影响反硝化细菌群落变异的主要环境因子。
结果1) 长期施用氮肥均在显著增加nirS基因拷贝数的同时,降低了nirS型反硝化细菌的群落多样性,而磷、钾肥对其影响并不显著。2) Proteobacteria是所有处理中的优势反硝化细菌门,相对丰度为16.96%~27.34%,且氮肥的施用促进了隶属于该菌门中Bradyrhizobium的生长。3) PCoA分析结果显示,8个施肥处理nirS型反硝化细菌群落主要分成施氮肥和不施氮肥两组,说明长期氮肥的施用显著改变了东北黑土反硝化细菌的群落结构。结合Mantel test的结果可知,土壤pH是影响nirS型反硝化细菌群落改变的主要因素。4) 分别构建施氮肥和不施氮肥的反硝化细菌分子生态网络,发现施氮肥和不施氮肥网络结构存在很大差异,长期施用氮肥明显简化了nirS型反硝化细菌的网络结构,同时使其网络结构稳定性降低,易受外界环境扰动。
结论尽管施用化学氮肥有利于土壤养分的增加,但其土壤nirS型反硝化细菌群落及网络结构发生了较大改变,而磷、钾肥的施用对反硝化细菌群落无显著影响。本试验结果为进一步研究东北黑土区农田土壤反硝化微生物对不同施肥管理的响应机制提供了重要科学依据。
Abstract:ObjectiveThis study aimed to explore the changes in nirS-type denitrifier community and network structures in response to long-term chemical fertilization for the goal of providing scientific basis for rational fertilization strategies and soil quality improvement.
MethodsThe experiment was conducted in the Scientific Observation and Experiment Station of Arable Land Conservation and Agriculture Environment (Heilongjiang), Ministry of Agriculture and Rural Affairs. Bulk soil samples were collected from eight fertilization treatments, including non-fertilized control (NoF), N (nitrogen), P (phosphorus), K (potassium), NK, NP, PK and NPK. The nirS gene copy numbers, communities and network structures were analyzed with qPCR (quantitative real-time PCR), Illumina MiSeq sequencing and molecular ecological network. In addition, the soil properties were determined to analyze the links with the changes of nirS-type denitrifier communities.
Results1) All the N contained treatments, whether nitrogen alone or combined with phosphorus or potassium, significantly increased the nirS gene copy numbers and reduced the community diversity of nirS-type denitrifiers, while P and K treatments had little influence compared with control. 2) Proteobacteria was the only phylum detected in this study with the relative abundance of 16.96%–27.34%, and all the N contained treatments promoted the growth of Bradyrhizobium affiliated to this phylum. 3) The PCoA analysis showed that the whole nirS-type denitrifier communities could be clustered into two groups including N fertilization and non-N fertilization, which indicated that long-term application of N fertilizer significantly changed the community structures of nirS-type denitrifier. Mantel test analysis revealed that soil pH was the most important factor in shifting nirS-type denitrifier communities. 4) Networks of N contained treatments and non-N contained ones were separately constructed based on sequencing data, and large differences were observed between the network structures of the two groups. Long-term N fertilization simplified the network structure of nirS-type denitrifiers, and decreased the network stability with more influence from environmental disturbances.
ConclusionsLong-term N fertilization, rather than P and K fertilizers, has larger influences on community and network structures of nirS-type denitrifier. The results may provide a new insight for understanding the changes of nirS-type denitrifier communities in response to different fertilization managements in a black soil of northeast China.
-
反硝化作用既是农田生态系统中氮素损失的重要途径,又是温室气体N2O的主要来源[1]。反硝化过程是在反硝化微生物作用下,由硝酸盐还原酶、亚硝酸盐还原酶、NO还原酶和N2O还原酶的连续催化下完成的[2]。其中,亚硝酸盐还原为NO反应是区分反硝化作用和其他硝酸盐代谢作用的标志性反应,因此,编码亚硝酸盐还原酶 (Nir) 的功能基因可作为研究反硝化微生物的主要分子标记[3-4]。亚硝酸还原酶包含结构不同但功能相同的两种酶,一种由含细胞色素cd1的nirS基因编码,另一种由含铜基的nirK基因编码[3]。这两种基因均适用于环境中反硝化微生物多样性和群落结构的研究[5-6]。
施肥是影响土壤质量和生产力最重要的农业措施之一,在改善土壤结构、养分含量和作物产量的同时,也会对土壤中反硝化微生物数量和群落结构产生深刻影响[7-8]。近年来,发现不同施肥处理对反硝化微生物群落的影响存在很大差异[9-10]。Chen等[11]通过长期定位试验,发现尿素的施用能够显著增加红壤中nirS型反硝化细菌的丰度,但对nirS型反硝化细菌的群落结构无显著影响。Tang等[10]研究发现,施用高量氮肥会降低杉木人工林土壤中nirS型反硝化细菌的丰度,而土壤速效磷含量却与nirS型反硝化细菌的基因拷贝数呈显著正相关关系。Yang等[12]发现,施用高量氮肥显著降低碱地土壤中nirS型反硝化细菌丰度并改变其群落结构,土壤pH、全氮、铵态氮和硝态氮的含量是影响nirS型反硝化细菌群落结构的主要因素。因此,土壤性质、施肥处理和土壤类型均对土壤中反硝化微生物群落产生显著影响。
作为世界四大黑土带之一的我国东北黑土,是我国重要的商品粮生产基地[13]。随着该地区粮食产量的不断增加,施肥等农业措施的应用也不断强化,化学肥料对土壤质量的不利影响也逐渐显现[14]。然而,针对东北黑土区反硝化微生物对长期施用化肥的响应研究却鲜有报道,或仅采用传统分子生物手段,如T-RFLP等[15],但具有一定局限性。此外,基于随机矩阵理论的分子生态网络技术已被广泛应用于土壤细菌和真菌网络结构研究[16-17],但借助该技术对东北黑土反硝化微生物的网络构建研究还未见报道。
试验以农业农村部黑龙江耕地保育与农业环境科学观测实验站为依托,通过qPCR、高通量测序技术以及分子生态网络平台,研究长期施用化肥对东北黑土nirS型反硝化细菌丰度、群落和网络结构的影响,为进一步阐明化肥调控农田土壤反硝化细菌结构,优化该地区养分资源利用管理提供一定科学参考。
1. 材料与方法
1.1 试验地概况和土壤样品采集
试验地点位于黑龙江哈尔滨市民主乡 (45°50′N,126°51′E)。土壤类型为典型黑土,成土母质为洪积黄土状粘土,试验地地势平坦,海拔高度151 m,气候属温带大陆性季风气候,年平均气温3.5℃,年降水量533 mm。本文涉及的长期施肥处理起始于1980年,轮作方式为小麦–大豆–玉米轮作,当季作物为大豆。试验地面积8060 m2,小区面积168 m2。氮、磷、钾肥分别为尿素、三料磷肥和硫酸钾,施用量分别为N 75 kg/hm2、P2O5 150 kg/hm2、K2O 75 kg/hm2。
于2014年9月4日在大豆鼓粒期 (R6) 采集0—20 cm耕层土壤样品。试验共设8个处理,包括不施肥 (NoF)、氮肥 (N)、磷肥 (P)、钾肥 (K)、氮钾肥 (NK)、氮磷肥 (NP)、磷钾肥 (PK)、氮磷钾肥 (NPK)。每个处理3个重复,共采集24个样品。样品采集后立即过2 mm筛,一部分保存于–80℃用于土壤总DNA的提取,另一部分室温风干用于土壤理化性质的测定,其中用于测定铵态氮和硝态氮含量的土壤样品为新鲜土样。
1.2 土壤总DNA提取,qPCR和Illumina MiSeq测序
利用FastDNA® SPIN Kit for Soil (MP Biomedicals,USA) 试剂盒,每个样品称取0.5 g土壤,按照说明提取土壤总DNA,之后采用NanoDrop 2000 spectrophotometer (Thermo Scientific,USA) 检测所提取总DNA的浓度和纯度。于–20℃冰箱中保存。
采用引物cd3aF (5′–GTSAACGTSAAGGARACSGG–3′) 和R3cd (5′–GASTTCGGRTGSGTCTTGA–3′) 对nirS基因进行扩增[18]。qPCR标准曲线采用阳性克隆质粒逐步稀释至104~109拷贝,保存于–20℃备用。qPCR反应在LightCycler® 480上进行。20 μL反应体系包括:10 μL SYBR Premix Ex TaqTM,7 μL灭菌高纯水,20 μnol/L的引物cd3aF和R3cd各1 μL,1 μL 10倍稀释的DNA模板。反应程序为:95℃预变性30 s,95℃变性5 s,60℃退火和延伸各30 s,共40个循环。
样品总DNA送至上海美吉生物公司,应用Illumina MiSeq测序仪对nirS基因 (cd3aF–R3cd) 进行测序。高通量测序步骤及数据质控参见文献[19]。将质控后的序列在FunGene平台[20]上进行翻译,将不能翻译成亚硝酸盐还原酶的序列去除,剩余的高质量序列利用UPARSE平台[21]按97%相似性聚类成OTU。对比GenBank数据库,得到每个OTU对应的物种分类信息。所测全部序列已提交至GenBank SRA (Sequence Read Archive),注册号为SRP175056。
1.3 分子生态网络构建
基于上述高通量测序获取的数据,在Molecular Ecological Network Analysis Pipeline (MENA) (http://ieg4.rccc.ou.edu/mena) 网站上进行反硝化细菌网络构建和参数获取[22-24]。具体步骤和参数解释见文献[25]。利用Cytoscape 3.4.0软件对获取的网络文件进行可视化处理,从而得到网络结构图及其相关特征参数。
1.4 数据分析
主坐标分析 (Principal coordinate analysis,PCoA)、Mantel test分析和分层聚类 (Hierarchical clustering) 分别使用R软件 (Version 3.2.5) (R Development Core Team,2016) 中的ape和vegan数据包进行。单因素ANOVA和Pearson相关性分析使用IBM Statistical Product and Service Solutions (SPSS) Statistics for Windows (Version 19.0),用Duncan显著差异法分析处理间在P < 0.05和P < 0.01水平上的差异显著性。
2. 结果与分析
2.1 不同处理的土壤理化性质
长期化学肥料的施用显著改变了黑土的理化性质 (表1)。与不施肥相比,所有施肥处理均降低了土壤pH,其中4个氮肥处理的土壤pH均降低到5.00左右,加剧了土壤的酸化(P < 0.05)。与不施肥对照相比,施用化学肥料均提高了土壤全碳、全氮和硝态氮的含量(P < 0.05),且氮肥的施用对土壤碳、氮养分的增加更为显著。此外,与不施肥相比,磷、钾肥的施用均显著增加相应的土壤全磷、有效磷、全钾和速效钾含量。
表 1 长期施用不同化肥处理的东北黑土理化性质Table 1. Physical and chemical properties of black soil in northeast China after long-term chemical fertilization处理
TreatmentpH
(1∶2.5 CaCl2)全碳
Total C
(g/kg)全氮
Total N
(g/kg)全磷
Total P
(g/kg)全钾
Total K
(g/kg)铵态氮
NH4+-N
(mg/kg)硝态氮
NO3–-N
(mg/kg)有效磷
Available P
(mg/kg)速效钾
Available K
(mg/kg)NoF 6.00 a 16.4 d 1.02 d 0.36 ef 18.6 b 54.90 ab 6.2 f 9.3 e 187 de P 5.89 b 17.5 abc 1.08 c 0.68 a 21.1 a 53.60 ab 7.0 e 100 a 173 e K 5.99 ab 16.9 bcd 1.10 c 0.38 de 21.1 a 54.16 ab 7.8 d 12.8 de 264 a PK 5.77 c 16.9 cd 1.11 c 0.59 b 21.9 a 52.81 b 7.1 de 72.3 b 172 e N 5.01 d 17.9 a 1.18 b 0.32 f 21.3 a 56.07 ab 10.1 b 8.5 e 183 de NK 5.11 d 18.2 a 1.24 a 0.40 d 21.4 a 56.68 a 9.2 c 23.1 d 198 cd NP 5.10 d 18.2 a 1.20 ab 0.45 c 21.7 a 54.90 ab 10.8 a 34.8 c 211 bc NPK 5.02 d 17.8 ab 1.20 ab 0.38 d 21.1 a 55.27 ab 8.9 c 21.9 d 226 b P-value < 0.001 0.002 < 0.001 < 0.001 0.004 0.178 < 0.001 < 0.001 < 0.001 注(Note):同列数值后不同小写字母表示处理间差异显著 (P<0.05) Values followed by different letters in a column indicate signficant difference among treatments (P<0.05). 2.2 不同处理反硝化细菌的丰度
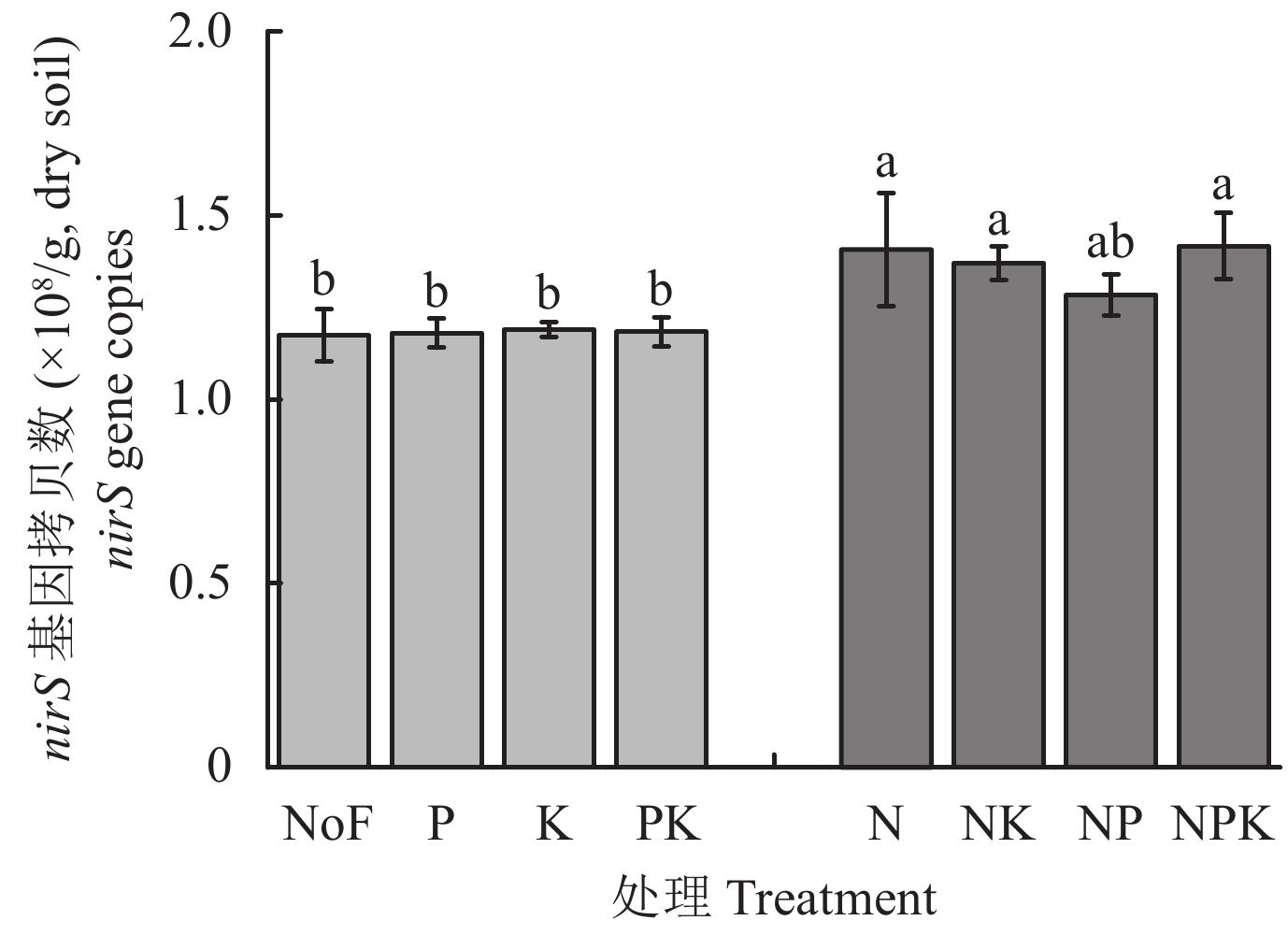
对反硝化细菌nirS基因拷贝数进行定量分析 (图1),表明8个处理的nirS基因拷贝数为1.17 × 108~1.42 × 108。P、K和PK处理的nirS基因拷贝数与NoF处理相比无显著差异,而N、NK和NPK处理的nirS基因拷贝数均显著高于NoF处理(P < 0.05)。
2.3 不同处理反硝化细菌的群落结构
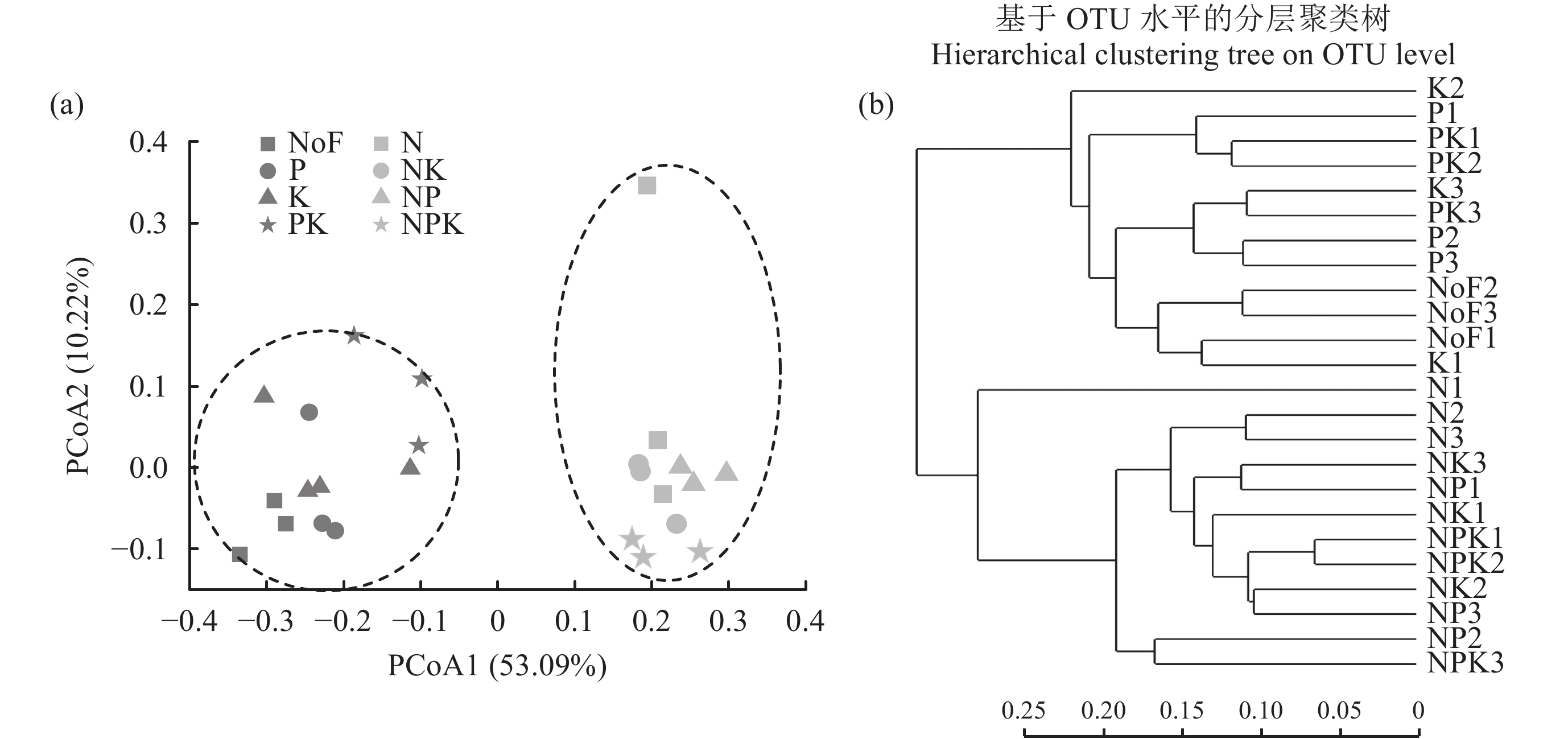
对24个土壤样品测序共获得了337381条有效序列,每个样品9053~18461条、平均14058条,在97%相似度下聚类得到396个OTU。nirS型反硝化细菌在门纲目科属上的主要分类如表2。在8个处理中只检测到一个门,为变形菌门(Proteobacteria),其相对丰度为17.14%~27.34%,其余序列为未分类nirS基因序列。不同处理之间Proteobacteria相对丰度无显著差异(P=0.492)。在属水平上,与NoF处理相比,施用磷钾肥降低了Bradyrhizobium的相对丰度,而4个氮肥处理均增加了该菌的相对丰度。与之相反,除NPK处理外,其余氮肥处理Rhodanobacter的相对丰度均低于磷、钾肥和NoF处理。由PCoA分析可以看出 (图2a),2轴共解释了63.31%的群落变异,并且8个处理沿PCoA1轴分成两个大组,其中NoF处理与P、K和PK处理聚在一起,而N、NK、NP和NPK处理聚在一起,分层聚类树中也可以看到类似的结果 (图2b)。说明氮肥的施用导致了东北黑土nirS型反硝化细菌群落的显著改变,而磷、钾肥对其影响不显著。
表 2 长期施用不同化肥处理的东北黑土nirS型反硝化细菌门、纲、目、科和属的相对丰度 (%)Table 2. Relative abundances of nirS-type denitrifying bacterial phyla, class, order, family and genus in a black soil of northeast China after long-term chemical fertilizationTaxa分类 NoF P K PK N NK NP NPK P-value 门Phylum 变形菌门Proteobacteria 22.27 a 24.73 a 23.61 a 27.34 a 27.22 a 16.96 a 17.14 a 20.62 a 0.492 纲Class α-变形菌纲Alphaproteobacteria 1.51 ab 0.49 b 0.52 ab 1.32 ab 2.60 a 2.98 a 2.34 ab 2.89 a 0.029 β-变形菌Betaproteobacteria 0.61 a 1.01 a 1.38 a 0.82 a 13.57 a 2.35 a 3.35 a 1.16 a 0.384 γ-变形菌Gammaproteobacteria 8.99 bc 12.70 ab 8.34 bcd 16.18 a 3.51 d 5.50 cd 5.18 cd 10.08 bc 0.001 目Order 根瘤菌目Rhizobiales 1.51 ab 0.48 b 0.52 b 1.32 ab 2.60 a 2.98 a 2.34 ab 2.89 a 0.028 伯克氏菌目Burkholderiales 0.24 a 0.27 a 0.72 a 0.21 a 0.35 a 0.25 a 0.08 a 0.39 a 0.269 红环菌目Rhodocyclales 0.07 a 0.32 a 0.15 a 0.18 a 0.27 a 0.04 a 0.00 a 0.04 a 0.181 黄单胞菌目Xanthomonadales 8.99 bc 12.70 ab 8.34 bcd 16.18 a 3.51 d 5.50 cd 5.18 cd 10.08 bc 0.001 科Family 慢生根瘤菌科Bradyrhizobiaceae 1.51 ab 0.48 b 0.52 b 1.32 ab 2.60 a 2.98 a 2.34 ab 2.89 a 0.028 伯克氏菌科Burkholderiaceae 0.00 a 0.00 a 0.00 a 0.00 a 0.00 a 0.00 a 0.00 a 0.00 a 0.466 草酸杆菌科Oxalobacteraceae 0.10 ab 0.08 ab 0.51 a 0.13 ab 0.18 ab 0.01 b 0.01 b 0.01 b 0.216 红环菌科Rhodocyclaceae 0.07 ab 0.32 a 0.15 ab 0.18 ab 0.27 ab 0.04 ab 0.00 b 0.04 ab 0.181 黄单胞菌科Xanthomonadaceae 8.99 bc 12.70 ab 8.34 bcd 16.18 a 3.51 d 5.50 cd 5.18 cd 10.08 bc 0.001 属Genus 慢生根瘤菌属Bradyrhizobium 1.5 ab 0.48 b 0.52 b 1.32 ab 2.60 a 2.98 a 2.34 ab 2.89 a 0.028 固氮螺菌属Azospira 0.04 a 0.14 a 0.09 a 0.04 a 0.04 a 0.01 a 0.00 a 0.00 a 0.507 固氮弓菌属Azoarcus 0.03 a 0.16 a 0.04 a 0.12 a 0.22 a 0.03 a 0.00 a 0.00 a 0.233 草螺菌属Herbaspirillum 0.10 ab 0.08 ab 0.51 a 0.13 ab 0.18 ab 0.011 b 0.01 b 0.01 b 0.216 红长命菌属Rubrivivax 0.06 ab 0.08 a 0.10 a 0.06 ab 0.00 b 0.00 b 0.00 b 0.00 b 0.010 产黄杆菌属Rhodanobacter 8.99 bc 12.70 ab 8.34 bcd 16.18 a 3.51 d 5.50 cd 5.18 cd 10.08 bc 0.001 注(Note):同列数值后不同小写字母表示处理间差异显著 (P<0.05) Values followed by different letters in a column indicate signficant difference among treatments (P<0.05). 2.4 不同处理反硝化细菌的多样性指数
由不同施肥处理土壤nirS型反硝化细菌的多样性指数OTUs、ACE、Chao、Shannon和Simpson (表3) 表明,与NoF处理相比,4个含氮肥处理均降低了nirS型反硝化细菌的多样性,而磷钾处理的多样性指数更接近于NoF处理的水平,说明长期施用氮肥降低了nirS型反硝化细菌多样性,而长期施用磷、钾肥无显著影响。
表 3 长期施用不同化肥处理的东北黑土nirS型反硝化细菌的多样性指数Table 3. α-Diversity index of nirS-type denitrifying bacteria in a black soil of northeast China after long-term chemical fertilization处理
TreatmentOTUs 丰富度Richness 多样性Diversity ACE Chao Shannon Simpson NoF 166 a 191 a 192 a 3.76 a 0.0355 c P 158 a 172 b 174 ab 3.67 ab 0.0455 c K 153 ab 166 bc 174 ab 3.69 a 0.0421 c PK 154 ab 176 ab 174 ab 3.43 bc 0.0641 bc N 142 bc 163 bc 170 ab 3.22 cd 0.0896 ab NK 136 c 158 bc 154 b 3.29 c 0.0718 b NP 129 c 148 c 148 b 2.94 e 0.1207 a NPK 128 c 150 c 152 b 3.00 de 0.1133 a P-value < 0.001 0.002 0.028 < 0.001 0.001 注(Note):同列数值后不同小写字母表示处理间差异显著 (P<0.05) Values followed by different letters in a column indicate signficant difference among treatments (P<0.05). 2.5 土壤反硝化细菌群落与理化性质的关系
采用Mantel分析检测了土壤理化性质与nirS型反硝化细菌群落变化之间的关系 (表4)。在所检测的9个土壤理化性质中,土壤pH、全碳、全氮、铵态氮和硝态氮含量显著影响该地区nirS型反硝化细菌的群落变异,其中土壤pH是引起群落结构改变的最主要因素。而全磷、全钾、有效磷和速效钾含量及其交互效应对nirS型反硝化细菌群落没有显著影响。该结果进一步说明长期施用氮肥,以及由氮肥引起的土壤理化性质的改变对东北黑土nirS型反硝化细菌群落产生深刻影响。
表 4 东北黑土土壤理化性质与nirS型反硝化细菌群落结构的Mantel分析Table 4. Mantel test of the correlations between soil properties and community structures of nirS-type denitrifying bacteria in a black soil of northeast China理化性质Soil property r P pH 0.838** 0.001 全碳 Total C (TC) 0.491** 0.001 全氮 Total N (TN) 0.654** 0.001 全磷 Total P (TP) 0.111 0.085 全钾 Total K (TK) 0.120 0.123 铵态氮NH4+-N 0.189* 0.021 硝态氮NO3–-N 0.658** 0.001 有效磷Available P (AP) 0.098 0.126 速效钾 Available K (AK) 0.125 0.087 TN × NH4+-N × NO3–-N 0.360** 0.001 TP × TK –0.002 0.986 AP × AK 0.038 0.750 TP × TK × AP × AK 0.039 0.757 注(Note):**—P<0.01; *—P<0.05. 2.6 东北黑土反硝化细菌的网络结构
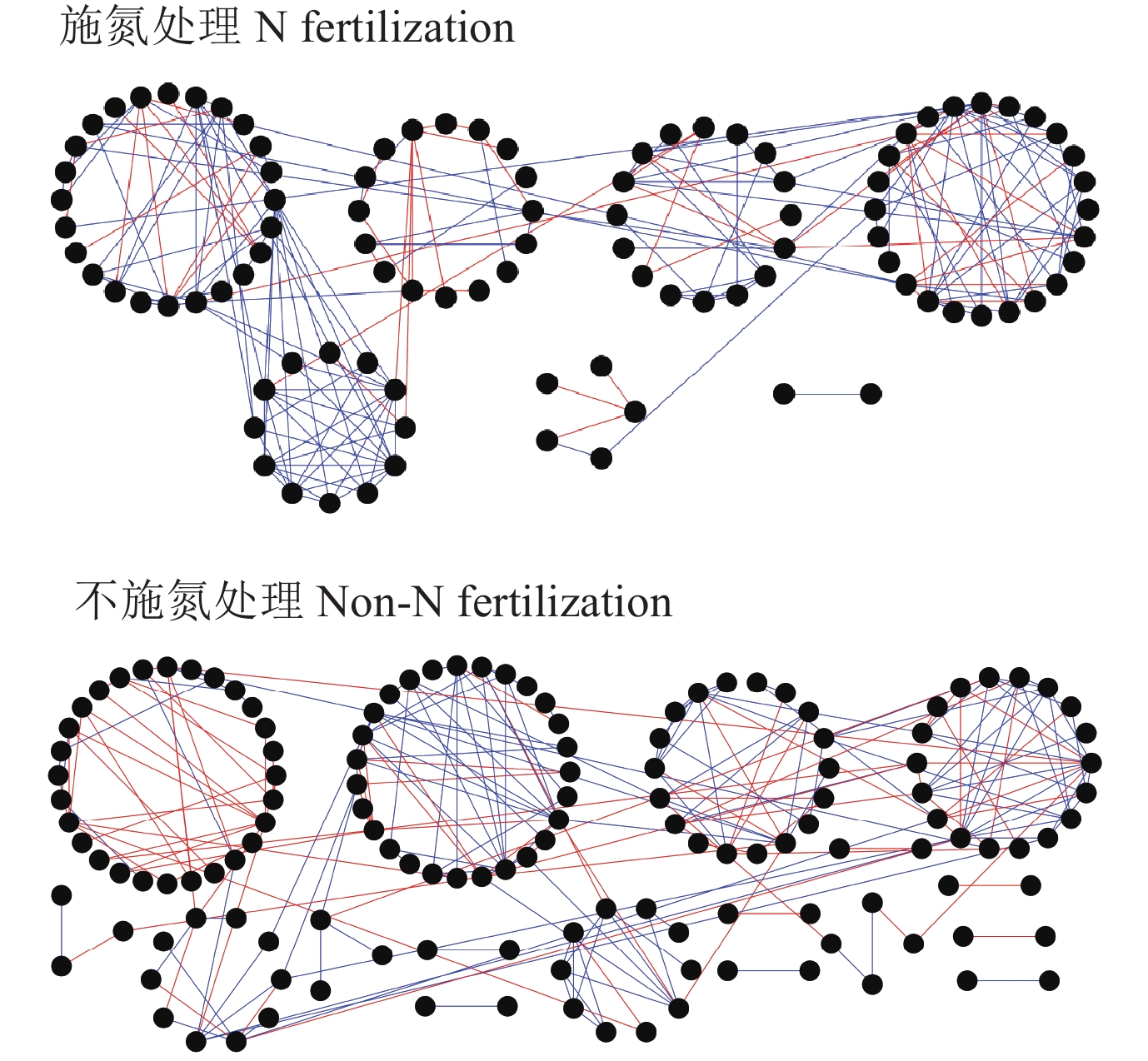
基于上述nirS型反硝化细菌的高通量测序结果,长期施用氮肥显著影响了反硝化细菌的群落,而磷钾肥对其无明显影响。故此,将4个施氮肥处理和4个不施氮肥处理看作两个整体即两大处理,分别利用各自的12个样品构建氮肥网络和不施氮肥网络 (图3)。由图3可以看出,施用氮肥处理的网络节点数和连线数明显少于不施氮肥处理的网络,且施用氮肥处理网络中节点之间的负相关连线占比 (0.27) 低于不施氮肥处理的网络 (0.41)。进一步对两个网络的相关参数进行解析 (表5),发现施用氮肥和不施氮肥处理网络的模块性均大于0.30,可以很好的表征网络特性[26]。其中施用氮肥处理的网络平均连通度,平均路径长和聚集系数均大于不施氮肥处理的网络,而不施氮肥处理的网络具有更高的模块性和更多的模块数。以上结果表明,长期化学氮肥的施用显著改变了nirS型反硝化细菌的网络结构和特性。
![]() 图 3 东北黑土施氮和不施氮处理nirS型反硝化细菌的网络结构[注(Note):每个节点代表1个OTU,也就是相应的反硝化细菌物种;蓝色线代表物种间的正相关关系,红色线代表物种间的负相关关系 Each node signifies an OTU,which could correspond to a denitrifying bacterial species;blue lines represent positive interactions between two individual nodes,while red lines represent negative interactions.]Figure 3. Network structures of nirS-type denitrifying bacteria under N and non-N fertilization in a black soil of northeast China表 5 东北黑土施氮和不施氮处理nirS型反硝化细菌网络参数Table 5. Parameters of nirS-type denitrifying bacterial network under N and non-N fertilizationin a black soil of northeast China
图 3 东北黑土施氮和不施氮处理nirS型反硝化细菌的网络结构[注(Note):每个节点代表1个OTU,也就是相应的反硝化细菌物种;蓝色线代表物种间的正相关关系,红色线代表物种间的负相关关系 Each node signifies an OTU,which could correspond to a denitrifying bacterial species;blue lines represent positive interactions between two individual nodes,while red lines represent negative interactions.]Figure 3. Network structures of nirS-type denitrifying bacteria under N and non-N fertilization in a black soil of northeast China表 5 东北黑土施氮和不施氮处理nirS型反硝化细菌网络参数Table 5. Parameters of nirS-type denitrifying bacterial network under N and non-N fertilizationin a black soil of northeast China处理
Treatment阈值
Similarity threshold
(St)节点数
Network size
(n)连线数
Link number平均连通度
Average connectivity
(avgK)平均路径长
Average geodesic distance
(GD)平均聚集系数
Average clustering coefficient
(avgCC)模块性 (模块数)
Modularity
(No. of modules)R2 of
power-law施氮处理
N fertilization
(N,NK,NP,NPK)0.70 99 192 3.88 4.90 0.231 0.618 (7) 0.734 不施氮处理
Non-N fertilization
(NoF,P,K,PK)0.71 137 229 3.34 4.62 0.166 0.644 (17) 0.847 3. 讨论
前期我们利用高通量测序和分子生态网络技术对长期化肥处理东北黑土细菌群落进行研究[27],发现氮肥而不是磷钾肥对东北黑土细菌群落结构产生显著影响,且长期施用氮肥简化了细菌的网络结构。微生物介导的反硝化作用是农田土壤氮素转化的重要过程之一,对氮肥利用率、硝酸盐淋失和温室气体N2O排放等农业及环境问题产生直接影响。目前,利用新一代测序技术解析长期施用化肥对东北黑土反硝化细菌群落和网络结构的影响还鲜有报道。本试验通过对亚硝酸还原酶nirS基因的研究,发现化学氮肥的长期施用在提高土壤全碳、全氮和硝态氮等养分的同时,显著降低了土壤pH,造成土壤酸化[27]。硝态氮的积累为反硝化微生物提供充足的反应底物进而促进其生长[2],使得所有氮肥处理中nirS基因拷贝数均高于不施氮肥处理。尽管所检测到的大多数nirS基因序列没有明确的分类信息,但我们发现Proteobacteria是东北农田土壤中主要的反硝化细菌门,这与大多数研究结果相一致[8, 28]。然而,与废水、底泥、稻田土壤等环境中主要的反硝化细菌属为Rhodocyclales和Burkholderiales等不同[11, 28-29],东北黑土中主要的反硝化细菌属为Bradyrhizobium和Rhodanobacter。值得注意的是,本试验样品采集时的当季作物是大豆,Bradyrhizobium通常作为豆科植物的共生固氮微生物定植于土壤中[30],但有研究发现隶属于该属的一些菌种也包含nirS基因,在低氧条件下可进行反硝化作用[31]。Bradyrhizobium大多属于异养微生物且喜酸性环境[32-33],因此,氮肥处理土壤碳氮等养分的增加及土壤pH的降低促进了该菌的大量生长。
尽管长期施用化学氮肥显著增加了东北黑土nirS型反硝化细菌的丰度,但降低了反硝化细菌的多样性,这与Tao等[34]对石灰土中反硝化细菌的研究结果相反。产生这种不同的原因可能与土壤pH有关,石灰土的pH呈碱性,而本研究中4个氮肥处理的土壤pH均在5.0左右,并且已有大量研究报道指出酸性土壤中细菌群落多样性与土壤pH具有显著地正相关关系[35-36]。同时,土壤pH也是影响东北黑土nirS型反硝化细菌群落结构的最主要因素。4个氮肥处理引起的土壤酸化使反硝化细菌群落与不施氮肥处理具有明显差异,这也与之前我们对该地区土壤细菌群落的研究结果相一致。此外,作为核酸主要成分之一的磷元素和维持细胞膜活性的钾元素的增加[37],在促进作物生长对磷钾元素需求的同时,也会对土壤微生物产生一定影响。但长期磷钾肥的施用并没有显著影响东北黑土nirS型反硝化细菌的多样性和群落结构。有研究指出,与nirK基因相比,nirS基因对磷的添加较不敏感,这可能是由于两种基因型的反硝化细菌生态位存在差异[10]。因此,还需对不同施肥处理nirK型反硝化细菌群落作进一步研究,以期全面掌握东北黑土反硝化微生物群落对长期不同施肥管理的响应特征。
在自然环境中,任何物种不能单独存在,而是与其他物种形成复杂的生态网络[38]。分析生态网络的结构特性不仅可以揭示物种之间的复杂关系,还可以表征生态网络结构的稳定性[39-40]。本研究对nirS型反硝化细菌的生态网络进行构建,发现长期施用氮肥明显简化了nirS型反硝化细菌的网络结构。氮肥网络比无氮肥网络具有更高的平均连通度和聚集系数,表明氮肥网络中节点之间的联系更加紧密,同时,紧密联系在一起的氮肥网络所具有的较低模块性也使其更易受环境波动的影响[41-42],而氮肥的施用导致的反硝化细菌网络结构稳定性的降低很可能是引起其群落多样性降低以及网络结构简化的关键[43],可能对维持土壤微生物生态系统的健康性和稳定性造成不利影响。此外,不施氮肥网络节点之间的负相关关系占比明显高于施氮肥网络,可能是由于不施氮肥处理土壤氮含量普遍较低,物种之间出现较强的养分竞争关系。
4. 结论
本研究通过对长期化肥施用下东北黑土中nirS型反硝化细菌群落和网络特征的研究,发现长期单独施用化学氮肥或与磷、钾肥配施,在显著增加土壤碳、氮养分含量的同时也导致了土壤酸化加剧。施用化学氮肥尽管显著增加了东北黑土nirS基因的拷贝数,但降低了nirS型反硝化细菌群落多样性并改变了其群落结构。长期施用氮肥引起的土壤pH改变,是影响该地区反硝化细菌群落变异的最主要因素。施氮肥和不施氮肥处理的nirS型反硝化细菌生态网络结构存在很大差异,施用氮肥降低了nirS型反硝化细菌网络结构的稳定性,并简化了其网络结构。本研究表明,长期施用氮肥比磷、钾肥对东北黑土nirS型反硝化细菌群落和网络结构的影响更为显著。该研究为东北黑土农田长期不同化肥管理介导nirS型反硝化细菌群落变化过程提供了一定的理论依据。
-
图 3 东北黑土施氮和不施氮处理nirS型反硝化细菌的网络结构
[注(Note):每个节点代表1个OTU,也就是相应的反硝化细菌物种;蓝色线代表物种间的正相关关系,红色线代表物种间的负相关关系 Each node signifies an OTU,which could correspond to a denitrifying bacterial species;blue lines represent positive interactions between two individual nodes,while red lines represent negative interactions.]
Figure 3. Network structures of nirS-type denitrifying bacteria under N and non-N fertilization in a black soil of northeast China
表 1 长期施用不同化肥处理的东北黑土理化性质
Table 1 Physical and chemical properties of black soil in northeast China after long-term chemical fertilization
处理
TreatmentpH
(1∶2.5 CaCl2)全碳
Total C
(g/kg)全氮
Total N
(g/kg)全磷
Total P
(g/kg)全钾
Total K
(g/kg)铵态氮
NH4+-N
(mg/kg)硝态氮
NO3–-N
(mg/kg)有效磷
Available P
(mg/kg)速效钾
Available K
(mg/kg)NoF 6.00 a 16.4 d 1.02 d 0.36 ef 18.6 b 54.90 ab 6.2 f 9.3 e 187 de P 5.89 b 17.5 abc 1.08 c 0.68 a 21.1 a 53.60 ab 7.0 e 100 a 173 e K 5.99 ab 16.9 bcd 1.10 c 0.38 de 21.1 a 54.16 ab 7.8 d 12.8 de 264 a PK 5.77 c 16.9 cd 1.11 c 0.59 b 21.9 a 52.81 b 7.1 de 72.3 b 172 e N 5.01 d 17.9 a 1.18 b 0.32 f 21.3 a 56.07 ab 10.1 b 8.5 e 183 de NK 5.11 d 18.2 a 1.24 a 0.40 d 21.4 a 56.68 a 9.2 c 23.1 d 198 cd NP 5.10 d 18.2 a 1.20 ab 0.45 c 21.7 a 54.90 ab 10.8 a 34.8 c 211 bc NPK 5.02 d 17.8 ab 1.20 ab 0.38 d 21.1 a 55.27 ab 8.9 c 21.9 d 226 b P-value < 0.001 0.002 < 0.001 < 0.001 0.004 0.178 < 0.001 < 0.001 < 0.001 注(Note):同列数值后不同小写字母表示处理间差异显著 (P<0.05) Values followed by different letters in a column indicate signficant difference among treatments (P<0.05). 表 2 长期施用不同化肥处理的东北黑土nirS型反硝化细菌门、纲、目、科和属的相对丰度 (%)
Table 2 Relative abundances of nirS-type denitrifying bacterial phyla, class, order, family and genus in a black soil of northeast China after long-term chemical fertilization
Taxa分类 NoF P K PK N NK NP NPK P-value 门Phylum 变形菌门Proteobacteria 22.27 a 24.73 a 23.61 a 27.34 a 27.22 a 16.96 a 17.14 a 20.62 a 0.492 纲Class α-变形菌纲Alphaproteobacteria 1.51 ab 0.49 b 0.52 ab 1.32 ab 2.60 a 2.98 a 2.34 ab 2.89 a 0.029 β-变形菌Betaproteobacteria 0.61 a 1.01 a 1.38 a 0.82 a 13.57 a 2.35 a 3.35 a 1.16 a 0.384 γ-变形菌Gammaproteobacteria 8.99 bc 12.70 ab 8.34 bcd 16.18 a 3.51 d 5.50 cd 5.18 cd 10.08 bc 0.001 目Order 根瘤菌目Rhizobiales 1.51 ab 0.48 b 0.52 b 1.32 ab 2.60 a 2.98 a 2.34 ab 2.89 a 0.028 伯克氏菌目Burkholderiales 0.24 a 0.27 a 0.72 a 0.21 a 0.35 a 0.25 a 0.08 a 0.39 a 0.269 红环菌目Rhodocyclales 0.07 a 0.32 a 0.15 a 0.18 a 0.27 a 0.04 a 0.00 a 0.04 a 0.181 黄单胞菌目Xanthomonadales 8.99 bc 12.70 ab 8.34 bcd 16.18 a 3.51 d 5.50 cd 5.18 cd 10.08 bc 0.001 科Family 慢生根瘤菌科Bradyrhizobiaceae 1.51 ab 0.48 b 0.52 b 1.32 ab 2.60 a 2.98 a 2.34 ab 2.89 a 0.028 伯克氏菌科Burkholderiaceae 0.00 a 0.00 a 0.00 a 0.00 a 0.00 a 0.00 a 0.00 a 0.00 a 0.466 草酸杆菌科Oxalobacteraceae 0.10 ab 0.08 ab 0.51 a 0.13 ab 0.18 ab 0.01 b 0.01 b 0.01 b 0.216 红环菌科Rhodocyclaceae 0.07 ab 0.32 a 0.15 ab 0.18 ab 0.27 ab 0.04 ab 0.00 b 0.04 ab 0.181 黄单胞菌科Xanthomonadaceae 8.99 bc 12.70 ab 8.34 bcd 16.18 a 3.51 d 5.50 cd 5.18 cd 10.08 bc 0.001 属Genus 慢生根瘤菌属Bradyrhizobium 1.5 ab 0.48 b 0.52 b 1.32 ab 2.60 a 2.98 a 2.34 ab 2.89 a 0.028 固氮螺菌属Azospira 0.04 a 0.14 a 0.09 a 0.04 a 0.04 a 0.01 a 0.00 a 0.00 a 0.507 固氮弓菌属Azoarcus 0.03 a 0.16 a 0.04 a 0.12 a 0.22 a 0.03 a 0.00 a 0.00 a 0.233 草螺菌属Herbaspirillum 0.10 ab 0.08 ab 0.51 a 0.13 ab 0.18 ab 0.011 b 0.01 b 0.01 b 0.216 红长命菌属Rubrivivax 0.06 ab 0.08 a 0.10 a 0.06 ab 0.00 b 0.00 b 0.00 b 0.00 b 0.010 产黄杆菌属Rhodanobacter 8.99 bc 12.70 ab 8.34 bcd 16.18 a 3.51 d 5.50 cd 5.18 cd 10.08 bc 0.001 注(Note):同列数值后不同小写字母表示处理间差异显著 (P<0.05) Values followed by different letters in a column indicate signficant difference among treatments (P<0.05). 表 3 长期施用不同化肥处理的东北黑土nirS型反硝化细菌的多样性指数
Table 3 α-Diversity index of nirS-type denitrifying bacteria in a black soil of northeast China after long-term chemical fertilization
处理
TreatmentOTUs 丰富度Richness 多样性Diversity ACE Chao Shannon Simpson NoF 166 a 191 a 192 a 3.76 a 0.0355 c P 158 a 172 b 174 ab 3.67 ab 0.0455 c K 153 ab 166 bc 174 ab 3.69 a 0.0421 c PK 154 ab 176 ab 174 ab 3.43 bc 0.0641 bc N 142 bc 163 bc 170 ab 3.22 cd 0.0896 ab NK 136 c 158 bc 154 b 3.29 c 0.0718 b NP 129 c 148 c 148 b 2.94 e 0.1207 a NPK 128 c 150 c 152 b 3.00 de 0.1133 a P-value < 0.001 0.002 0.028 < 0.001 0.001 注(Note):同列数值后不同小写字母表示处理间差异显著 (P<0.05) Values followed by different letters in a column indicate signficant difference among treatments (P<0.05). 表 4 东北黑土土壤理化性质与nirS型反硝化细菌群落结构的Mantel分析
Table 4 Mantel test of the correlations between soil properties and community structures of nirS-type denitrifying bacteria in a black soil of northeast China
理化性质Soil property r P pH 0.838** 0.001 全碳 Total C (TC) 0.491** 0.001 全氮 Total N (TN) 0.654** 0.001 全磷 Total P (TP) 0.111 0.085 全钾 Total K (TK) 0.120 0.123 铵态氮NH4+-N 0.189* 0.021 硝态氮NO3–-N 0.658** 0.001 有效磷Available P (AP) 0.098 0.126 速效钾 Available K (AK) 0.125 0.087 TN × NH4+-N × NO3–-N 0.360** 0.001 TP × TK –0.002 0.986 AP × AK 0.038 0.750 TP × TK × AP × AK 0.039 0.757 注(Note):**—P<0.01; *—P<0.05. 表 5 东北黑土施氮和不施氮处理nirS型反硝化细菌网络参数
Table 5 Parameters of nirS-type denitrifying bacterial network under N and non-N fertilizationin a black soil of northeast China
处理
Treatment阈值
Similarity threshold
(St)节点数
Network size
(n)连线数
Link number平均连通度
Average connectivity
(avgK)平均路径长
Average geodesic distance
(GD)平均聚集系数
Average clustering coefficient
(avgCC)模块性 (模块数)
Modularity
(No. of modules)R2 of
power-law施氮处理
N fertilization
(N,NK,NP,NPK)0.70 99 192 3.88 4.90 0.231 0.618 (7) 0.734 不施氮处理
Non-N fertilization
(NoF,P,K,PK)0.71 137 229 3.34 4.62 0.166 0.644 (17) 0.847 -
[1] Yin C, Fan F, Song A, et al. Denitrification potential under different fertilization regimes is closely coupled with changes in the denitrifying community in a black soil[J]. Applied Microbiology and Biotechnology, 2015, 99(13): 5719-5729. DOI: 10.1007/s00253-015-6461-0
[2] 宋亚娜, 吴明基, 林艳. 稻田土壤nirS型反硝化细菌群落对氮肥水平的响应[J]. 中国农业科学, 2013, 46(9): 1818-1826. DOI: 10.3864/j.issn.0578-1752.2013.09.009 Song Y, Wu M, Yan L. Response of the denitrifying bacterial nirS gene community to nitrogen fertilizer in paddy field[J]. Scientia Agricultura Sinica, 2013, 46(9): 1818-1826. DOI: 10.3864/j.issn.0578-1752.2013.09.009
[3] Zumft W G. Cell biology and molecular basis of denitrification[J]. Microbiology and Molecular Biology Reviews. 1997, 61(4): 533-616
[4] 曾希柏, 王亚男, 王玉忠, 等. 施肥对设施菜地nirK型反硝化细菌群落结构和丰度的影响[J]. 应用生态学报, 2014, 25(2): 505-514. Zeng X, Wang Y, Wang Y, et al. Effects of different fertilization regimes on abundance and community structure of the nirK-type denitrifying bacteria in greenhouse vegetable soils[J]. Chinese Journal of Applied Ecology, 2014, 25(2): 505-514.
[5] Coyne M S, Arunakumari A, Averill B A, et al. Immunological identification and distribution of dissimilatory heme cd1 and nonheme copper nitrite reductase in denitrifying bacteria[J]. Applied and Environmental Microbiology, 1989, 55(11): 2924-2931.
[6] Jones C M, Stres B, Rosenquist M, et al. Phylogenetic analysis of nitrite, nitric oxide, and nitrous oxide respiratory enzymes reveal a complex evolutionary history for denitrification[J]. Molecular Biology and Evolution, 2008, 25(9): 1955-1966. DOI: 10.1093/molbev/msn146
[7] Sun R, Guo X, Wang D, et al. Effects of long-term application of chemical and organic fertilizers on the abundance of microbial communities involved in the nitrogen cycle[J]. Applied Soil Ecology, 2015, 95: 171-178. DOI: 10.1016/j.apsoil.2015.06.010
[8] Hou S, Ai C, Zhou W, et al. Structure and assembly cues for rhizospheric nirK- and nirS-type denitrifier communities in long-term fertilized soils[J]. Soil Biology and Biochemistry, 2018, 119: 32-40. DOI: 10.1016/j.soilbio.2018.01.007
[9] Maeda K, Morioka R, Hanajima D, et al. The impact of using mature compost on nitrous oxide emission and the denitrifier community in the cattle manure composting process[J]. Microbial Ecology, 2010, 59(1): 25-36. DOI: 10.1007/s00248-009-9547-3
[10] Tang Y, Zhang X, Li D, et al. Impacts of nitrogen and phosphorus additions on the abundance and community structure of ammonia oxidizers and denitrifying bacteria in Chinese fir plantations[J]. Soil Biology and Biochemistry, 2016, 103: 284-293. DOI: 10.1016/j.soilbio.2016.09.001
[11] Chen Z, Luo X, Hu R, et al. Impact of long-term fertilization on the composition of denitrifier communities based on nitrite reductase analyses in a paddy soil[J]. Microbial Ecology, 2010, 60(4): 850-861. DOI: 10.1007/s00248-010-9700-z
[12] Yang Y, Zhao J, Jiang Y, et al. Response of bacteria harboring nirS and nirK genes to different N fertilization rates in an alkaline northern Chinese soil[J]. European Journal of Soil Biology, 2017, 82: 1-9. DOI: 10.1016/j.ejsobi.2017.05.006
[13] 丁建莉, 姜昕, 马鸣超, 等. 长期有机无机肥配施对东北黑土真菌群落结构的影响[J]. 植物营养与肥料学报, 2017, 23(4): 914-923. DOI: 10.11674/zwyf.16357 Ding J, Jiang X, Ma M, et al. Structure of soil fungal communities under long-term inorganic and organic fertilization in black soil of Northeast China[J]. Journal of Plant Nutrition and Fertilizers, 2017, 23(4): 914-923. DOI: 10.11674/zwyf.16357
[14] Zhou J, Guan D, Zhou B, et al. 2015. Influence of 34-years of fertilization on bacterial communities in an intensively cultivated black soil in northeast China[J]. Soil Biology and Biochemistry, 2015, 90: 42-51 DOI: 10.1016/j.soilbio.2015.07.005
[15] 尹昌, 范分良, 李兆君, 等. 长期施用有机和无机肥对黑土nirS型反硝化菌种群结构和丰度的影响[J]. 环境科学, 2012, 33(11): 3967-3975. Yin C, Fan F, Li Z, et al. Influences of long-term application of organic and inorganic fertilizers on the composition and abundance of nirS-type denitrifiers in black soil[J]. Environmental Science, 2012, 33(11): 3967-3975.
[16] 郑远, 李玉英, 丁传雨, 等. 能源植物修复镉污染土壤对根际细菌网络结构的影响[J]. 环境科学学报, 2016, 36(7): 2605-2612. Zheng Y, Li Y, Ding C, et al. Effects of bioenergy cropping on rhizosphere bacteria networks structure in Cd-contaminated soil[J]. Acta Scientiae Circumstantiae, 2016, 36(7): 2605-2612.
[17] Lu L, Yin S, Liu X, et al. Fungal networks in yield-invigorating and -debilitating soils induced by prolonged potato monoculture[J]. Soil Biology and Biochemistry, 2013, 65: 186-194. DOI: 10.1016/j.soilbio.2013.05.025
[18] Throbäck I N, Enwall K, Jarvis A, et al. Reassessing PCR primers targeting nirS, nirK and nosZ genes for community surveys of denitrifying bacteria with DGGE[J]. FEMS Microbiology Ecology, 2004, 49(3): 401-417. DOI: 10.1016/j.femsec.2004.04.011
[19] Hu X, Liu J, Zhu P, et al. Long-term manure addition reduces diversity and changes community structure of diazotrophs in a neutral black soil of northeast China[J]. Journal of Soils and Sediments, 2018, 18(5): 2053-2062. DOI: 10.1007/s11368-018-1975-6
[20] Fish J A, Chai B, Wang Q, et al. FunGene: the functional gene pipeline and repository[J]. Frontiers in Microbiology, 2013, 4: 291.
[21] Edgar RC. UPARSE: highly accurate OTU sequences from microbial amplicon reads[J]. Nature Methods, 2013, 10: 996-998. DOI: 10.1038/nmeth.2604
[22] Zhou J, Deng Y, Luo F, et al. Phylogenetic molecular ecological network of soil microbial communities in response to elevated CO2[J]. mBio, 2011, 2(4): e00122-11.
[23] Zhou J, Deng Y, Luo F, et al. Functional molecular ecological networks[J]. mBio, 2010, 1(14): 1592-1601.
[24] Deng Y, Jiang Y, Yang Y, et al. Molecular ecological network analyses[J]. BMC Bioinformatics, 2012, 13(1): 113. DOI: 10.1186/1471-2105-13-113
[25] 胡晓婧, 刘俊杰, 魏丹, 等. 东北黑土区不同维度农田土壤真菌分子生态网络比较[J]. 应用生态学报, 2018, 29(11): 3802-3810. Hu X, Liu J, Wei D, et al. Comparison on fungal molecular ecological networks of agricultural soils with different latitudes in the black soil region of Northeast China[J]. Chinese Journal of Applied Ecology, 2018, 29(11): 3802-3810.
[26] Clauset A, Newman M E J, Moore C. Finding community structure in very large networks[J]. Physical Review, 2004, 70: 1-6.
[27] Yu Z, Hu X, Wei D, et al
[28] Zhou S, Huang T, Zhang C, et al. Illumina MiSeq sequencing reveals the community composition of NirS-Type and NirK-Type denitrifiers in Zhoucun reservoir-a large shallow eutrophic reservoir in northern China[J]. RSC Advances, 2016, 6(94): 91517-91528. DOI: 10.1039/C6RA18017E
[29] Zeng W, Zhang J, Wang A, et al. Denitrifying phosphorus removal from municipal wastewater and dynamics of “Candidatus Accumulibacter” and denitrifying bacteria based on genes of ppk1, narG, nirS and nirK[J]. Bioresource Technology, 2016, 207: 322-331. DOI: 10.1016/j.biortech.2016.02.016
[30] Pereira E S M C, Schloter-Hai B, Schloter M, et al. Temporal dynamics of abundance and composition of nitrogen-fixing communities across agricultural soils[J]. PLoS One, 2013, 8(9): 1-15.
[31] Sánchez C, Minamisawa K. Redundant roles of Bradyrhizobium oligotrophicum Cu-type (NirK) and cd1-type (NirS) nitrite reductase genes under denitrifying conditions[J]. FEMS Microbiology Letters, 2018, 365(5): 1-7.
[32] van Berkum P. Expression of uptake hydrogenase and hydrogen oxidation during heterotrophic growth of Bradyrhizobium japonicum[J]. Journal of Bacteriology, 1987, 169(10): 4565-4569. DOI: 10.1128/jb.169.10.4565-4569.1987
[33] Wang Y, Li C, Kou Y, et al. Soil pH is a major driver of soil diazotrophic community assembly in Qinghai-Tibet alpine meadows[J]. Soil Biology and Biochemistry, 2017, 115: 547-555. DOI: 10.1016/j.soilbio.2017.09.024
[34] Tao R, Wakelin SA, Liang Y, et al. Nitrous oxide emission and denitrifier communities in drip-irrigated calcareous soil as affected by chemical and organic fertilizers[J]. Science of the Total Environment, 2018, 612: 739-749. DOI: 10.1016/j.scitotenv.2017.08.258
[35] Rousk J, Bååth E, Brookes PC, et al. Soil bacterial and fungal communities across a pH gradient in an arable soil[J]. The ISME Journal, 2010, 4(10): 1340-1351. DOI: 10.1038/ismej.2010.58
[36] Lauber C L, Hamady M, Knight R, et al. Pyrosequencing-based assessment of soil pH as a predictor of soil bacterial community structure at the continental scale[J]. Applied Environmental Microbiology, 2009, 75(15): 5111-5120. DOI: 10.1128/AEM.00335-09
[37] Tang Y, Zhang M, Chen A, et al. Impact of fertilization regimes on diazotroph community compositions and N2-fixation activity in paddy soil[J]. Agriculture, Ecosystems and Environment, 2017, 247: 1-8. DOI: 10.1016/j.agee.2017.06.009
[38] Freilich S, Kreimer A, Meilijson I, et al. The large-scale organization of the bacterial network of ecological co-occurrence interactions[J]. Nucleic Acids Research, 2010, 38(12): 3857-3868. DOI: 10.1093/nar/gkq118
[39] Montoya J M, Pimm S L, Solé R V. Ecological networks and their fragility[J]. Nature, 2006, 442(7100): 259-264. DOI: 10.1038/nature04927
[40] Xiao X, Liang Y, Zhou S, et al. Fungal community reveals less dispersal limitation and potentially more connected network than that of bacteria in bamboo forest soils[J]. Molecular Ecology, 2018, 27(2): 550-563. DOI: 10.1111/mec.14428
[41] Kitano H. Biological robustness[J]. Nature Reviews Genetics, 2004, 5: 826-837.
[42] Wang Y, Zhang R, Zheng Q, et al. Bacterioplankton community resilience to ocean acidification: evidence from microbial network analysis[J]. Ices Journal of Marine Science, 2016, 73: 865-875. DOI: 10.1093/icesjms/fsv187
[43] Yao M, Rui J, Li J, et al. Rate-specific responses of prokaryotic diversity and structure to nitrogen deposition in the Leymus chinensis steppe[J]. Soil Biology and Biochemistry, 2014, 79: 81-90. DOI: 10.1016/j.soilbio.2014.09.009
-
期刊类型引用(9)
1. 苏亚飞,包文彬,李彦生,于镇华,刘晓冰. 农田土壤酸化:原理、危害及缓解措施. 土壤与作物. 2025(01): 1-14 .  百度学术
百度学术
2. 孙鹏洲,罗珠珠,李玲玲,牛伊宁,王晓菲,田建霞,刘家鹤. 黄土高原紫花苜蓿种植对土壤反硝化细菌群落的影响. 中国生态农业学报(中英文). 2023(01): 67-78 .  百度学术
百度学术
3. 晁韶良,郇环,周向同,刘媚. 包气带反硝化菌多样性和群落结构垂向分布规律与主控环境因子研究:以北京市潮白河冲洪积扇为例. 环境科学研究. 2023(07): 1354-1367 .  百度学术
百度学术
4. 刘耕苑,肖杰,高明霞,孙本华,张树兰,杨学云,冯浩,张彤勋. 长期施肥对土nirS型反硝化细菌的影响及其与N_2O排放的关系. 水土保持研究. 2022(02): 99-105 .  百度学术
百度学术
5. 李德文,季倩如,刘英,周文玲,吴嘉仪,赵雨森. 农田防护林根际与非根际土壤氮素形态特征. 森林工程. 2022(03): 8-15+25 .  百度学术
百度学术
6. 喻江,李彦生,谢志煌,高志颖,刘晓冰,于镇华. 不同施肥方式对东北旱田黑土nirK和nirS型反硝化细菌群落结构的影响及黑土保护建议. 自然资源学报. 2022(09): 2306-2318 .  百度学术
百度学术
7. 雷善钰,辜运富,张亚洁,叶田会,杨鹏,余伟,闫芳芳,张宗锦. 攀枝花干热河谷区不同海拔高度农田土壤nirS型反硝化细菌的群落结构分析. 土壤通报. 2022(06): 1368-1375 .  百度学术
百度学术
8. 李玉倩,马俊伟,高超,霍守亮,夏星辉. 青藏高原高寒湿地春夏两季根际与非根际土壤反硝化速率及nirS型反硝化细菌群落特征分析. 环境科学. 2021(10): 4959-4967 .  百度学术
百度学术
9. 宋延静,马兰,李俊林,单燕,王洁,赵玲玲,王向誉. 山东省盐渍化地区芦苇根际反硝化细菌群落结构研究. 山东农业科学. 2021(12): 137-142 .  百度学术
百度学术
其他类型引用(14)



 下载:
下载: