Genome-wide association analysis for yield and nitrogen efficiency related traits of wheat at maturity stage
-
摘要:目的
探明控制产量及氮效率相关性状的稳定基因关联位点,为高产和氮素高效小麦育种及养分管理提供参考。
方法采用134个小麦品种 (系) 为试验材料,依据小麦产量水平600和400 kg/hm2的需氮量设置正常氮 (T1) 和低氮 (T2) 2个处理,进行了2年田间试验,共形成4个处理环境。对小麦成熟期与产量及氮效率相关的14个性状进行了表型鉴定,采用GLM + Q一般线性模型和MLM + K + Q混合线性模型相结合的方法,利用群体差异SNP分子标记 (90K SNP芯片) 对小麦产量和氮效率相关性状进行全基因组关联分析。
结果与正常氮处理相比,低氮处理条件下小麦籽粒产量、秸秆产量显著下降;所有性状的遗传力均在75%以上,其中小穗数的遗传力最高 (95.12%)。利用9329个SNP标记进行关联分析,共检测到382个SNP标记位点与供试14个性状存在显著关联(P ≤ 0.001),分布在21条染色体上。有305个 (79.84%) SNP标记位点仅在一个关联分析环境中被检测到;有77个位点在至少两种处理环境 (包含平均值环境) 中被检测到与同一性状显著关联 (稳定关联标记)。其中9个SNP标记位点在至少3个环境中被检测到。在4个环境下 (包括平均值环境) 均检测到的稳定关联位点有4个:BobWhite_c47168 _598、Kukri_c31599_1456、wsnp_CAP11_c1761_958064和Excalibur_c62826_254,分别与穗粒数、籽粒氮含量和小穗数显著关联;同时与至少3个性状显著关联的SNP标记位点共12个,分别位于2A、3A、4A、5B和7B染色体上,贡献率为11.14%~22.97%,其中,标记BobWhite_c47168_598和Kukri_c31599_1456还分别定位了两个多环境 (4种环境) 稳定位点。根据12个与多性状共同定位位点和4个多环境 (4个环境) 稳定位点关联的SNP标记位置获得稳定位点附近区域的基因 (基于LD block等方式估算的区间大小),使用NCBI中CDD工具和EnsemblPlants网站对这些基因进行基因功能注释,根据功能注释,共得到10个与产量和氮效率性状相关的候选基因。
结论氮供应水平对小麦成熟期产量和氮效率相关性状均有显著影响,氮素累积量与小麦产量显著正相关。本试验中检测到的与产量及氮效率相关性状显著关联的位点中,79.84%的SNP标记位点仅在一个氮处理环境中出现,环境稳定性较差;4个位点在4个环境条件下均被检测到,环境稳定性较好;12个SNP标记位点同时与至少3个性状显著关联,涉及性状均为产量及氮效率相关性状,反映了籽粒产量与氮素效率之间的显著相关关系,也可能是同时控制这些性状的遗传热点位点。根据这些热点位点和环境稳定性好的位点筛选到10个与产量及氮效率相关性状有关候选基因,值得深入探讨。
Abstract:ObjectivesTo identify and locate stable molecular markers which are significantly correlated to the traits of yield and N efficiency of wheat under different N nutrition levels.
MethodsA group of 134 wheat cultivars or lines were subjected to normal level N (required for 600 kg/hm2 yield) and low level N (required for 400 kg/hm2 yield) and four environmental treatments over two years. Fourteen traits related to yield and N efficiency of wheat were measured. Genome-wide association analysis using 90K SNP molecular markers was carried out for the tested traits by GLM + Q general linear model and MLM + K + Q mixed linear model.
ResultsThe grain and straw yield under low N treatment were significantly lower than those under normal N treatment. The heritability of all the tested traits were above 75%. According to genome-wide association analysis on the 9329 SNPs, a total of 382 molecular marker sites located on 21 chromosomes were identified associating with 14 traits (P ≤ 0.001). Of all the identified marker sites, 305 (79.84%) were detected in only one environment; 77 sites were detected to be significantly associated with the same trait in at least two treatment environments; 9 SNP marker sites were detected in at least 3 environments and four environmental stable SNP marker sites were identified in at least four environments. The four stable markers (BobWhite_c47168 _598, Kukri_c31599_1456, wsnp_CAP11_c1761_958064 and Excalibur_c62826_254) were significantly related to number of grains per ear, grain N content, and number of spikelets. Twelve multi-trait co-location SNP marker sites which were simultaneously associated with at least three traits were located on chromosomes 2A, 3A, 4A, 5B, and 7B. Their average contribution rate ranged from 11.14% to 22.97%. The marker BobWhite_c47168_598 and Kukri_c31599_1456 were also located at two environmental stable sites (four environments). Candidate gene prediction was conducted in the region near stable sites (interval size estimated based on LD block and other methods) according to the SNP markers position of the 12 multi-trait co-location sites and four environmental stable sites. Gene function annotations of these genes were performed using the CDD tool of NCBI and Ensembl Plants website. According to the function annotations, a total of 10 candidate genes related to yield and nitrogen efficiency traits were determined.
ConclusionsN accumulation was significantly and positively correlated with wheat yield. About 79.84% SNP marker sites which were significantly correlated with yield and N efficiency traits were detected only in specific environments. In addition, four environmental stable SNP markers were detected in four environments. Twelve molecular marker sites were significantly associated with three yield and nitrogen efficiency related traits simultaneously. This reflects the genetic hotspot which simultaneously control the yield and nitrogen efficiency traits. In summary, according to the environmental stable SNP marker sites and hotspots for different traits, we screened ten candidate genes related to yield and nitrogen efficiency. However, it will be worthy of further exploration.
-
Keywords:
- wheat /
- genome-wide association analysis /
- yield /
- nitrogen efficiency
-
小麦 (Triticum aestivum L.) 是我国的第三大粮食作物和战略储备物资,保证小麦高产稳产具有非常重要的战略意义。氮元素是植物生长发育必需的大量营养元素,也是生产中施用量最大的肥料元素,在植物生长和发育等生命活动中发挥着不可替代的作用。“绿色革命”培育出的一大批耐肥水小麦品种,能够在充足的肥水供应条件下获得产量的大幅度提高,为解决我国人民的温饱问题做出了巨大贡献。但是,为了获得高产,近年来小麦生产过程中氮肥施用量严重超出实际需求量[1]。大量的氮肥投入导致肥料利用率大幅度降低[2],只有21.2%~35.9%,过量施氮还导致了一系列环境污染问题[3]。提高氮素利用效率、降低施氮量是缓解氮肥环境压力的重要途径之一[4]。
作物的氮素利用效率是指单位土壤有效氮可生产的籽粒产量,由作物的氮素吸收效率 (地上部氮积累量/土壤供氮量) 和氮素利用效率 (籽粒产量/地上部氮积累量) 共同决定[5-7]。小麦不同基因型对氮素的吸收、利用存在显著的差异[5, 8-11]。因此,对小麦进行遗传改良,充分挖掘其氮营养高效基因潜力,是提高氮素利用效率,促进农业可持续发展的有效途径[4]。利用产量、生物量、氮吸收累积等相关指标可以在一定程度上反映小麦的氮效率情况。通过关联分析可以找到与这些氮效率相关指标显著关联的分子标记,为进一步挖掘相关候选基因,培育氮高效小麦品种提供参考。
关联分析是理解表型变异与遗传多态性之间关系的有效方法。目前,GWAS分析已成功应用于鉴定小麦的产量及产量构成因素[12-13]、非生物胁迫抗性[14-15]、抗病性[16-17]和谷物品质[18-19]等性状的QTL。小麦中有很多重要的经济和农艺性状都是数量性状,如品质、产量相关性状,抗性以及养分效率相关性状等。由于数量性状表现为连续变异、易受环境影响、遗传基础十分复杂、受多基因的数量性状位点 (quantitative trait locus,QTL) 控制、表现型与基因型之间并没有明确的对应关系等,因此氮效率相关性状的遗传基础研究一直进展缓慢。QTL分析和GWAS分析仍然是研究此类性状的重要途经。本研究选用134个小麦品种/系,在不同氮处理条件下,进行产量氮效率相关性状的测定,结合高通量的SNP技术获得的分子标记遗传图谱,对连续两年的产量和氮效率相关性状进行全基因组关联分析,为小麦氮效率相关性状的分子标记辅助选择育种和重要QTL的克隆奠定基础。
1. 材料与方法
1.1 试验材料
采用近年来育成的134个小麦品种(系)为试验材料组成自然品种(系)群体,其中包括中国山东66个、山西8个、河南16个、河北24个、陕西14个、江苏2个、四川2个及澳大利亚和加拿大各1个 (表1)。
表 1 供试小麦品种Table 1. Wheat cultivars used in this study序号
No.品种(系)名称
Variety/line name序号
No.品种(系)名称
Variety/line name序号
No.品种(系)名称
Variety/line name序号
No.品种(系)名称
Variety/line name序号
No.品种(系)名称
Variety/line name1 昌乐5号 28 山农45 55 淄12 82 新麦18 109 石新616 2 德选1号 29 山农483 56 954 (7)-8 83 新麦26 110 石新618 3 济麦20 30 山农664 57 BSS 84 豫麦49 111 石新828 4 济麦21 31 山农8355 58 LS3283 85 豫麦54 112 石优17号 5 济麦22 32 山农紫麦1号 59 LS4211 86 郑麦9023 113 唐麦8号 6 济南17 33 山融3号 60 LS4697 87 中优9507 114 小偃81 7 济宁16 34 泰农18 61 LS4942 88 周99233 115 AN2 8 济宁17 35 泰山21 62 LS6045 89 周黑麦1号 116 AN3 9 科信9号 36 泰山22 63 LS6109 90 周麦24 117 蓝58 10 莱州137 37 泰山23 64 M8008-2 91 藁优9618 118 陕627 11 莱州95021 38 泰山24 65 贵农35选21 92 邯00-7086 119 陕农138 12 良星99 39 郯麦98 66 薛紫麦 93 河农827 120 陕农534 13 临麦4号 40 潍麦7号 67 黑小麦76 94 河农4198 121 西农85 14 临麦6号 41 潍麦8号 68 晋麦33号 95 河农6049 122 西农213 15 鲁麦14 42 汶农5号 69 临丰3号 96 衡0628 123 西农889 16 鲁麦21 43 汶农6号 70 临旱822 97 衡6599 124 西农9871 17 鲁麦22 44 鑫289 71 临抗16号 98 冀5265 125 小偃22 18 鲁麦23 45 烟5072 72 临优145 99 冀丰703 126 小偃92 19 齐丰1号 46 烟99102 73 品资旱99-2 100 金麦1号 127 小偃216 20 山农0431 47 烟99603 74 平阳298 101 科农199 128 运丰139 21 山农1186 48 烟农0428 75 洛旱6号 102 廊研43号 129 淮麦16 22 山农12 49 烟农15 76 洛旱8号 103 石54 130 淮麦18 23 山农15 50 烟农19 77 洛旱21号 104 石4185 131 川35050 24 山农17 51 烟农21 78 洛旱23号 105 石家庄8号 132 中国春 25 山农18 52 烟农22 79 洛旱24号 106 石麦15号 133 澳大利亚红麦 26 山农21 53 烟农23 80 漯珍1号 107 石麦16号 134 加拿大超强筋麦 27 山农23 54 烟农836 81 新麦16 108 石麦18号 注(Note):表中品种 (系) 来源 Source of cultivar (line) listed in the table:1~66,中国山东 Shandong,China;67~74,中国山西Shanxi,China;75~90,中国河南 Henan,China;91~114,中国河北 Hebei,China;115~128,中国陕西 Shaanxi,China;129~130,中国江苏 Jiangsu,China;131~132,中国四川 Sichuan,China;133,澳大利亚 Australia;134,加拿大 Canada. 1.2 试验设计
1.2.1 试验处理及实施
试验设在山东省泰安市山东农业大学农学实验站,土壤类型为褐土,pH为7.4~7.8。设置正常氮对照 (normal N control,T1) 和低氮 (low N input,T2) 2个处理。参考目标产量法,正常对照、低氮处理小麦产量分别为600 、400 kg/hm2需要的施氮量,2012—2013年度分别为N 195和120 kg/hm2,2013—2014年度分别为N 182和120 kg/hm2。每个处理重复两次,试验重复两年 (E1和E2)。供试小麦群体每个株系每年、每次重复种植3行,行长50 cm,每行点播25粒种子,越冬前按株距约10 cm每行定苗5株,在收获时,各小麦品种(系)选择生长均匀的一行进行田间性状调查并收获。池栽小区面积均为110 m2,各小区之间用水泥墙 (深1.5 m) 隔开,经多年匀地处理,地力条件基本一致。磷、钾肥和40%的氮肥以基肥形式一次施入,剩余的60%氮肥返青拔节期施入。
1.2.2 性状调查及方法
收获前10天左右,各品种(系)小麦选择长势均匀的一行 (原则上为中间一行) 进行田间性状调查。调查性状包括:整行总穗数,换算成单株穗数;每个单株选择主穗 (共5穗) 调查基部不育小穗数、顶部不育小穗数、总小穗数和穗粒数。
小麦收获后,各株系地上部分全部收获至网袋中,晾干后称取地上部总重,然后脱粒,籽粒和秸秆分别收集,称取籽粒重量并换算成单株籽粒重 (g/plant)。根据地上部总重与籽粒重的差值换算单株秸秆重 (g/plant);收获指数 (单株籽粒重/地上部总重)。每株小麦取籽粒100粒,调查并计算千粒重,3个重复。籽粒氮含量和秸秆氮含量采用H2SO4–H2O2消煮—凯氏法测定;籽粒氮累积量 = 单株籽粒重 × 籽粒氮含量/1000;秸秆氮累积量 = 单株秸秆重 × 秸秆氮含量/1000;籽粒氮利用效率 = 单株籽粒重/籽粒氮含量;秸秆氮利用效率 = 单株秸秆重/秸秆氮含量。
1.3 数据统计方法
1.3.1 表型数据分析
采用SPSS 19.0软件和R语言对试验相关数据进行方差及相关统计分析。性状广义遗传力计算公式:hB2 = σg2/ (σg2 + σe2)[20],其中σg2代表遗传方差,σe2代表环境方差。
1.3.2 群体结构分析
用Powermarker 3.25软件分析等位变异数,并计算多态性信息含量 (polymorphism information content,PIC)。用Structure 2.3.1软件对供试材料进行群体结构分析,估测群体结构,并计算材料相应的Q矩阵[21]。估计最佳群体数K,其取值范围为1~15,将MCMC (Markov Chain Monte Carlo) 开始时的不作数迭代 (length of burn-in period) 设为50000次,再将不作数迭代后的MCMC设为100000次,每个K值重复运行5次,依据似然值最大的原则选取合适的K值作为群体数目[22]。
1.3.3 关联分析
本研究用包含81587个SNP的小麦90K基因芯片对研究群体进行扫描,在得到的多态性SNP中,去除最小等位基因频率 (MAF) 小于5%,缺失率大于15%的SNP位点,剩余9329个SNP。参考Wang等[23]公布的SNP标记染色体位置信息,大约85.24% (7952/9329) SNP有遗传位置信息,分布在21条染色体上。在小麦21条染色体中,染色体间标记数量差异较大,每条染色体分布的标记数量为27~794个,4D染色体上的标记数最少,5B染色体上的标记数最多;SNP标记在A、B和D基因组间分布不均匀,B基因组的标记数最多,有4022个,占43.11%,D基因组的标记数最少,仅740个,占7.93%,A、B、D基因组上的标记密度分别是2.79、3.63和0.66个标记/cM。采用TASSEL 2.1软件中的GLM (general linear model) 模型和MLM (mixed linear model) 模型相结合分子标记数据、群体结构数据和氮效率相关性状数据进行标记-性状的关联分析,当性状与标记关联的P ≤ 0.001时认为存在显著关联。
1.3.4 小麦产量和氮效率相关性状候选基因筛选
根据12个与多性状共同定位位点和4个多环境 (4个环境) 稳定位点关联的SNP标记位置获得稳定位点附近区域的基因 (基于LD block等方式估算的区间大小),使用NCBI (https://www.ncbi.nlm.nih.gov) 中CDD工具和Ensembl Plants (http://plants.ensembl.org/index.html) 网站对这些基因进行基因功能注释,根据功能注释,筛选出可能与氮效率相关性状相关的候选基因。
2. 结果与分析
2.1 成熟期产量及氮效率相关性状的统计分析及其遗传力分析
成熟期产量和氮效率相关性状的统计分析结果 (表2)表明,与正常氮处理 (T1) 相比,在低氮处理 (T2) 下,单株籽粒重量、秸秆重量、秸秆氮累积量显著下降。14个性状中遗传力最大为总小穗数 (95.12%),最小为秸秆氮含量 (75.16%)。
表 2 小麦成熟期产量和氮效率相关性状的统计参数和遗传力Table 2. Statistical parameters and heritability of traits related to yield and nitrogen efficiency of wheat at maturity性状
Traits处理
Treatment均值
Average变异系数
CV (%)遗传力 (%)
Heritability性状
Traits处理
Treatment均值
Average变异系数
CV (%)遗传力 (%)
Heritability穗数 SN T1E1 12.80 a 20.9 88.35 单株秸秆 StDW (g/plant) T1E1 28.69 a 18.3 92.97 T2E1 12.57 a 20.4 T2E1 22.12 b 26.3 T1E2 10.43 b 22.7 T1E2 27.80 a 20.9 T2E2 9.59 b 26.9 T2E2 21.09 b 22.1 小穗数 TSS T1E1 19.40 a 5.9 95.12 籽粒氮含量 GNC (g/kg) T1E1 22.95 b 9.3 91.96 T2E1 19.33 a 6.5 T2E1 21.59 b 10.2 T1E2 19.18 a 6.3 T1E2 25.33 a 9.4 T2E2 19.41 a 6.3 T2E2 23.35 ab 11.8 基部不育小穗数 BSSS T1E1 0.44 a 83.6 87.44 秸秆氮含量 StNC (g/kg) T1E1 5.48 b 22.2 75.16 T2E1 0.50 a 66.7 T2E1 3.99 c 18.0 T1E2 0.62 a 64.8 T1E2 7.30 a 14.9 T2E2 0.57 a 73.5 T2E2 4.95 b 18.7 顶部不育小穗数 TSSS T1E1 0.35 a 124.6 80.88 籽粒氮累积量 GNA (g/plant) T1E1 0.54 ab 18.7 83.96 T2E1 0.38 a 123.8 T2E1 0.42 c 22.9 T1E2 0.04 b 320.6 T1E2 0.60 a 23.4 T2E2 0.05 b 273.6 T2E2 0.49 bc 30.8 穗粒数 GN T1E1 58.22 a 10.3 93.03 秸秆氮累积量 StNA (g/plant) T1E1 0.16 b 26.7 82.59 T2E1 56.14 a 10.5 T2E1 0.09 c 24.4 T1E2 57.89 a 12.1 T1E2 0.20 a 19.4 T2E2 57.59 a 12.1 T2E2 0.10 bc 30.2 千粒重 TGW (g) T1E1 45.05 b 9.5 85.30 籽粒氮利用率 GNUE (g/kg) T1E1 1.03 a 21.8 87.67 T2E1 44.58 b 8.7 T2E1 0.90 a 26.1 T1E2 50.53 a 8.9 T1E2 0.93 a 24.7 T2E2 49.54 a 9.0 T2E2 0.90 a 29 籽粒重 GWP (g/plant) T1E1 23.59 a 18.3 88.17 秸秆氮利用率 StNUE (g/kg) T1E1 5.24 a 24.8 85.98 T2E1 19.45 b 24.3 T2E1 5.54 a 24.7 T1E2 23.53 a 22.5 T1E2 3.81 b 32.0 T2E2 21.08 ab 28.5 T2E2 4.26 b 27.5 注(Note):SN—Sprike number;TSS—Total spikelet number per spike;BSSS—Bottom sterile spikelet number per spike;TSSS—Top sterile spikelet number per spike;GN—Grain number per spike;TGW—Thousand grains weight;GWP—Grain dry weight per plant;StDW—Straw dry weight per plant;GNC—Grain N content;StNC—Straw N content per plant;GNA—Grain N accumulation per plant;StNA—Straw N accumulation per plant;GNUE—Grain nitrogen use efficiency;StNUE—Straw nitrogen use efficiency. E1和E2代表两个试验年份 E1 and E2 represent the two experimental years;T1和T2分别代表正常氮和低氮处理 T1 and T2 represent normal and low nitrogen supply treatments;同列数据后不同小写字母代表两个处理之间差异显著 (P < 0.05) Values followed by different lowercase letters indicate significant difference between two treatments at 0.05 level. 2.2 成熟期产量及氮效率相关性状的相关分析
成熟期8个产量相关性状 (SN、TSS、BSSS、TSSS、GN、GWP、StDW、TGW) 之间和6个氮效率相关性状 (GNC、StNC、GNA、StNA、GNUE、StNUE) 之间均显著相关。4个产量相关性状 (SN、GWP、StDW、TGW) 和6个氮效率相关性状绝大多数均显著相关 (图1)。
![]() 图 1 小麦成熟期产量及氮效率相关性状间的相关系数[注(Note):SN—穗数 Sprike number;TSS—小穗数 Total spikelet number per spike;BSSS—基部不育小穗数 Bottom sterile spikelet number per spike;TSSS—顶部不育小穗数 Top sterile spikelet number per spike;GN—穗粒数 Grain number per spike;TGW— 千粒重 Thousand grains weight;GWP—单株籽粒重Grain weight per plant;StDW—单株秸秆干重 Straw dry weight per plant;GNC—籽粒氮含量 Grain N content ;StNC—秸秆氮含量 Straw N content;GNUE—籽粒氮利用效率 Grain nitrogen use efficiency;StNUE—秸秆氮利用效率 Straw nitrogen use efficiency.]Figure 1. Correlation coefficients (r) between traits related to yield and nitrogen efficiency of wheat at maturity
图 1 小麦成熟期产量及氮效率相关性状间的相关系数[注(Note):SN—穗数 Sprike number;TSS—小穗数 Total spikelet number per spike;BSSS—基部不育小穗数 Bottom sterile spikelet number per spike;TSSS—顶部不育小穗数 Top sterile spikelet number per spike;GN—穗粒数 Grain number per spike;TGW— 千粒重 Thousand grains weight;GWP—单株籽粒重Grain weight per plant;StDW—单株秸秆干重 Straw dry weight per plant;GNC—籽粒氮含量 Grain N content ;StNC—秸秆氮含量 Straw N content;GNUE—籽粒氮利用效率 Grain nitrogen use efficiency;StNUE—秸秆氮利用效率 Straw nitrogen use efficiency.]Figure 1. Correlation coefficients (r) between traits related to yield and nitrogen efficiency of wheat at maturity2.3 群体结构分析
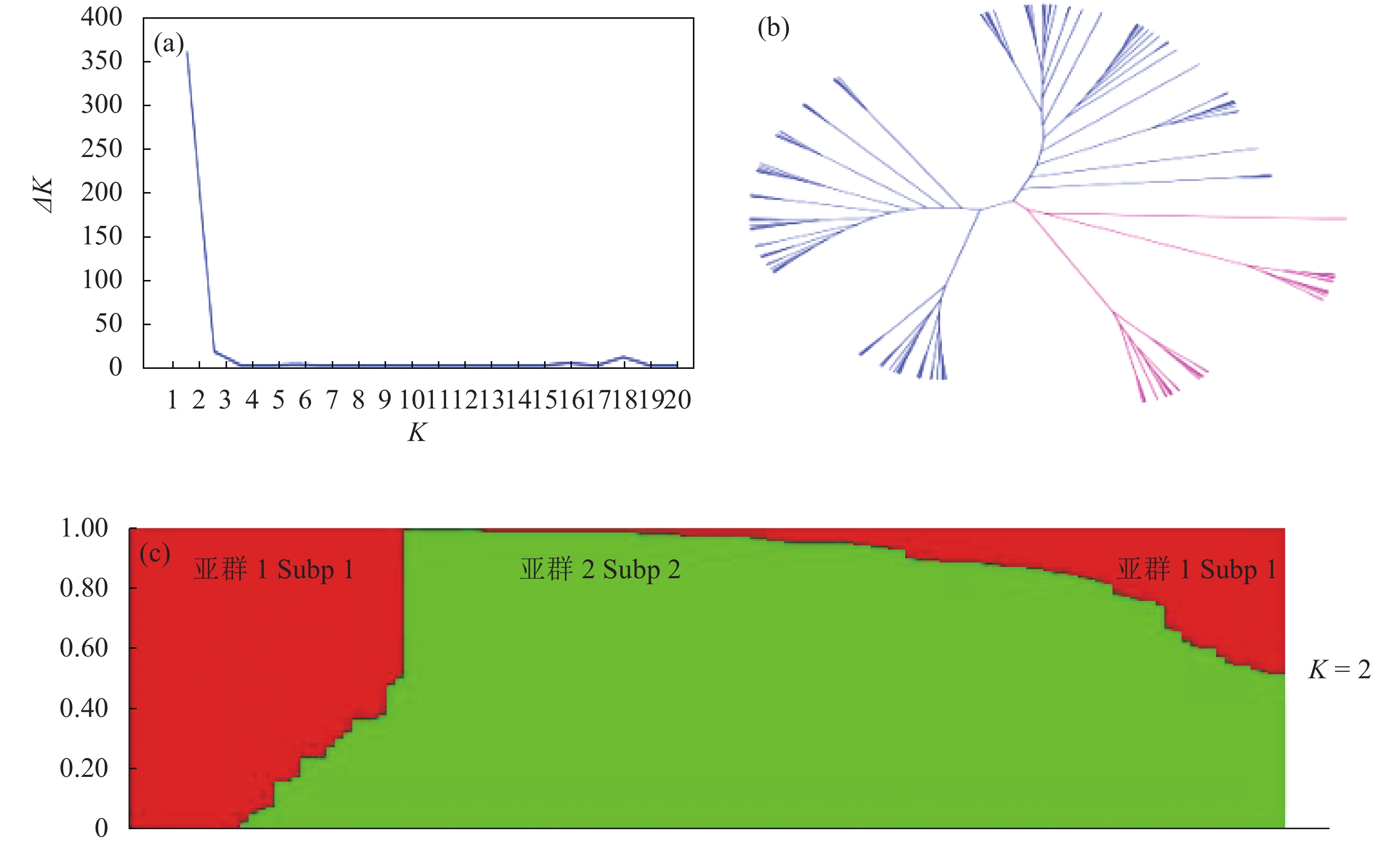
选择在小麦的21条染色体长臂和短臂上均匀分布的229个SNP标记,用Structure 22.3.4软件进行群体结构分析,结果显示,K = 2时,ΔK出现明显峰值 (图2a,c)。利用Power Marker V3.25计算了134个小麦品种两两间的遗传距离,并构建了NJ聚类图 (图2b),聚类结果将所有材料分为2个亚群。另外用Tassel软件进行主成分分析,前两个主成分可解释14.85%的群体信息。综合以上3种方法研究群体结构,确定为两个亚群。分别包括13个小麦品种/系 (亚群) 和121个小麦品种/系 (亚群2)。
![]() 图 2 供试小麦材料的群体结构[注(Note):图a为供试材料的K值与ΔK的关系;图b为基于遗传距离的NJ聚类图;图c为群体结构;2个亚群用不同颜色表示。Figure a shows graphical relationship between K and ΔK for wheat accessions;Figure b shows neighbor-joining tree of 134 wheat accessions. Figure c shows population structure of wheat accessions. The two subgroups identified from the tree are in different colors.]Figure 2. Population structure analysis of tested wheat materials based on unlinked SNP markers
图 2 供试小麦材料的群体结构[注(Note):图a为供试材料的K值与ΔK的关系;图b为基于遗传距离的NJ聚类图;图c为群体结构;2个亚群用不同颜色表示。Figure a shows graphical relationship between K and ΔK for wheat accessions;Figure b shows neighbor-joining tree of 134 wheat accessions. Figure c shows population structure of wheat accessions. The two subgroups identified from the tree are in different colors.]Figure 2. Population structure analysis of tested wheat materials based on unlinked SNP markers2.4 成熟期产量及氮效率相关性状的关联分析
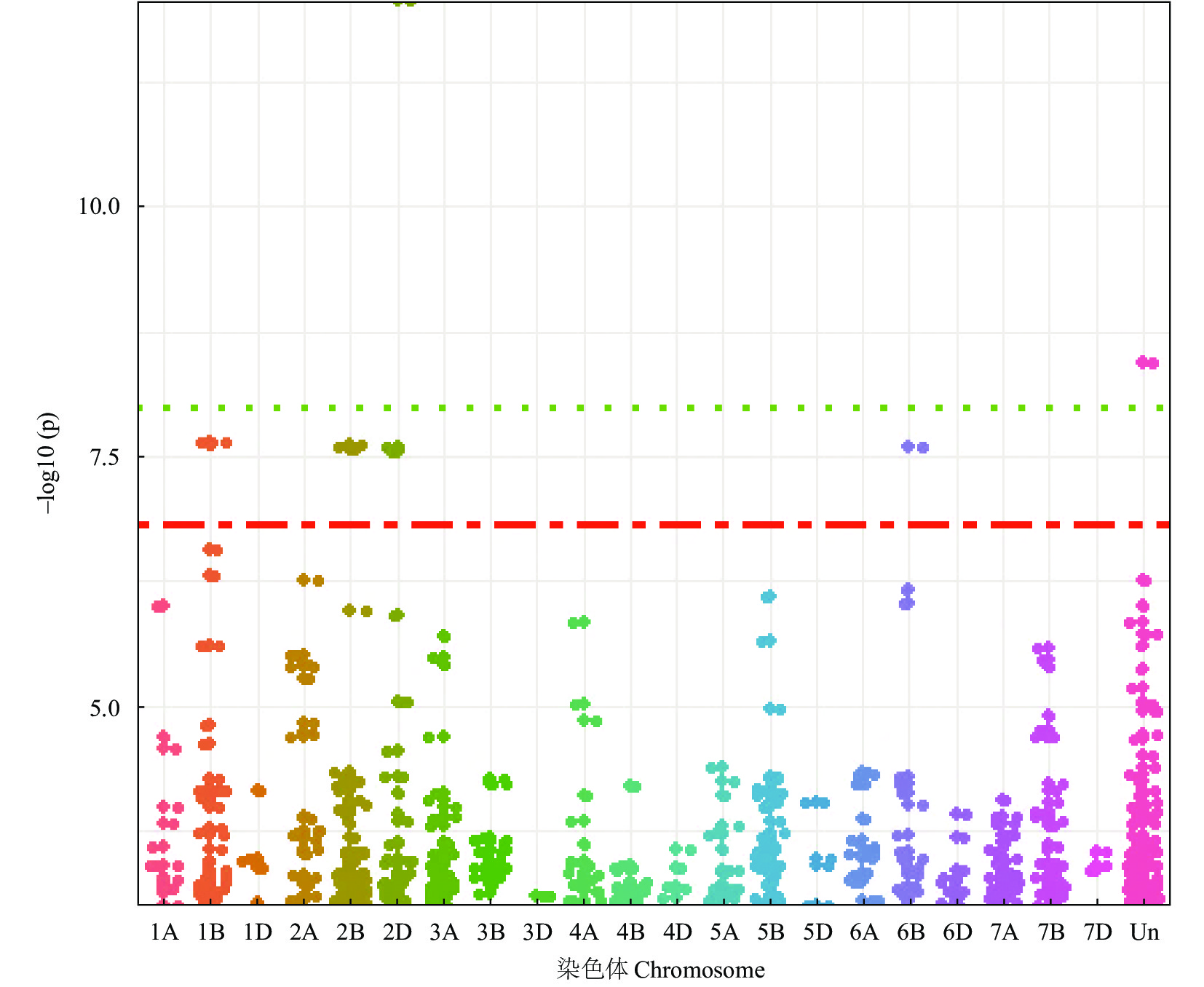
将134份试验材料的产量和氮效率相关性状与9329个SNP标记进行全基因组关联分析,在21条染色体上共检测到382个显著关联标记 (图3,具体数据见网址http://www.plantnutrifert.org)。关联分析结果中,R2为特定表型性状 (测定值) 在不同氮处理环境下产生的变异 (表型变异) 由该分子标记/基因贡献的比例 (贡献率)。本研究检测到的显著关联SNP标记位点对相应性状表型变异的贡献率 (R2) 范围是8.71%~53.06%。其中,与顶部不育小穗数 (TSSS) 显著关联的位点最多,共24个,表型变异贡献率 (R2) 为8.71%~53.06%。在定位到的全部382个显著关联的SNP标记位点中,有305个SNP标记位点仅在1个关联分析环境中检测到,有77个位点在至少两种处理环境 (包含平均值) 中被检测到与同一性状显著关联 (稳定关联标记)。其中9个显著关联的SNP标记位点在至少3个环境中被检测到 (表3),其中可能存在部分假阳性关联结果。在4个环境下 (包括平均值) 均检测到稳定关联位点有4个:BobWhite_c47168_598、Kukri_c31599_1456、wsnp_CAP11_c1761_958064和Excalibur_c62826_ 254,分别与穗粒数、籽粒全氮含量和小穗数显著关联。其中与籽粒全氮含量稳定关联的wsnp_CAP11_c1761 _958064贡献率达到15.09%。
表 3 小麦成熟期产量及氮效率相关性状稳定关联标记位点Table 3. The environmental stable markers which associated with maturity yield and N efficiency related traitsQTL 性状
Traits环境
Environment标记
Marker染色体
Chr位置
Pos (bp)P值 P-value 贡献率
R2 (%)最大值
Max最小值
MinQGn-2D-1 GN T1AV,T2AV,T2E2 BS00011740_51 2D 4004568 8.03 × 10–4 6.06 × 10–4 8.85~9.28 QTss-3B-1 TSS T1AV,T2AV,T2E2 BS00032692_51 3B 7069765 4.18 × 10–4 2.21 × 10–4 9.85~10.85 QTss-3B-5 TSS T1AV,T2AV,T2E2 Excalibur_c5977_1409 3B 7069765 4.18 × 10–4 2.21 × 10–4 9.85~10.85 QTss-3B-6 TSS T1AV,T2AV,T2E2 Excalibur_c5977_440 3B 7069765 4.18 × 10–4 2.21 × 10–4 9.85~10.85 QTss-3B-7 TSS T1AV,T1E1,T2AV,T2E2 Excalibur_c62826_254 3B 7069765 6.27 × 10–4 5.56 × 10–5 9.23~13.05 QGn-4A-1 GN T1E1,T1E2,T1AV,T2E2 BobWhite_c47168_598 4A 14437692 9.20 × 10–3 3.89 × 10–4 11.27~12.54 QGn-4A-2 GN T1E1,T2AV,T2E2 GENE-0689_791 4A 12712246 9.99 × 10–4 3.78 × 10–4 10.96~12.59 QGn-5B GN T1AV,T1E1,T1E2,T2AV Kukri_c31599_1456 5B 10124835 6.50 × 10–4 1.06 × 10–4 11.68~15.02 QGnc-7A-3 GNC T1AV,T1E1,T1E2,T2E1 wsnp_CAP11_c1761_958064 7A 13553770 8.58 × 10–4 8.03 × 10–5 11.30~15.09 注(Note):AV—平均值 Average;GN—穗粒数 Number of grains per spike;TSS—小穗数 Total spikelets;GNC—籽粒氮含量 Grain N concentration. E1和E2代表两个试验年份,T1和T2分别代表正常氮和低氮处理 E1 and E2 represent the two experimental years, and T1 and T2 represent normal and low nitrogen treatments,respectively. 2.5 成熟期产量及氮效率相关性状共同定位位点
试验结果还表明,许多SNP标记位点同时与多个性状显著关联。本试验中同时与至少3个性状显著关联的SNP标记位点共12个 (表4),分别位于2A、3A、4A、5B和7B染色体上,贡献率为11.14%~22.97%。其中,标记BobWhite_c47168_598和Kukri_c31599_1456还分别定位了两个多环境 (4个环境) 稳定位点。在12个多性状共同定位位点中,有10个位点均共同定位了单株籽粒重、籽粒氮累积量和秸秆氮累积量,有2个位点共同定位了穗粒数、秸秆氮含量和秸秆氮累积量。
表 4 小麦成熟期产量及氮效率相关性状簇集位点Table 4. The marker locations wheat yield and N efficiency related traits clustered at maturity stage簇
Cluster染色体
ChromosomeQTL数
No. of QTLQTL 处理
Treatment标记
Marker贡献率 (%)
R2C1 2A 3 QGwp-2A T2AV,T2E2 BobWhite_c10673_330 11.14~20.87 QGna-2A T2AV,T2E2 QStna-2A T2E2 C2 2A 3 QGwp-2A T2AV,T2E2 BobWhite_c26296_83 11.17~21.03 QGna-2A T2AV,T2E2 QStna-2A T2E2 C3 2A 3 QGwp-2A T2E2 Ex_c721_2127 12.23~20.85 QGna-2A T2AV,T2E2 QStna-2A T2E2 C4 2A 3 QGwp-2A T2E2 Kukri_c42616_467 13.61~18.36 QGna-2A T2AV,T2E2 QStna-2A T2E2 C5 2A 3 QGwp-2A T2E2 tplb0045j05_1405 13.61~18.36 QGna-2A T2AV,T2E2 QStna-2A T2E2 C6 3A 3 QGwp-3A T2E2 BS00011612_51 12.52~21.31 QGna-3A T2AV,T2E2 QStna-3A T2E2 C7 3A 4 QGwp-3A T2AV,T2E2 Kukri_c80104_809 12.23~22.18 QGna-3A T2AV,T2E2 2.6 候选基因预测
根据12个与多性状共同定位位点和4个多环境 (4个环境) 稳定位点关联的SNP标记位置获得稳定位点附近区域的基因 (基于LD block等方式估算的区间大小),使用EnsemblPlants网站和NCBI中CDD工具进行基因功能注释,从所有目标区段基因中筛选得到10个与产量和氮效率相关性状相关的基因 (表5)。在这些候选基因的注释中,TraesCS7A02G405600编码真核天冬氨酰蛋白酶家族蛋白 (eukaryotic aspartyl protease family protein),参与蛋白水解;TraesCS3B02G365500编码脱氢酶样蛋白 (dehydrogenase-like protein),与氧化还原过程有关;TraesCS2A02G022700编码ATP依赖性Clp蛋白酶衔接子蛋白clps (ATP-dependent Clp protease adapter protein clpS);TraesCS2A02G140400编码DNA修复蛋白类蛋白 (DNA repair protein-like protein);TraesCS2A02G142200编码F-box family protein,在泛素-蛋白酶体途径 (ubiquitin-protcasomcpathway,UPP) 中因特异识别底物蛋白而参与细胞周期调控、转录调控、细胞凋亡、细胞信号转导等生命活动;TraesCS3A02G319900和TraesCS4A02G461700分别编码Conserved oligomeric Golgi complex subunit 1和NBS-LRR resistance-like protein,Conserved oligomeric Golgi complex subunit 1可调节高尔基体内的运输和真核细胞中高尔基体的完整性;TraesCS7B02G343600编码精氨酸-谷氨酸二肽重复蛋白 (arginine-glutamic acid dipeptide repeats protein);TraesCS7B02G347900编码Ran-binding protein 1 domain-containing结构域,TraesCS7B02G329600编码含有五肽重复序列的蛋白质 (pentatricopeptide repeat-containing protein)。
表 5 筛选获得候选基因信息Table 5. Screening for candidate gene information性状
Trait物理位置
Pos (bp)染色体
Chr基因
Gene标记
Marker基因注释或编码蛋白
Gene annotation or coding proteinGNA 13553770 7A TraesCS7A02G405600 wsnp_CAP11_c1761_958064 真核天冬氨酰蛋白酶家族蛋白
Eukaryotic aspartyl protease family proteinTSS 7069765 3B TraesCS3B02G365500 Excalibur_c62826_254 脱氢酶样蛋白
Dehydrogenase-like proteinGWP,GNC,StNC 2597017 2A TraesCS2A02G022700 BobWhite_c10673_330 ATP依赖性Clp蛋白酶衔接子蛋白clpS
ATP-dependent Clp protease adapter protein clpSGWP,GNC,StNC 10197360 2A TraesCS2A02G140400 Ex_c721_2127 DNA修复蛋白类蛋白
DNA repair protein-like proteinGWP,GNC,StNC 10197360 2A TraesCS2A02G142200 Kukri_c42616_467 F-box家族蛋白
F-box family proteinGWP,GNC,StNC 8801680 3A TraesCS3A02G319900 BS00011612_51 保守的低聚高尔基复合体亚基1
Conserved oligomeric Golgi complex subunit 1GN,StNA,StNC 14437692 4A TraesCS4A02G461700 BobWhite_c47168_598 NBS-LRR耐药类蛋白
NBS-LRR resistance-like proteinGN,GWP,GNC,StNC 8288762 7B TraesCS7B02G343600 RAC875_c1742_2710 精氨酸-谷氨酸二肽重复蛋白
Arginine-glutamic acid dipeptide repeats proteinGWP,GNC,StNC 8398084 7B TraesCS7B02G347900 RAC875_c7251_656 含Ran结合蛋白1结构域
Ran-binding protein 1 domain-containingGWP,GNC,StNC 7843667 7B TraesCS7B02G329600 Tdurum_contig19852_242 含有五肽重复序列的蛋白质
Pentatricopeptide repeat-containing protein注(Note):TSS—小穗数 Total spikelet number per spike;GN—穗粒数 Grain number per spike;GWP—单株籽粒重 Grain weight per plant;GNC—籽粒氮含量 Grain N content;StNC—秸秆氮含量 Straw N content;GNA—单株籽粒氮累积量 Grain N accumulation per plant;StNA—单株秸秆氮累积量 Straw N accumulation per plant. 3. 讨论
3.1 小麦产量和氮效率相关性状的显著关联位点
本研究采用GLM + Q一般线性回归和MLM + Q + K混合线性模型相结合的方法,并在P ≤ 0.001下筛选显著关联SNP位点,可以有效地消除由群体结构存在而引起的伪关联,增加关联分析结果的可信度。不同环境条件下检测出的位点存在不一致的现象,很有可能是环境影响的结果,这也是多基因控制的数量性状的突出特点,但也很难排除关联结果的假阳性可能。而有些关联的位点则在多个环境均能被检测到,这些位点可视为相对稳定的位点。在本研究定位结果中,发现关联不同性状的标记有些位于染色体同一区域,即表现一因多效,有些位于相近区域即表现紧密连锁。无论是单个标记的“一因多效”还是不同标记间的“紧密连锁”,它们往往在染色体上形成多性状共定位位点,这与QTL簇的功能大致相同。
尽管目前在小麦上的关联分析研究已有大量报道,但是针对于小麦养分效率尤其是氮效率方面的关联分析研究仍罕见报道。利用遗传群体进行QTL分析也可获得与目标性状遗传控制有关的分子标记位点/基因,人们在不同氮营养条件下检测到大量的氮效率相关性状的QTL,这些QTL在小麦21条染色体上均有分布[24-29]。这些研究的结果表明QTL作图是挖掘小麦氮效率重要基因,解析遗传机制的有效方法。由于试验材料和研究条件的不同加上氮养分效率性状遗传控制的复杂性,不同的研究结果虽然具有一定的参考价值,但是共定位的标记又很难找到,因此大量的QTL研究在目前的条件下仍然是必需的。
与前人研究结果相似,本研究中产量和氮效率相关性状的QTL也几乎定位在小麦的全部21条染色体 (1A、3D和7D除外),分布广泛。其中氮素吸收效率相关性状的13个QTL分布在6条染色体上 (2A、2B、3A、3B、4A和7B);氮素利用效率相关性状的4个QTL位点定位在4条染色体上 (1D、2D、6A和7B)。成熟期产量性状的55个QTL分布更为广泛,除了1A、1D、3D、5D、6A、6B、6D和7D外都有分布。本研究在不同氮处理下检测到了与成熟期产量及氮效率相关性状显著关联的382个SNP标记位点,其中有305个SNP标记位点仅在1个关联分析环境中检测到,占总数的79.84%,这表明产量和氮效率相关性状是极为复杂的数量性状,由众多微效基因控制,这些微效基因可能与不同氮素环境的适应性有关,受环境影响显著。由于受到关联群体大小、分子标记图谱精密度以及环境对表型鉴定的干扰等诸多因素的影响,也很难排除这些关联定位结果存在部分假阳性的可能。试验也检测到至少4个环境下与同一性状稳定关联的SNP标记位点4个 (BobWhite_c47168_598、Kukri_c31599_1456、wsnp_CAP11_c1761_958064和Excalibur_c62826_254),它们分别与穗粒数、籽粒全氮含量和小穗数显著关联,这些SNP标记位点具有较强的环境稳定性,可靠性较高,值得深入研究。前人针对小麦氮素利用效率遗传机制也开展了多项研究,在小麦21条染色体上发现一批控制小麦氮素利用效率相关遗传位点[24-25, 28],但是由于多数研究采用的标记为RFLP、SSR或者DArT标记,根据现有小麦遗传或物理图谱,无法进行可靠的对比,暂时无法确定是否与本研究发现位点一致。
此外,试验还发现了一些同时定位了多个性状的位点,共有12个位点至少同时定位了3个性状,它们分别为BobWhite_c10673_330、BobWhite_c26296_83、Ex_c721_2127、Kukri_c42616_467、tplb0045j05_1405、BS00011612_51、Kukri_c80104_809、BobWhite_c47168_598、Kukri_c31599_1456、RAC875_c1742_2710、RAC875_c7251_656和Tdurum_contig19852_242。许多养分效率性状QTL分析结果也表明,在染色体上存在着热点QTL区域[26, 30]。张国华[30]研究结果也显示了多个性状的相互关联现象,这种现象可能是控制这些性状的QTL相互连锁或是由于某个QTL的一因多效引起的。同一标记与多个相关性状的关联可用于解释数量性状之间可能存在的遗传相关性。我们定位到的这12个位点涉及的性状主要有单株籽粒重、籽粒氮累积量、秸秆氮累积量、穗粒数、秸秆全氮含量,结合性状表型数据之间的相关性可知,这5个性状几乎都在P < 0.01的水平上均存在极显著相关。其中10个位点均与单株籽粒重、籽粒氮累积量和/或秸秆氮累积量这3个性状显著关联,这在QTL水平上间接证明了单株籽粒重与氮效率性状特别是氮素吸收累积量之间的显著关系。
3.2 小麦产量和氮效率相关性状的候选基因预测
之前的小麦氮效率研究中,已确定的候选基因分为4类:氨基酸生物合成、转录因子 (TF)、硝酸盐和铵转运蛋白,在小麦基因组最新测序数据中,已经报道了16个低亲和力硝酸盐转运蛋白 (TaNPFs)、1个双亲和力硝酸盐转运体、4个高亲和力硝酸盐转运体 (TaNRT2)的基因。这些基因在氮素的吸收、转运、同化、再利用过程中发挥作用,从而提高小麦氮效率[31]。本研究基于全基因组关联分析结果,利用环境稳定性强的4个位点标记和与产量、氮效率共定位的12个位点标记 (共14个位点) 进行了LD block划分,并对目标区段所有候选基因进行功能注释查询,从中筛选出与小麦产量和氮效率相关性状相关的10个候选基因。其中有7个基因在核酸代谢、氨基酸代谢及蛋白质代谢过程中发挥作用,其余3个基因在蛋白水解和离子跨膜运输过程中发挥作用 (表5)。例如,TraesCS7A02G405600编码真核天冬氨酰蛋白酶家族蛋白,在细胞组成过程中,这类家族蛋白比较集中于内膜系统,除此之外,在植物细胞壁以及膜锚定中也有富集;在生物学过程中,这类蛋白家族成员集中在蛋白水解功能;在分子功能上,它们则集中体现了天冬氨酸内切酶活性。TraesCS3B02G365500编码脱氢酶样蛋白,在生物学过程中,与氧化还原过程 相关;在分子功能上,它们则集中体现了氧化还原酶活性。TraesCS7B02G329600编码含有五肽重复序列的蛋白质,五肽重复序列 (PPR) 蛋白质是陆地植物中最大的蛋白质家族之一,它们在细胞中发挥作用,进而在植物生长和发育过程中发挥生理作用。典型的PPR蛋白靶向线粒体或叶绿体,结合一个或几个细胞器转录物,并通过改变RNA序列、转换、加工或翻译影响其表达。它们的联合作用对细胞器的生物发生和功能产生深远影响,因此对光合作用、呼吸作用、植物发育和环境响应产生深远的影响;TraesCS2A02G142200编码1个F-box家族蛋白,不仅作为SCF复合体中的成分参与泛素介导的蛋白降解途径,而且通过非SCF复合体的方式行使多种生理功能;这些基因可能通过影响植物细胞氮素的基础代谢过程最终影响氮效率,可以作为重要的氮效率候选基因开展更进一步的研究。
氮代谢是一个非常复杂的生物学过程,参与了植物生命活动过程中的方方面面,除了基础的氮同化系统外,还存在很多未知基因在氮代谢过程中发挥作用,本研究利用遗传图谱直接定位到的SNP标记位点预测到10个参与氮转运和代谢过程的基因,根据其可能的功能,它们很有可能在遗传群体中存在有利的等位变异,并对小麦氮效率和籽粒产量的形成具有重要价值。然而,这些候选基因仍需要进一步的确认和验证,这将有助于加深人们对小麦氮效率分子生理基础的认识,也将有助于推动小麦氮效率遗传改良的发展。
4. 结论
氮供应水平对小麦成熟期产量和氮效率相关性状及其显著关联SNP标记位点均有显著影响。本试验中与产量及养分效率有关的14个性状共检测到382个显著关联位点,其中79.84%的SNP标记仅在一个氮处理环境中出现,环境稳定性可能较差。同时,有4个位点在4个环境条件下均被检测到,有12个SNP标记位点同时与至少3个性状显著关联,这些位点是与成熟期产量及氮效率相关性状的遗传控制有关的热点位点。根据这些热点位点预测了10个与产量及氮效率相关性状相关的候选基因,值得深入探讨。
-
图 1 小麦成熟期产量及氮效率相关性状间的相关系数
[注(Note):SN—穗数 Sprike number;TSS—小穗数 Total spikelet number per spike;BSSS—基部不育小穗数 Bottom sterile spikelet number per spike;TSSS—顶部不育小穗数 Top sterile spikelet number per spike;GN—穗粒数 Grain number per spike;TGW— 千粒重 Thousand grains weight;GWP—单株籽粒重Grain weight per plant;StDW—单株秸秆干重 Straw dry weight per plant;GNC—籽粒氮含量 Grain N content ;StNC—秸秆氮含量 Straw N content;GNUE—籽粒氮利用效率 Grain nitrogen use efficiency;StNUE—秸秆氮利用效率 Straw nitrogen use efficiency.]
Figure 1. Correlation coefficients (r) between traits related to yield and nitrogen efficiency of wheat at maturity
图 2 供试小麦材料的群体结构
[注(Note):图a为供试材料的K值与ΔK的关系;图b为基于遗传距离的NJ聚类图;图c为群体结构;2个亚群用不同颜色表示。Figure a shows graphical relationship between K and ΔK for wheat accessions;Figure b shows neighbor-joining tree of 134 wheat accessions. Figure c shows population structure of wheat accessions. The two subgroups identified from the tree are in different colors.]
Figure 2. Population structure analysis of tested wheat materials based on unlinked SNP markers
表 1 供试小麦品种
Table 1 Wheat cultivars used in this study
序号
No.品种(系)名称
Variety/line name序号
No.品种(系)名称
Variety/line name序号
No.品种(系)名称
Variety/line name序号
No.品种(系)名称
Variety/line name序号
No.品种(系)名称
Variety/line name1 昌乐5号 28 山农45 55 淄12 82 新麦18 109 石新616 2 德选1号 29 山农483 56 954 (7)-8 83 新麦26 110 石新618 3 济麦20 30 山农664 57 BSS 84 豫麦49 111 石新828 4 济麦21 31 山农8355 58 LS3283 85 豫麦54 112 石优17号 5 济麦22 32 山农紫麦1号 59 LS4211 86 郑麦9023 113 唐麦8号 6 济南17 33 山融3号 60 LS4697 87 中优9507 114 小偃81 7 济宁16 34 泰农18 61 LS4942 88 周99233 115 AN2 8 济宁17 35 泰山21 62 LS6045 89 周黑麦1号 116 AN3 9 科信9号 36 泰山22 63 LS6109 90 周麦24 117 蓝58 10 莱州137 37 泰山23 64 M8008-2 91 藁优9618 118 陕627 11 莱州95021 38 泰山24 65 贵农35选21 92 邯00-7086 119 陕农138 12 良星99 39 郯麦98 66 薛紫麦 93 河农827 120 陕农534 13 临麦4号 40 潍麦7号 67 黑小麦76 94 河农4198 121 西农85 14 临麦6号 41 潍麦8号 68 晋麦33号 95 河农6049 122 西农213 15 鲁麦14 42 汶农5号 69 临丰3号 96 衡0628 123 西农889 16 鲁麦21 43 汶农6号 70 临旱822 97 衡6599 124 西农9871 17 鲁麦22 44 鑫289 71 临抗16号 98 冀5265 125 小偃22 18 鲁麦23 45 烟5072 72 临优145 99 冀丰703 126 小偃92 19 齐丰1号 46 烟99102 73 品资旱99-2 100 金麦1号 127 小偃216 20 山农0431 47 烟99603 74 平阳298 101 科农199 128 运丰139 21 山农1186 48 烟农0428 75 洛旱6号 102 廊研43号 129 淮麦16 22 山农12 49 烟农15 76 洛旱8号 103 石54 130 淮麦18 23 山农15 50 烟农19 77 洛旱21号 104 石4185 131 川35050 24 山农17 51 烟农21 78 洛旱23号 105 石家庄8号 132 中国春 25 山农18 52 烟农22 79 洛旱24号 106 石麦15号 133 澳大利亚红麦 26 山农21 53 烟农23 80 漯珍1号 107 石麦16号 134 加拿大超强筋麦 27 山农23 54 烟农836 81 新麦16 108 石麦18号 注(Note):表中品种 (系) 来源 Source of cultivar (line) listed in the table:1~66,中国山东 Shandong,China;67~74,中国山西Shanxi,China;75~90,中国河南 Henan,China;91~114,中国河北 Hebei,China;115~128,中国陕西 Shaanxi,China;129~130,中国江苏 Jiangsu,China;131~132,中国四川 Sichuan,China;133,澳大利亚 Australia;134,加拿大 Canada. 表 2 小麦成熟期产量和氮效率相关性状的统计参数和遗传力
Table 2 Statistical parameters and heritability of traits related to yield and nitrogen efficiency of wheat at maturity
性状
Traits处理
Treatment均值
Average变异系数
CV (%)遗传力 (%)
Heritability性状
Traits处理
Treatment均值
Average变异系数
CV (%)遗传力 (%)
Heritability穗数 SN T1E1 12.80 a 20.9 88.35 单株秸秆 StDW (g/plant) T1E1 28.69 a 18.3 92.97 T2E1 12.57 a 20.4 T2E1 22.12 b 26.3 T1E2 10.43 b 22.7 T1E2 27.80 a 20.9 T2E2 9.59 b 26.9 T2E2 21.09 b 22.1 小穗数 TSS T1E1 19.40 a 5.9 95.12 籽粒氮含量 GNC (g/kg) T1E1 22.95 b 9.3 91.96 T2E1 19.33 a 6.5 T2E1 21.59 b 10.2 T1E2 19.18 a 6.3 T1E2 25.33 a 9.4 T2E2 19.41 a 6.3 T2E2 23.35 ab 11.8 基部不育小穗数 BSSS T1E1 0.44 a 83.6 87.44 秸秆氮含量 StNC (g/kg) T1E1 5.48 b 22.2 75.16 T2E1 0.50 a 66.7 T2E1 3.99 c 18.0 T1E2 0.62 a 64.8 T1E2 7.30 a 14.9 T2E2 0.57 a 73.5 T2E2 4.95 b 18.7 顶部不育小穗数 TSSS T1E1 0.35 a 124.6 80.88 籽粒氮累积量 GNA (g/plant) T1E1 0.54 ab 18.7 83.96 T2E1 0.38 a 123.8 T2E1 0.42 c 22.9 T1E2 0.04 b 320.6 T1E2 0.60 a 23.4 T2E2 0.05 b 273.6 T2E2 0.49 bc 30.8 穗粒数 GN T1E1 58.22 a 10.3 93.03 秸秆氮累积量 StNA (g/plant) T1E1 0.16 b 26.7 82.59 T2E1 56.14 a 10.5 T2E1 0.09 c 24.4 T1E2 57.89 a 12.1 T1E2 0.20 a 19.4 T2E2 57.59 a 12.1 T2E2 0.10 bc 30.2 千粒重 TGW (g) T1E1 45.05 b 9.5 85.30 籽粒氮利用率 GNUE (g/kg) T1E1 1.03 a 21.8 87.67 T2E1 44.58 b 8.7 T2E1 0.90 a 26.1 T1E2 50.53 a 8.9 T1E2 0.93 a 24.7 T2E2 49.54 a 9.0 T2E2 0.90 a 29 籽粒重 GWP (g/plant) T1E1 23.59 a 18.3 88.17 秸秆氮利用率 StNUE (g/kg) T1E1 5.24 a 24.8 85.98 T2E1 19.45 b 24.3 T2E1 5.54 a 24.7 T1E2 23.53 a 22.5 T1E2 3.81 b 32.0 T2E2 21.08 ab 28.5 T2E2 4.26 b 27.5 注(Note):SN—Sprike number;TSS—Total spikelet number per spike;BSSS—Bottom sterile spikelet number per spike;TSSS—Top sterile spikelet number per spike;GN—Grain number per spike;TGW—Thousand grains weight;GWP—Grain dry weight per plant;StDW—Straw dry weight per plant;GNC—Grain N content;StNC—Straw N content per plant;GNA—Grain N accumulation per plant;StNA—Straw N accumulation per plant;GNUE—Grain nitrogen use efficiency;StNUE—Straw nitrogen use efficiency. E1和E2代表两个试验年份 E1 and E2 represent the two experimental years;T1和T2分别代表正常氮和低氮处理 T1 and T2 represent normal and low nitrogen supply treatments;同列数据后不同小写字母代表两个处理之间差异显著 (P < 0.05) Values followed by different lowercase letters indicate significant difference between two treatments at 0.05 level. 表 3 小麦成熟期产量及氮效率相关性状稳定关联标记位点
Table 3 The environmental stable markers which associated with maturity yield and N efficiency related traits
QTL 性状
Traits环境
Environment标记
Marker染色体
Chr位置
Pos (bp)P值 P-value 贡献率
R2 (%)最大值
Max最小值
MinQGn-2D-1 GN T1AV,T2AV,T2E2 BS00011740_51 2D 4004568 8.03 × 10–4 6.06 × 10–4 8.85~9.28 QTss-3B-1 TSS T1AV,T2AV,T2E2 BS00032692_51 3B 7069765 4.18 × 10–4 2.21 × 10–4 9.85~10.85 QTss-3B-5 TSS T1AV,T2AV,T2E2 Excalibur_c5977_1409 3B 7069765 4.18 × 10–4 2.21 × 10–4 9.85~10.85 QTss-3B-6 TSS T1AV,T2AV,T2E2 Excalibur_c5977_440 3B 7069765 4.18 × 10–4 2.21 × 10–4 9.85~10.85 QTss-3B-7 TSS T1AV,T1E1,T2AV,T2E2 Excalibur_c62826_254 3B 7069765 6.27 × 10–4 5.56 × 10–5 9.23~13.05 QGn-4A-1 GN T1E1,T1E2,T1AV,T2E2 BobWhite_c47168_598 4A 14437692 9.20 × 10–3 3.89 × 10–4 11.27~12.54 QGn-4A-2 GN T1E1,T2AV,T2E2 GENE-0689_791 4A 12712246 9.99 × 10–4 3.78 × 10–4 10.96~12.59 QGn-5B GN T1AV,T1E1,T1E2,T2AV Kukri_c31599_1456 5B 10124835 6.50 × 10–4 1.06 × 10–4 11.68~15.02 QGnc-7A-3 GNC T1AV,T1E1,T1E2,T2E1 wsnp_CAP11_c1761_958064 7A 13553770 8.58 × 10–4 8.03 × 10–5 11.30~15.09 注(Note):AV—平均值 Average;GN—穗粒数 Number of grains per spike;TSS—小穗数 Total spikelets;GNC—籽粒氮含量 Grain N concentration. E1和E2代表两个试验年份,T1和T2分别代表正常氮和低氮处理 E1 and E2 represent the two experimental years, and T1 and T2 represent normal and low nitrogen treatments,respectively. 表 4 小麦成熟期产量及氮效率相关性状簇集位点
Table 4 The marker locations wheat yield and N efficiency related traits clustered at maturity stage
簇
Cluster染色体
ChromosomeQTL数
No. of QTLQTL 处理
Treatment标记
Marker贡献率 (%)
R2C1 2A 3 QGwp-2A T2AV,T2E2 BobWhite_c10673_330 11.14~20.87 QGna-2A T2AV,T2E2 QStna-2A T2E2 C2 2A 3 QGwp-2A T2AV,T2E2 BobWhite_c26296_83 11.17~21.03 QGna-2A T2AV,T2E2 QStna-2A T2E2 C3 2A 3 QGwp-2A T2E2 Ex_c721_2127 12.23~20.85 QGna-2A T2AV,T2E2 QStna-2A T2E2 C4 2A 3 QGwp-2A T2E2 Kukri_c42616_467 13.61~18.36 QGna-2A T2AV,T2E2 QStna-2A T2E2 C5 2A 3 QGwp-2A T2E2 tplb0045j05_1405 13.61~18.36 QGna-2A T2AV,T2E2 QStna-2A T2E2 C6 3A 3 QGwp-3A T2E2 BS00011612_51 12.52~21.31 QGna-3A T2AV,T2E2 QStna-3A T2E2 C7 3A 4 QGwp-3A T2AV,T2E2 Kukri_c80104_809 12.23~22.18 QGna-3A T2AV,T2E2 表 5 筛选获得候选基因信息
Table 5 Screening for candidate gene information
性状
Trait物理位置
Pos (bp)染色体
Chr基因
Gene标记
Marker基因注释或编码蛋白
Gene annotation or coding proteinGNA 13553770 7A TraesCS7A02G405600 wsnp_CAP11_c1761_958064 真核天冬氨酰蛋白酶家族蛋白
Eukaryotic aspartyl protease family proteinTSS 7069765 3B TraesCS3B02G365500 Excalibur_c62826_254 脱氢酶样蛋白
Dehydrogenase-like proteinGWP,GNC,StNC 2597017 2A TraesCS2A02G022700 BobWhite_c10673_330 ATP依赖性Clp蛋白酶衔接子蛋白clpS
ATP-dependent Clp protease adapter protein clpSGWP,GNC,StNC 10197360 2A TraesCS2A02G140400 Ex_c721_2127 DNA修复蛋白类蛋白
DNA repair protein-like proteinGWP,GNC,StNC 10197360 2A TraesCS2A02G142200 Kukri_c42616_467 F-box家族蛋白
F-box family proteinGWP,GNC,StNC 8801680 3A TraesCS3A02G319900 BS00011612_51 保守的低聚高尔基复合体亚基1
Conserved oligomeric Golgi complex subunit 1GN,StNA,StNC 14437692 4A TraesCS4A02G461700 BobWhite_c47168_598 NBS-LRR耐药类蛋白
NBS-LRR resistance-like proteinGN,GWP,GNC,StNC 8288762 7B TraesCS7B02G343600 RAC875_c1742_2710 精氨酸-谷氨酸二肽重复蛋白
Arginine-glutamic acid dipeptide repeats proteinGWP,GNC,StNC 8398084 7B TraesCS7B02G347900 RAC875_c7251_656 含Ran结合蛋白1结构域
Ran-binding protein 1 domain-containingGWP,GNC,StNC 7843667 7B TraesCS7B02G329600 Tdurum_contig19852_242 含有五肽重复序列的蛋白质
Pentatricopeptide repeat-containing protein注(Note):TSS—小穗数 Total spikelet number per spike;GN—穗粒数 Grain number per spike;GWP—单株籽粒重 Grain weight per plant;GNC—籽粒氮含量 Grain N content;StNC—秸秆氮含量 Straw N content;GNA—单株籽粒氮累积量 Grain N accumulation per plant;StNA—单株秸秆氮累积量 Straw N accumulation per plant. -
[1] 崔振岭, 石立委, 徐久飞, 等. 氮肥施用对冬小麦产量、品质和氮素表观损失的影响研究[J]. 应用生态学报, 2005, 16(11): 2071–2075. DOI: 10.3321/j.issn:1001-9332.2005.11.012 Cui Z L, Shi L W, Xu J F, et al. Effects of nitrogen fertilizer application on winter wheat yield, quality and apparent nitrogen loss[J]. Chinese Journal of Applied Ecology, 2005, 16(11): 2071–2075. DOI: 10.3321/j.issn:1001-9332.2005.11.012
[2] Liang S, Li Y, Zhang X, et al. Response of crop yield and nitrogen use efficiency for wheat–maize cropping system to future climate change in northern China[J]. Agricultural and Forest Meteorology, 2018, 262: 310–321.
[3] Davidson E A. The contribution of manure and fertilizer nitrogen to atmospheric nitrous oxide since 1860[J]. Nature Geoscience, 2009, 2(9): 659–662. DOI: 10.1038/ngeo608
[4] Frels K, Guttieri, M, Joyce, B, et al. Evaluating canopy spectral reflectance vegetation indices to estimate nitrogen use traits in hard winter wheat[J]. Field Crops Research, 2018, 217: 82–92.
[5] Hitz K, Clark A J, Van Sanford D A. Identifying nitrogen-use efficient soft red winter wheat lines in high and low nitrogen environments[J]. Field Crops Research, 2017, 200: 1–9.
[6] Kong F M, Guo Y, Liang X, et al. Potassium (K) effects and QTL mapping for K efficiency traits at seedling and adult stages in wheat[J]. Plant and Soil, 2013, 373(1–2): 877–892. DOI: 10.1007/s11104-013-1844-4
[7] Moll R H, Kamprath E J, Jackson, Jackson W A. Analysis and interpretation of factors which contribute to efficiency of nitrogen utilization1[J]. Agronomy Journal, 1982, 74(3): 562–564. DOI: 10.2134/agronj1982.00021962007400030037x
[8] Clárk R B. Plant genotype differences in the uptake, translocation, accumulation, and use of mineral elements required for plant growth[J]. Plant and Soil, 1983, 72(2–3): 49–70.
[9] Gouis J L, Béghin D, Heumez E, et al. Genetic differences for nitrogen uptake and nitrogen utilisation efficiencies in winter wheat[J]. European Journal of Agronomy, 2000, 12(3): 163–173.
[10] Mahjourimajd S, Kuchel H, Langridge P, et al. Evaluation of Australian wheat genotypes for response to variable nitrogen application[J]. Plant and Soil, 2015, 399(1–2): 247–255.
[11] Wang Y Y, Sun X Y, Zhao Y, et al. Enrichment of a common wheat genetic map and QTL mapping for fatty acid content in grain[J]. Plant Science, 2011, 181(1): 65–75. DOI: 10.1016/j.plantsci.2011.03.020
[12] Sukumaran S, Dreisigacker S, Lopes M, et al. Genome-wide association study for grain yield and related traits in an elite spring wheat population grown in temperate irrigated environments[J]. Theoretical and Applied Genetics, 2015, 128(2): 353–363. DOI: 10.1007/s00122-014-2435-3
[13] Suraj G, Sujan M, Michael B J, et al. Genome--wide association study reveals novel quantitative trait loci associated with resistance to multiple leaf spot diseases of spring wheat[J]. PLoS ONE, 2014, 9(9): e108179. DOI: 10.1371/journal.pone.0108179
[14] Mengistu D K, Kidane Y G, Catellani M, et al. High-density molecular characterization and association mapping in Ethiopian durum wheat landraces reveals high diversity and potential for wheat breeding[J]. Plant Biotechnology Journal, 2016, 14(9): 1800–1812. DOI: 10.1111/pbi.12538
[15] Oyiga B C, Sharma R C, Baum M, et al. Allelic variations and differential expressions detected at quantitative trait loci for salt stress tolerance in wheat[J]. Plant, Cell & Environment, 2018, 41(1): 919–935.
[16] Jighly A, Alagu M, Makdis F, et al. Genomic regions conferring resistance to multiple fungal pathogens in synthetic hexaploid wheat[J]. Molecular Breeding, 2016, 36(9): 127. DOI: 10.1007/s11032-016-0541-4
[17] Naruoka Y, Garland-Campbell K A, Carter A H J T, et al. Genome-wide association mapping for stripe rust (Puccinia striiformis F. sp. tritici) in US Pacific Northwest winter wheat (Triticum aestivum L.)[J]. Theoretical and Applied Genetics, 2015, 128(6): 1083–1101. DOI: 10.1007/s00122-015-2492-2
[18] Guo Z, Chen D, Alqudah A M, et al. Genome-wide association analyses of 54 traits identified multiple loci for the determination of floret fertility in wheat[J]. New Phytologist, 2017, 214(1): 257–270. DOI: 10.1111/nph.14342
[19] Sanu A, Narinder S, Satinder K, et al. Genome-wide association study of grain architecture in wild wheat aegilops tauschii[J]. Frontiers in Plant Science, 2017, 8: 886. DOI: 10.3389/fpls.2017.00886
[20] Knapp S J, Stroup W W, Ross W M J C. Exact confidence intervals for heritability on a progeny mean basis[J]. Crop Science, 1985, 25(1): 192–194. DOI: 10.2135/cropsci1985.0011183X002500010046x
[21] Pritchard J K, Stephens M, Donnelly P. Inference of population structure using multilocus genotype data[J]. Genetics, 2000, 155(2): 945–959. DOI: 10.1093/genetics/155.2.945
[22] Evanno G, Regnaut S, Goudet J. Detecting the number of clusters of individuals using the software structure: A simulation study[J]. Molecular Ecology, 2005, 14(8): 2611–2620. DOI: 10.1111/j.1365-294X.2005.02553.x
[23] Wang S, Wong D, Forrest K, et al. Characterization of polyploid wheat genomic diversity using a high-density 90, 000 single nucleotide polymorphism array[J]. Plant Biotechnology Journal, 2014, 12: 787–796. DOI: 10.1111/pbi.12183
[24] Cui F, Zhao C, Ding A, et al. Construction of an integrative linkage map and QTL mapping of grain yield-related traits using three related wheat RIL populations[J]. Theoretical and Applied Genetics, 2014, 127(3): 659–675. DOI: 10.1007/s00122-013-2249-8
[25] Fontaine J X, Ravel C, Pageau K, et al. A quantitative genetic study for elucidating the contribution of glutamine synthetase, glutamate dehydrogenase and other nitrogen-related physiological traits to the agronomic performance of common wheat[J]. Theoretical and Applied Genetics, 2009, 119(4): 645–662. DOI: 10.1007/s00122-009-1076-4
[26] Guo Y, Kong F M, Xu Y F, et al. QTL mapping for seedling traits in wheat grown under varying concentrations of N, P and K nutrients[J]. Theoretical and Applied Genetics, 2012, 124(5): 851–865. DOI: 10.1007/s00122-011-1749-7
[27] Habash D Z, Bernard S, Schondelmaier J, et al. The genetics of nitrogen use in hexaploid wheat: N utilisation, development and yield[J]. Theoretical and Applied Genetics, 2007, 114(3): 403–419. DOI: 10.1007/s00122-006-0429-5
[28] Quarrie S A, Steed A, Calestani C, et al. A high-density genetic map of hexaploid wheat (Triticum aestivum L.) from the cross Chinese Spring—SQ1 and its use to compare QTLs for grain yield across a range of environments[J]. Theoretical and Applied Genetics, 2005, 110(5): 865–880. DOI: 10.1007/s00122-004-1902-7
[29] Xu Y, Wang R, Tong Y, et al. Mapping QTLs for yield and nitrogen-related traits in wheat: Influence of nitrogen and phosphorus fertilization on QTL expression[J]. Theoretical and Applied Genetics, 2014, 127(1): 59–72. DOI: 10.1007/s00122-013-2201-y
[30] 张国华. 黄淮麦区小麦品种(系)产量性状与分子标记的关联分析[D]. 山东泰安: 山东农业大学硕士学位论文, 2013. Zhang G H. Association analysis of yield traits and molecular markers of wheat varieties (lines) in Huang-huai wheat region[D]. Tai'an, Shandong: MS Thesis of Shandong Agricultural University, 2013.
[31] Kumar A, Sharma M, Kumar S, et al. Functional and structural insights into candidate genes associated with nitrogen and phosphorus nutrition in wheat (Triticum aestivum L.)[J]. International Journal of Biological Macromolecules, 2018, 118: 76–91. DOI: 10.1016/j.ijbiomac.2018.06.009
-
期刊类型引用(3)
1. 康利允,李晓慧,高宁宁,梁慎,常高正,李海伦,王慧颖,徐小利,赵卫星. 甜瓜耐低氮且氮高效种质资源评价与筛选. 山东农业科学. 2023(04): 24-33 .  百度学术
百度学术
2. 康利允,李晓慧,高宁宁,梁慎,常高正,李海伦,王慧颖,徐小利,赵卫星. 氮高效甜瓜种质资源的筛选与评价. 西南农业学报. 2023(06): 1271-1278 .  百度学术
百度学术
3. 史亚歌,窦春蕊,郭柏寿. 科技论文图表一体化应用与实践. 学报编辑论丛. 2022(00): 162-169 .  百度学术
百度学术
其他类型引用(7)
-
其他相关附件
-
PDF格式
20549_print 点击下载(983KB)
-



 下载:
下载: