Associations between viral communities in rice straw-incorporated soil and nitrogen cycle-related genes in associated bacteria
-
摘要:目的
病毒是稻田土壤微生物群落的重要组成部分,在调节宿主群落和土壤元素生物地球化学循环过程中发挥着不可忽视的作用。探析稻草还田下稻田土壤病毒与宿主相互作用机制以及辅助代谢基因(AMGs) 的表达,有助于揭示病毒在稻田土壤氮循环过程中的深远影响,为了解病毒在土壤元素生物地球化学循环过程中的作用提供新视角。
方法长期稻草还田定位试验位于广东省惠州市,始于2015年。选择稻草不还田对照 (CK)、1.1倍稻草还田 (S1,单季稻草还田量7075 kg/hm2)、1.2倍稻草还田 (S2,单季稻草还田量7750 kg/hm2)、1.3倍稻草还田 (S3,单季稻草还田量8425 kg/hm2)、稻草全量还田 (S,单季稻草还田量6400 kg/hm2) 5个处理土壤样品,采用宏基因组和宏病毒组测序技术,分析了稻田土壤病毒、细菌以及AMGs结构。
结果稻田土壤病毒优势科为圆环病毒科 (Circoviridae)、微小噬菌体科 (Microviridae)、双生病毒科(Geminiviridae) 和类双生病毒科 (Genomoviridae),但仍有84.62%~93.92%病毒科未被分类或未知。稻草还田量对稻田土壤病毒的影响显著,S处理中圆环病毒科 (Circoviridae) 的丰度显著增加 (P<0.05),S1、S2和S3处理中类双生病毒科 (Genomoviridae)的丰度显著降低,而未分类或未知病毒科的相对丰度显著增加(P<0.05)。稻田土壤优势细菌门为变形菌门 (Proteobacteria)、绿弯菌门 (Chloroflexi) 和酸杆菌门 (Acidobacteria),稻草还田量和水稻生育期对土壤优势细菌门均无显著影响。共检测到8条病毒contigs携带有机氮转化基因glnA、gltD、gdhA和ncd2/npd,这些病毒contigs在稻草还田处理中的丰度几乎全部高于CK。宿主预测结果表明,2条携带氮循环基因的病毒contigs宿主为假单胞菌门 (Pseudomonadota) 和放线菌门 (Actinomycetota),其余病毒contigs未预测到宿主。细菌ncd2/npd基因丰度与微生物量氮含量呈显著正相关(P=0.015,n=10),而与铵态氮、硝态氮总量之间没有显著相关性(P=0.108,n=10)。偏最小二乘路径模型显示,稻草还田显著影响了稻田土壤细菌的α多样性 (P<0.0007);细菌携带的有机氮转化基因ncd2/npd显著影响了稻田土壤微生物量氮的含量 (P<0.0039)。
结论稻草还田显著影响稻田土壤中病毒类群的相对丰度,促进病毒所携带的有机氮转化基因表达出更多的辅助代谢基因,进而有利于土壤氮循环。高稻草还田量通过影响土壤病毒和细菌的α多样性,间接影响细菌携带的ncd2/npd基因的相对丰度,最终对有机氮转化过程产生重要影响。
Abstract:ObjectivesViruses are an important component of soil microbial communities in paddy fields, and play a non-negligible role in regulating host communities and elemental biogeochemical cycles. Exploring the interaction mechanism between viruses and hosts and the expression of accessory metabolic genes (AMGs) in paddy fields under rice straw returning is helpful to reveal the far-reaching effects of viruses on soil nitrogen cycling in paddy fields, aiming to provide a new perspective for understanding the role of viruses in the process of soil element biogeochemical cycling.
MethodsA long-term straw return positioning experiment has been conducted in Huizhou, Guangdong Province, since 2015. Soil samples were collected in 2023 from five treatment plots: no straw returning (CK), total straw returning (S, the amount of straw returned to the field in a single season is 6400 kg/hm2), and 1.1, 1.2, and 1.3 times of the whole straw return amount (S1, S2, and S3, the amount of straw returned to the field in a single season are 7075 kg/hm2, 7750 kg/hm2, 8425 kg/hm2, respectively). The community composition of soil viruses and bacteria, and the relative abundances of viruses, bacteria, and AMGs involved in the nitrogen cycle process were analyzed by metagenomic and metavirome sequencing technology.
ResultsThe dominant families of soil viruses in paddy fields were Circoviridae, Microviridae, Geminiviridae and Genomoviridae, but 84.62%−93.92% of the viridae were still unclassified or unknown. S treatment increased the abundance of Circoviridae significantly, while S1, S2 and S3 treatments decreased the abundance of Genomoviridae but increased the relative abundances of unclassified or unknown viridae significantly (P<0.05). The dominant bacterial phylum in paddy field were Proteobacteria, Chloroflexi and Acidobacteria, and the straw return amount and the growth period of rice had no significant effect on the dominant bacterial phylum. A total of 8 viral contigs carrying the organic nitrogen transformation genes glnA, gltD, gdhA and ncd2/npd, were identified, and the abundance of these viral contigs in the straw returning treatment was almost all higher than that of CK. The results of host prediction showed that the hosts of two virus contigs carrying nitrogen cycle genes were Pseudomonas and Actinomycetota, while the hosts for the other virus contigs were unable to predict. The abundance of bacterial ncd2/npd gene was positively correlated with microbial biomass nitrogen content (P=0.015, n=10), but not with total ammonium nitrogen and nitrate nitrogen (P=0.108, n=10). The partial least squares path model showed that straw return significantly affected the alpha diversity of soil bacteria (P<0.0007). The organic nitrogen transformation gene ncd2/npd carried by bacteria significantly affected the content of soil microbial biomass nitrogen (P<0.0039).
ConclusionsStraw returning significantly affected the relative abundance of virus taxa in paddy field, which could promote the expression of more AMGs encoded by viruses and play an important role in soil nitrogen cycling. By affecting the alpha diversity of soil viruses and bacteria, the amount of rice straw returned to the field indirectly affects the relative abundance of ncd2/npd genes carried by bacteria, and ultimately has an important impact on the organic nitrogen transformation process.
-
Keywords:
- paddy soil /
- rice straw return /
- virus /
- bacteria /
- nitrogen cycle-related genes
-
病毒是迄今为止地球上最丰富的生物类群,广泛分布于水体和陆地环境中[1]。但由于土壤环境的异质性和复杂性,对土壤病毒的研究面临着巨大的挑战,目前已发现的土壤病毒数量约为4.80×1031个[2],这可能仅仅只是冰山一角[3]。近年来,随着宏基因组和宏病毒组等测序技术的快速发展,土壤病毒的研究焦点已从探究不同土壤中病毒的群落组成、物种和遗传多样性[4−7],转向评估其生态环境功能。
土壤病毒具有多种生态功能,包括调节土壤微生物群落组成、结构和功能,介导水平基因转移和表达辅助代谢基因,在碳、氮、磷等元素生物地球化学循环等过程中具有至关重要的作用[8−9]。Zhu等[10]报道,在珠穆朗玛峰不同海拔高度土壤中,土壤的养分状况对病毒群落组成和多样性有显著影响,并发现有多种病毒携带碳水化合物代谢基因,参与珠穆朗玛峰土壤碳循环。Jin等[11]发现,土壤病毒可能通过编码碳水化合物活性酶(CAZyme),参与红树林土壤复杂多糖的生物质循环过程。Emerson等[12]发现,在永久冻土融化过程中,土壤病毒通过感染或者裂解产甲烷菌和表达辅助代谢基因编码糖苷水解酶,来参与土壤碳循环。Huang等[13]也报道,在不同的土壤养分条件下,土壤病毒可以表达辅助代谢基因phoB、gdh,从而参与土壤磷、氮元素循环过程。Han等[14]指出农田土壤中,病毒表达5种磷代谢基因dUTPase、MazG、phoH、Thy1、RNR,驱动宿主内部自身核苷酸合成,从而影响土壤农田生态系统磷循环过程。
稻田系统是最重要的农业系统,目前更多的研究主要集中在细菌组学特征上[15−16],而对稻田系统中病毒及病毒−细菌互作的相关研究仍然较少,尤其是稻草还田条件下土壤病毒群落特征及其在氮循环过程中的作用研究较少。鉴于土壤病毒在稻田生态系统中的重要性,本研究依托于广东省惠州市惠阳区惠阳农科服务中心试验站长期稻草还田定位试验,通过宏基因组和宏病毒组测序技术,比较不同量稻草还田的土壤病毒和细菌组学特征及其差异,并探讨这种差异是否也反映在水稻生育周期上。通过分析稻草还田下稻田土壤病毒与细菌之间的联系,以及病毒AMGs在氮循环过程中的作用,以期为了解土壤病毒在土壤氮素生物地球化学循环过程中的作用提供新视角。
1. 材料与方法
1.1 试验点概况
长期稻草还田定位试验始于2015年3月,试验点位于广东省惠州市惠阳区惠阳农科服务中心(113°20′E,23°08′N)。全年日照时数约为2020.6 h,平均气温为22℃,多年降水量为800~1500 mm,属于南亚热带季风气候。一年两熟,早稻于每年三月中下旬种植,六月底收割;晚稻于每年8月上旬种植,10月底收割。土壤类型为赤红壤,其基本化学性质如下:pH 5.20、碱解氮 191.50 mg/kg、有效磷 49.60 mg/kg、速效钾 149.40 mg/kg、土壤有机质22.80 g/kg。
1.2 试验设计
试验地稻草还田模式为早稻稻草粉碎还田+晚稻稻草粉碎还田,早稻与晚稻稻草还田量相同。田间试验设置5个处理:1)稻草不还田对照(CK);2) 1.1倍稻草还田,单季稻草还田量为7075 kg/hm2 (S1);3) 1.2倍稻草还田,单季稻草还田量为7750 kg/hm2 (S2);4) 1.3倍稻草还田,单季稻草还田量为8425 kg/hm2 (S3);5)稻草全量还田,单季稻草还田量为6400 kg/hm2 (S)。小区面积为72 m2 (6 m×12 m),随机区组排列。
稻草含水率为49.2%,N、P、K含量分别为9.20、2.12和16.42 g/kg。每个处理化肥用量除了钾肥(氯化钾,K2O 60%)略有差异外,氮肥(尿素,N 46%)和磷肥(钙镁磷肥,P2O5 15%)用量相同,早稻和晚稻的具体施肥量见表1。磷肥100%基肥深施;氮肥和钾肥60%基肥深施,剩余40%于水稻移栽15天后追施。
表 1 各处理早稻和晚稻化肥施用量(kg/hm2)Table 1. Chemical fertilizer input in each experimental treatment for early and late rice处理
Treatment总计 Total 早稻 Early rice 晚稻 Late rice N P2O5 K2O N P2O5 K2O N P2O5 K2O CK 310.5 94.5 270.0 155.3 47.3 135.0 155.3 47.3 135.0 S1 310.5 94.5 243.0 155.3 47.3 121.5 155.3 47.3 121.5 S2 310.5 94.5 216.0 155.3 47.3 108.0 155.3 47.3 108.0 S3 310.5 94.5 189.0 155.3 47.3 94.5 155.3 47.3 94.5 S 310.5 94.5 270.0 155.3 47.3 135.0 155.3 47.3 135.0 1.3 土壤样品采集与测定
在2023年8月10日(晚稻分蘖期)和2023年10月27日(晚稻成熟期),用土钻从各处理小区采集0—20 cm土层土壤,每个小区随机采集5点,剔除根系、枝叶和石砾等杂物,混匀后分3部分分别装至样品袋中,每个样品3个重复。一部分至4℃车载冰箱带回实验室−80℃保存,用于微生物宏基因组和宏病毒组测序;一部分自然风干后用于测定全氮(TN)含量;一部分新鲜土样用于测定硝态氮(NO3−-N)、铵态氮(NH4+-N)和微生物量氮(MBN)含量[17]。
1.4 土壤宏基因组测序
称取0.5 g储存于−80℃的新鲜土样,采用FastDNA SPIN试剂盒 (MP Bio-medicals, Solon, OH, USA) 提取土壤样品微生物总DNA[18−20]。对DNA样品质量进行检测,将检测合格的DNA样品加入fragmentation buffer,采用超声破碎仪进行随机打断,得到的短片段DNA用于文库构建,并进行质检。对于质检合格的文库采用Illumina NovaSeq 6000高通量测序平台进行PE150测序,测序得到的原始图像数据文件,经碱基识别(Base Calling)分析转化为原始测序序列 (Raw Reads)。采用质控流程软件Trimmomactic (参数为:LEADING:3TRAILING:3 SLIDINGWINDOW:5:20 MINLEN:50) 对获得的样品宏基因组数据进行质量控制,获得的高质量序列用于下游数据分析,得到Clean reads共1.58×109条。利用MEGAHIT (https://github.com/voutcn/megahit) 对质量控制之后的Clean Data进行denovo拼接 (K-mer选取k-min 35,k-max 95,k-step 20),并将各样品未被利用上的Reads放在一起进行混合组装,以期发现样品中较低丰度物种的信息,筛选拼接长度在500 bp以上的Scaftigs进行后续分析。采用Mmseqs软件进行基因聚类及去冗余,非冗余基因集的Unigenes平均长度为500.13 bp,平均GC含量为60.98%。使用Diamond (Version 0.9.30)软件将非冗余的Unigenes序列与NCBI-NR数据库比对进行物种的同源性BLAST比对 (设定阈值为e-value≤0.0001),共有11688293个冗余基因集的Unigenes被注释。宏基因组测序工作委托广东美格基因科技有限公司完成。原始序列数据已上传至NCBI数据库,注册编号ID:PRJNA1166634。
1.5 土壤宏病毒组测序
称取3.0 g储存于−80℃的新鲜土壤样品,进行组织破碎和匀浆处理,低速离心去除组织和细胞的碎片,运用过滤、超速离心或沉淀等多种病毒学的分离方法对溶液中的病毒样颗粒(VLP)进行纯化和浓缩,然后采用病毒DNA (dsDNA、ssDNA)和RNA (ssRNA、dsRNA)共提取的方法同时提取核酸[21]。检测合格的DNA或经扩增的DNA样品采用超声破碎仪进行随机打断,将打断后得到的短片段DNA用于测序文库构建,质检合格的文库将采用Illumina平台进行测序,测序读长PE150。高通量测序得到的图像经Base Calling转化为原始测序序列(Reads),使用Fastp软件对原始数据(Raw Data)进行一系列处理,得到高质量的clean reads。使用Prokka (v1.13)软件对病毒contigs进行基因预测,过滤基因核酸长度小于200 bp的contigs序列。利用预测出来的基因蛋白序列与UniProtKB/Swiss-Prot/KEGG数据库的病毒序列 (ViralZone, reviewed proteins,https://viralzone.expasy.org/) 比对,获得功能注释信息。得到上述潜在的病毒序列集后,使用PhaGCN2软件对序列进行病毒分类学注释。将reads比对到鉴定后的病毒contigs上,计算每个contig的丰度 [RPKM值=contig reads/(total mapped reads×contig length)]。鉴定得到的病毒序列,采用CheckV软件中封装的Prodigal包对组装的contigs进行基因注释,并将这些片段注释的基因与美格基因自建的病毒数据库[V4版,主要包含参考数据库:KEGG,VOGDB,PfamA,PfamB,IMG/VR,TIGRFAM,RVDB,NCBI GenBank (viral),GVD,GPD,MGV,CHVD,Riboviria等]进行比对,鉴定微生物和病毒的基因。使用CHERRY软件 (参数:-len1000-model pretrain-topk1) 和PHP软件 (参数:-nhostKmer_60105_kmer4) 根据病毒基因组的序列信息,分别对过滤后的病毒序列进行宿主预测分析,并对结果做阈值筛选,得到最终结果。采用DRAM软件对Virsorter2预测的噬菌体序列按基因区域进行拆分,根据其对这些基因的注释,将噬菌体注释为辅助代谢基因的序列进行划分和归类。宏病毒组测序工作委托广东美格基因科技有限公司完成。原始序列数据已上传至NCBI数据库,注册编号ID:PRJNA1166100。
1.6 数据处理
采用SPSS Statistics 23 (IBM,USA)软件对稻田土壤病毒与细菌群落的相对丰度、氮循环基因丰度等数据进行Shapiro-Wilk正态性检验,符合正态分布的数据采用双因素方差分析(two-way ANOVA)计算其差异显著性。采用origin 2022 (OriginLab,Northampton,MA,USA)软件绘制土壤病毒与细菌群落物种丰度百分比柱状堆叠图、病毒与细菌网络图和基因丰度柱状图。采用R语言中的“plspm”包构建偏最小二乘路径模型。
2. 结果与分析
2.1 土壤病毒类群丰度
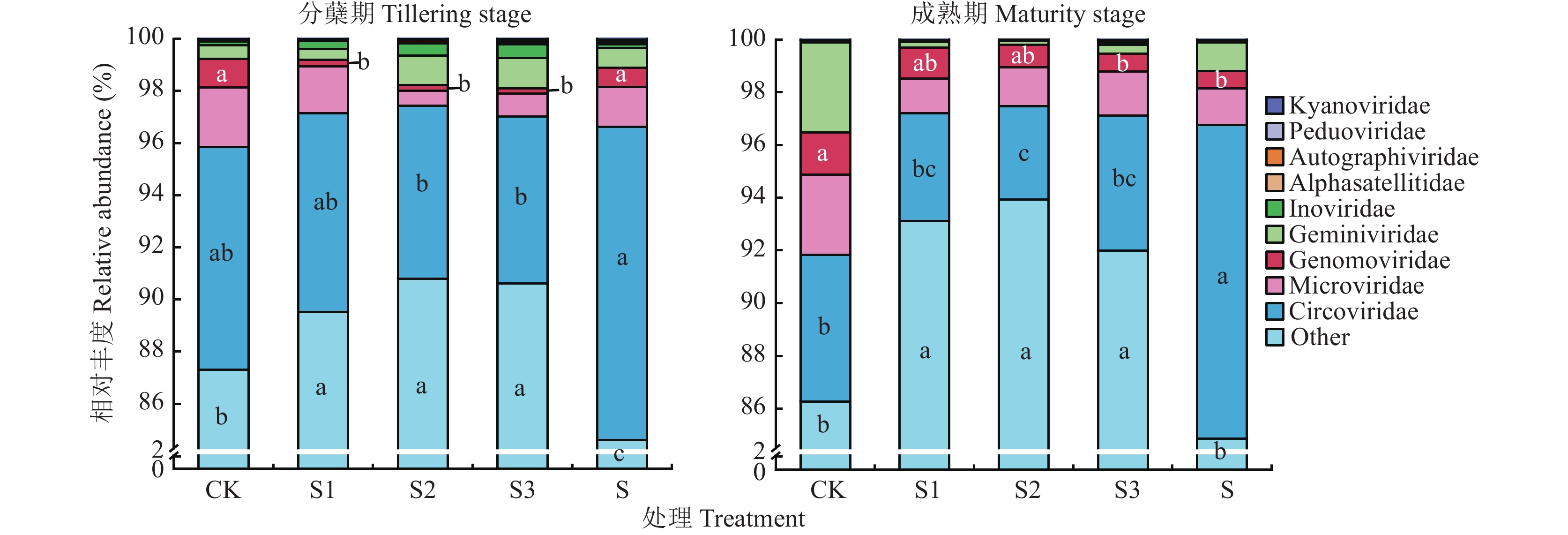
图1显示,供试稻田土壤病毒科丰度最高的是圆环病毒科(Circoviridae),相对丰度3.54%~12.00%,其次是微小噬菌体科(Microviridae)、类双生病毒科(Genomoviridae)和双生病毒科(Geminiviridae),分别占0.56%~3.04%、0.43%~1.61%和0.20%~3.41%,其余5科病毒丰度合计占0.05%~0.73%,但是,84.62%~93.92%病毒科未被分类或未知。其中,S处理的圆环病毒科(Circoviridae)的丰度显著增加(P<0.05),在水稻分蘖期和成熟期分别较CK处理增加了3.47%和6.33%,而其他处理则有所降低。水稻分蘖期S1、S2和S3处理的类双生病毒科(Genomoviridae)的丰度分别较CK显著降低了0.86%、0.86%、0.89%。成熟期S3和S处理双生病毒科(Geminiviridae)的丰度分别较CK显著降低了0.89%和0.35%。值得注意的是,无论是水稻分蘖期还是成熟期,未被分类或未知病毒科的相对丰度在S1、S2和S3处理中显著增加(P<0.05)。水稻分蘖期与成熟期相比较,各处理稻田土壤病毒群落的相对丰度变化趋势基本相同。综上所述,稻草还田量对稻田土壤病毒丰度产生了显著影响,而水稻不同生育期对土壤病毒类群无显著影响。
![]() 图 1 不同稻草还田量处理晚稻分蘖期和成熟期土壤前10位病毒科的相对丰度注:CK为稻草不还田,S为全部稻草还田(单季稻草还田量6400 kg/hm2),S1、S2、S3处理的稻草还田量为全部稻草还田的1.1、1.2、1.3倍(单季稻草还田量分别为7075、7750、8425 kg/hm2)。柱中不同小写字母表示处理间差异显著(P<0.05).Figure 1. The relative abundance of top 10 soil virus families in the paddy soil at tillering and maturity stages of late rice receiving different rice straw incorporation ratesNote: CK is the straw not returned, S is the whole straw returned (the amount of straw returned to the field in a single season is 6400 kg/hm2), and the amount of straw returned to the field in S1, S2 and S3 is 1.1, 1.2 and 1.3 times that of all the straw returned (the amount of straw returned to the field in a single season are 7075, 7750 and 8425 kg/hm2, respectively). Different lowercase letters in the bar indicate significant difference among treatments (P<0.05).
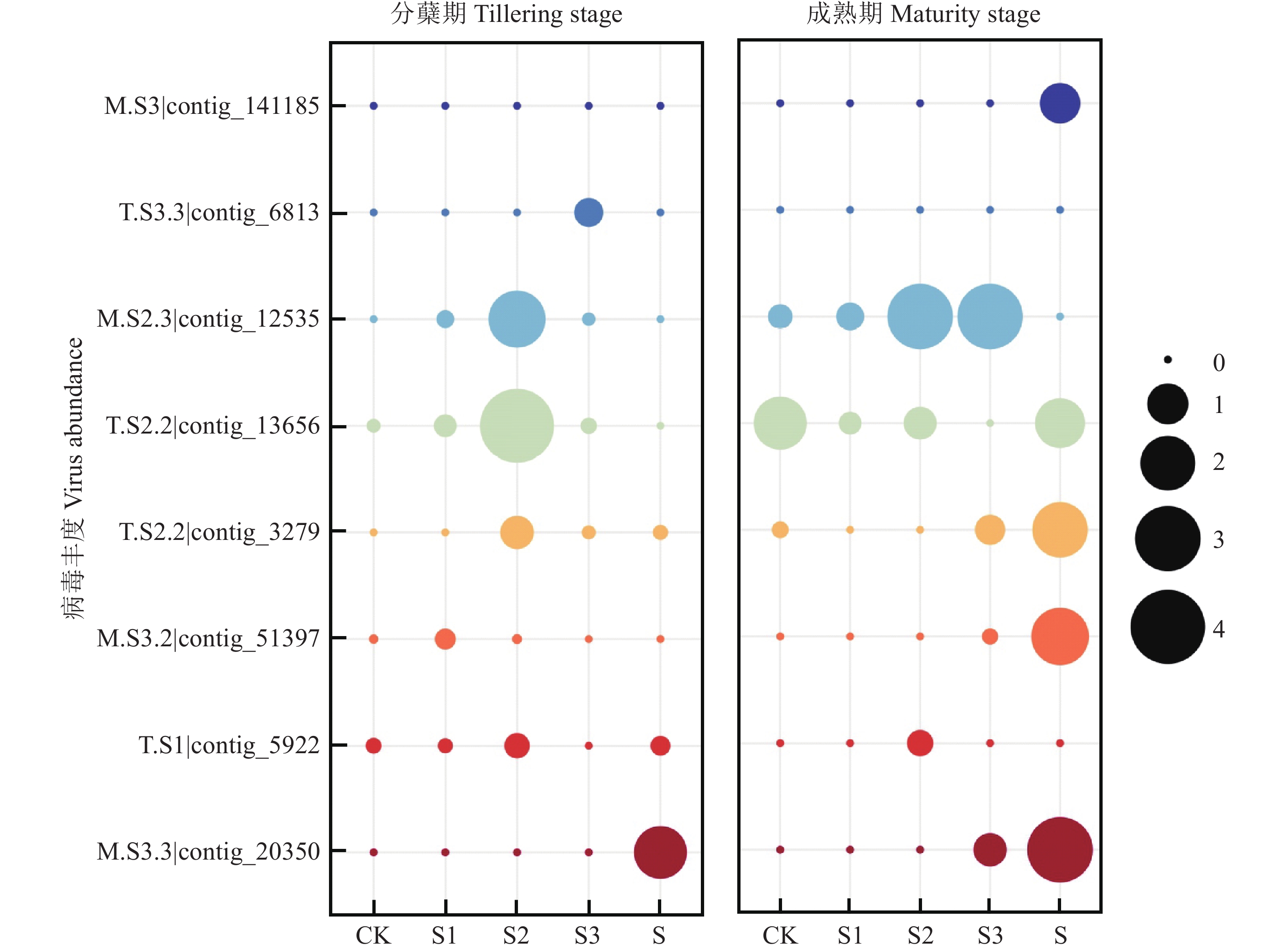
图 1 不同稻草还田量处理晚稻分蘖期和成熟期土壤前10位病毒科的相对丰度注:CK为稻草不还田,S为全部稻草还田(单季稻草还田量6400 kg/hm2),S1、S2、S3处理的稻草还田量为全部稻草还田的1.1、1.2、1.3倍(单季稻草还田量分别为7075、7750、8425 kg/hm2)。柱中不同小写字母表示处理间差异显著(P<0.05).Figure 1. The relative abundance of top 10 soil virus families in the paddy soil at tillering and maturity stages of late rice receiving different rice straw incorporation ratesNote: CK is the straw not returned, S is the whole straw returned (the amount of straw returned to the field in a single season is 6400 kg/hm2), and the amount of straw returned to the field in S1, S2 and S3 is 1.1, 1.2 and 1.3 times that of all the straw returned (the amount of straw returned to the field in a single season are 7075, 7750 and 8425 kg/hm2, respectively). Different lowercase letters in the bar indicate significant difference among treatments (P<0.05).在对稻田土壤中携带有机氮转化基因的病毒contigs进行进一步统计后,我们发现共有8条病毒contigs,且在水稻分蘖期和成熟期,这些携带有机氮转化基因的病毒contigs在稻草还田处理中的相对丰度几乎全部高于CK处理,病毒序列M.S3|contig_141185和T.S3.3|contig_6813分别在水稻成熟期和分蘖期表现出独有性(图2)。除序列T.S3.3|contig_6813以外,其他contigs均在纲水平上被归类为有尾噬菌体纲(Caudoviricetes),仅有M.S3.2|contig_51397被注释到科分类水平,为P2噬菌体科(Peduoviridae)。水稻分蘖期,病毒序列M.S3.2|contig_51397和M.S3.3|contig_20350分别在S1处理和S处理中相对丰度最高,而其他病毒序列均在S2处理中相对丰度最高。水稻成熟期,病毒序列M.S3.2|contig_51397和M.S3.3|contig_20350均在S处理中相对丰度最高。
![]() 图 2 稻草还田下土壤氮循环相关的病毒contigs丰度Bubble图注:CK为稻草不还田,S为全部稻草还田(单季稻草还田量6400 kg/hm2),S1、S2、S3处理的稻草还田量为全部稻草还田的1.1、1.2、1.3倍(单季稻草还田量分别为7075、7750、8425 kg/hm2)。图中实心圆大小表示稻草还田下土壤氮循环相关病毒contigs的丰度(基于RPKM值)大小,不同颜色代表不同病毒contig。Figure 2. Bubble diagram of virus contigs abundance associated with soil nitrogen cycling under straw returnNote: CK is the straw not returned, S is all the straw returned (the amount of straw returned to the field in a single season is 6400 kg/hm2), and the amount of straw returned to the field in S1, S2 and S3 is 1.1, 1.2 and 1.3 times that of all straws returned (the amount of straw returned to the field in a single season are 7075, 7750 and 8425 kg/hm2, respectively). The size of the circle in the figure represents the abundance of virus contigs related to soil nitrogen cycling under rice straw return (based on RPKM values), and different colors represent different virus contigs.
图 2 稻草还田下土壤氮循环相关的病毒contigs丰度Bubble图注:CK为稻草不还田,S为全部稻草还田(单季稻草还田量6400 kg/hm2),S1、S2、S3处理的稻草还田量为全部稻草还田的1.1、1.2、1.3倍(单季稻草还田量分别为7075、7750、8425 kg/hm2)。图中实心圆大小表示稻草还田下土壤氮循环相关病毒contigs的丰度(基于RPKM值)大小,不同颜色代表不同病毒contig。Figure 2. Bubble diagram of virus contigs abundance associated with soil nitrogen cycling under straw returnNote: CK is the straw not returned, S is all the straw returned (the amount of straw returned to the field in a single season is 6400 kg/hm2), and the amount of straw returned to the field in S1, S2 and S3 is 1.1, 1.2 and 1.3 times that of all straws returned (the amount of straw returned to the field in a single season are 7075, 7750 and 8425 kg/hm2, respectively). The size of the circle in the figure represents the abundance of virus contigs related to soil nitrogen cycling under rice straw return (based on RPKM values), and different colors represent different virus contigs.2.2 土壤细菌类群丰度
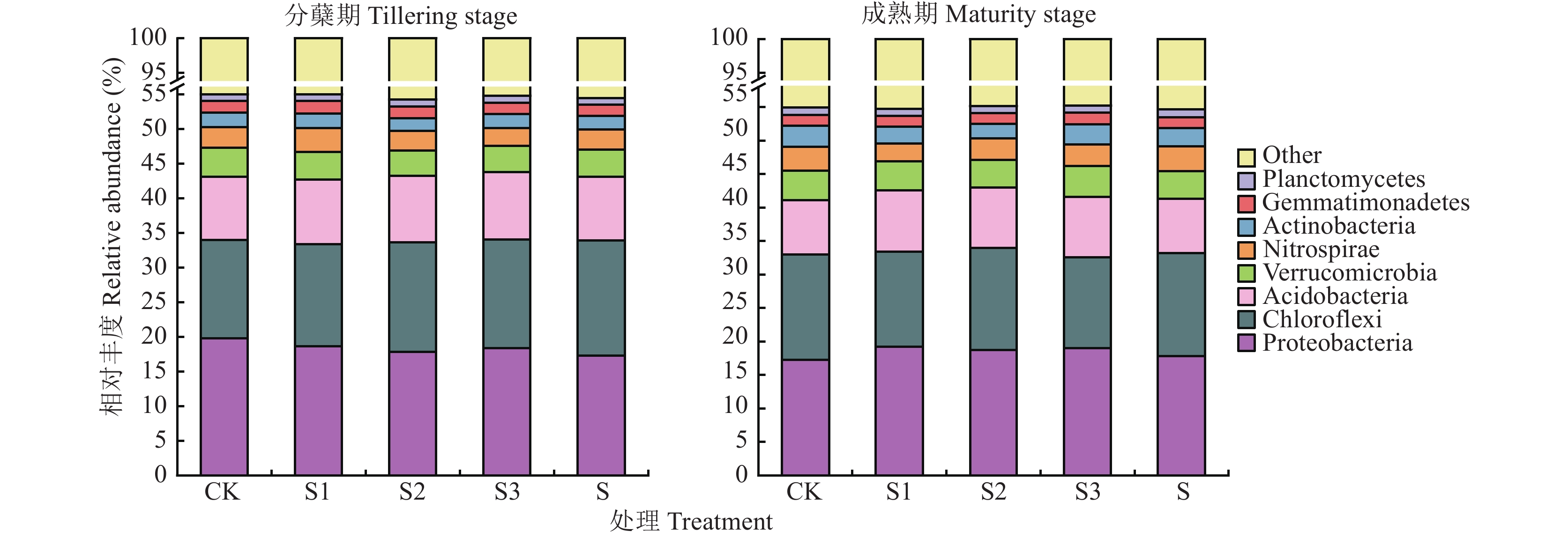
由图3可知,供试的稻田土壤优势细菌门丰度最高的是变形菌门(Proteobacteria,17.3%~19.8%),其次是绿弯菌门(Chloroflexi,14.2%~16.7%)、酸杆菌门(Acidobacteria,8.1%~9.7%)和疣微菌门(Verrucomicrobia,3.7%~4.7%)。不同稻草还田量处理之间细菌优势群落的相对丰度没有显著差异(P>0.05)。此外,对比水稻分蘖期与成熟期细菌群落的相对丰度,其变化趋势基本相同。综上所述,稻草还田量和水稻生育期对土壤优势细菌门均无显著影响。
![]() 图 3 不同处理下水稻分蘖期与成熟期土壤细菌门相对丰度柱状堆叠图注:CK为稻草不还田,S为全部稻草还田(单季稻草还田量6400 kg/hm2),S1、S2、S3处理的稻草还田量为全部稻草还田的1.1、1.2、1.3倍(单季稻草还田量分别为7075、7750、8425 kg/hm2)。Figure 3. Histogram of relative abundance of soil bacteria at tillering and maturity stages under different treatmentsNote: CK is the straw not returned, S is all the straw returned (the amount of straw returned to the field in a single season is 6400 kg/hm2), and the amount of straw returned to the field in S1, S2 and S3 is 1.1, 1.2 and 1.3 times that of all straws returned (the amount of straw returned to the field in a single season are 7075, 7750 and 8425 kg/hm2, respectively).
图 3 不同处理下水稻分蘖期与成熟期土壤细菌门相对丰度柱状堆叠图注:CK为稻草不还田,S为全部稻草还田(单季稻草还田量6400 kg/hm2),S1、S2、S3处理的稻草还田量为全部稻草还田的1.1、1.2、1.3倍(单季稻草还田量分别为7075、7750、8425 kg/hm2)。Figure 3. Histogram of relative abundance of soil bacteria at tillering and maturity stages under different treatmentsNote: CK is the straw not returned, S is all the straw returned (the amount of straw returned to the field in a single season is 6400 kg/hm2), and the amount of straw returned to the field in S1, S2 and S3 is 1.1, 1.2 and 1.3 times that of all straws returned (the amount of straw returned to the field in a single season are 7075, 7750 and 8425 kg/hm2, respectively).2.3 携带有机氮转化基因的土壤病毒与细菌互作
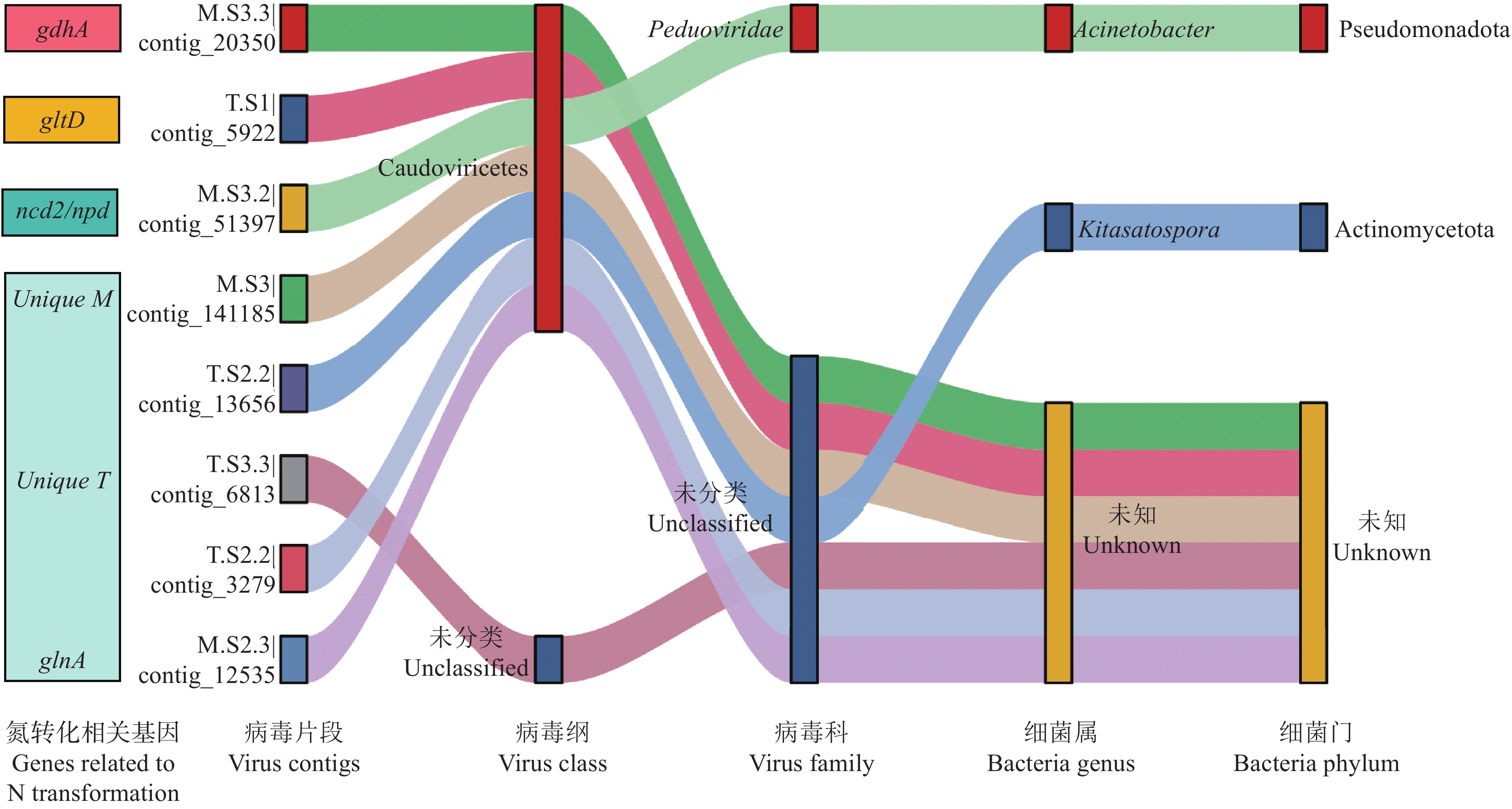
通过CHERRY和PHP软件对稻田土壤病毒进行宿主预测,稻田土壤病毒预测到的宿主跨越71个细菌门,其中假单胞菌门(Pseudomonadota)是被预测最多的细菌门。由图4可以看出,共有4个与有机氮转化相关的基因被病毒和细菌共同拥有,分别是gdhA、gltD、ncd2/npd和glnA。对携带这些有机氮转化基因的病毒contigs进一步预测宿主,结果表明,有2条病毒contigs预测到宿主。其中,携带ncd2/npd基因的病毒序列M.S3.2|contig_51397在科水平上被鉴定为P2噬菌体科(Peduoviridae),预测的宿主为不动杆菌属(Acinetobacter),属于假单胞菌门(Pseudomonadota)。而携带glnA基因的病毒序列T.S2.2|contig_13656在科水平上未被分类,其宿主为北里孢菌属(Kitasatospora),属于放线菌门(Actinomycetota)。
2.4 土壤病毒与细菌携带的有机氮转化基因与土壤氮素指标的关系及病毒AMGs
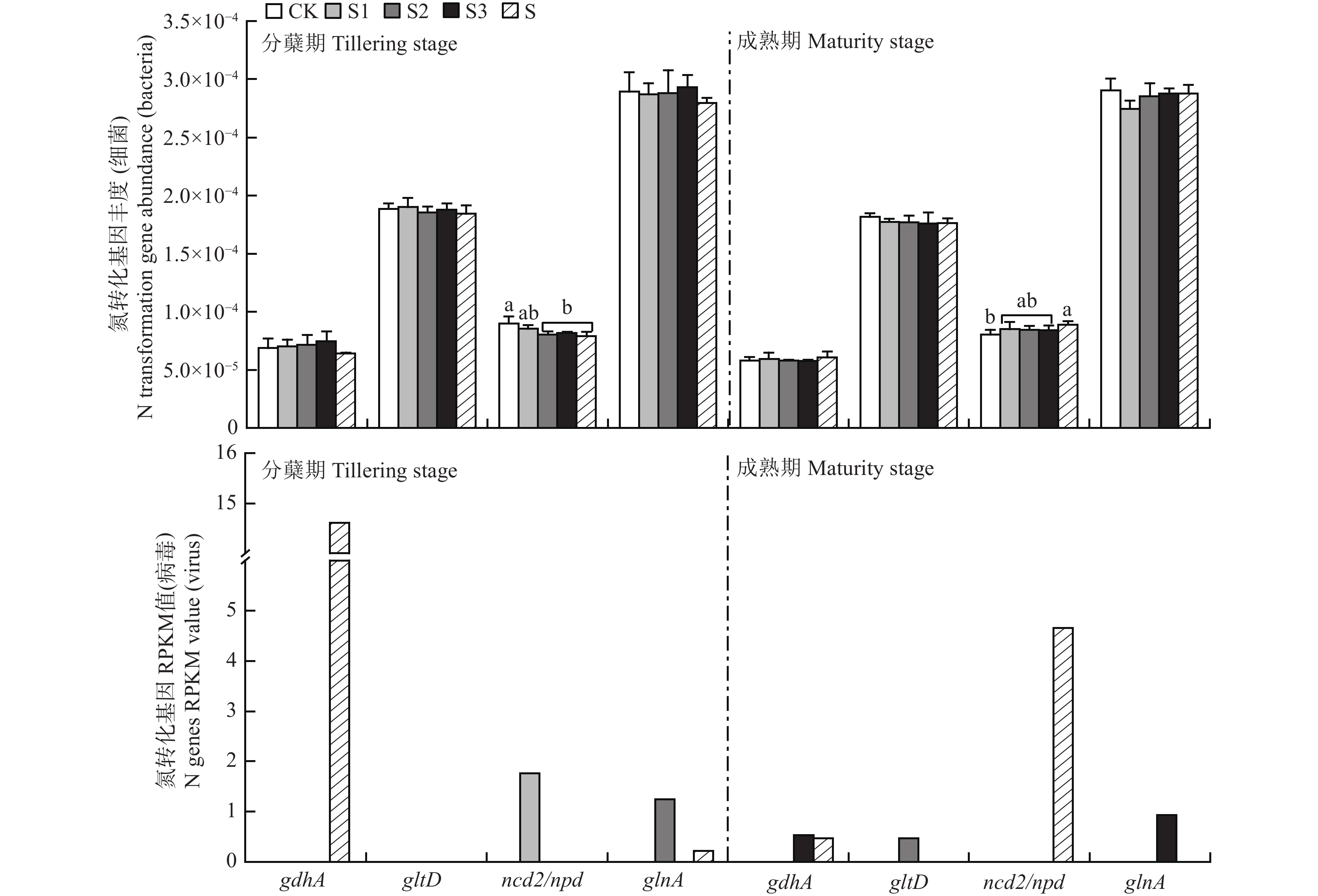
图5显示,在水稻分蘖期和成熟期,晚稻田土壤中病毒携带的有机氮转化基因在稻草不还田(CK)处理中未被检测到。而在稻草还田的处理中,仅有少数处理检测到了个别有机氮转化相关基因。水稻分蘖期,基因glnA在S2处理中丰度最高;基因ncd2/npd在S1处理中丰度最高;基因gltD在所有处理中丰度均为零;基因gdhA在S处理中丰度最高。水稻成熟期,基因glnA在S3处理中丰度最高;基因ncd2/npd在S处理中丰度最高;基因gltD在S2处理中丰度最高;基因gdhA在S3处理中丰度最高。稻草还田量显著影响了细菌携带的有机氮转化基因ncd2/npd的丰度(P<0.05),而对基因gdhA、gltD和glnA的丰度影响不显著。具体而言,在水稻分蘖期,S2、S3和S处理中基因ncd2/npd的丰度显著低于CK处理(P<0.05)。水稻成熟期,S处理中基因ncd2/npd的丰度显著高于CK处理(P<0.05)。
![]() 图 5 水稻分蘖期与成熟期土壤细菌和病毒所含的有机氮转化基因丰度注:CK为稻草不还田,S为全部稻草还田(单季稻草还田量6400 kg/hm2),S1、S2、S3处理的稻草还田量为全部稻草还田的1.1、1.2、1.3倍(单季稻草还田量分别为7075、7750、8425 kg/hm2)。Figure 5. Abundance of organic nitrogen transformation genes contained in soil bacteria and viruses at tillering and maturity stages of riceNote: CK is the straw not returned, S is all the straw returned (the amount of straw returned to the field in a single season is 6400 kg/hm2), and the amount of straw returned to the field in S1, S2 and S3 is 1.1, 1.2 and 1.3 times that of all straws returned (the amount of straw returned to the field in a single season are 7075 kg/hm2, 7750 kg/hm2, 8425 kg/hm2, respectively).
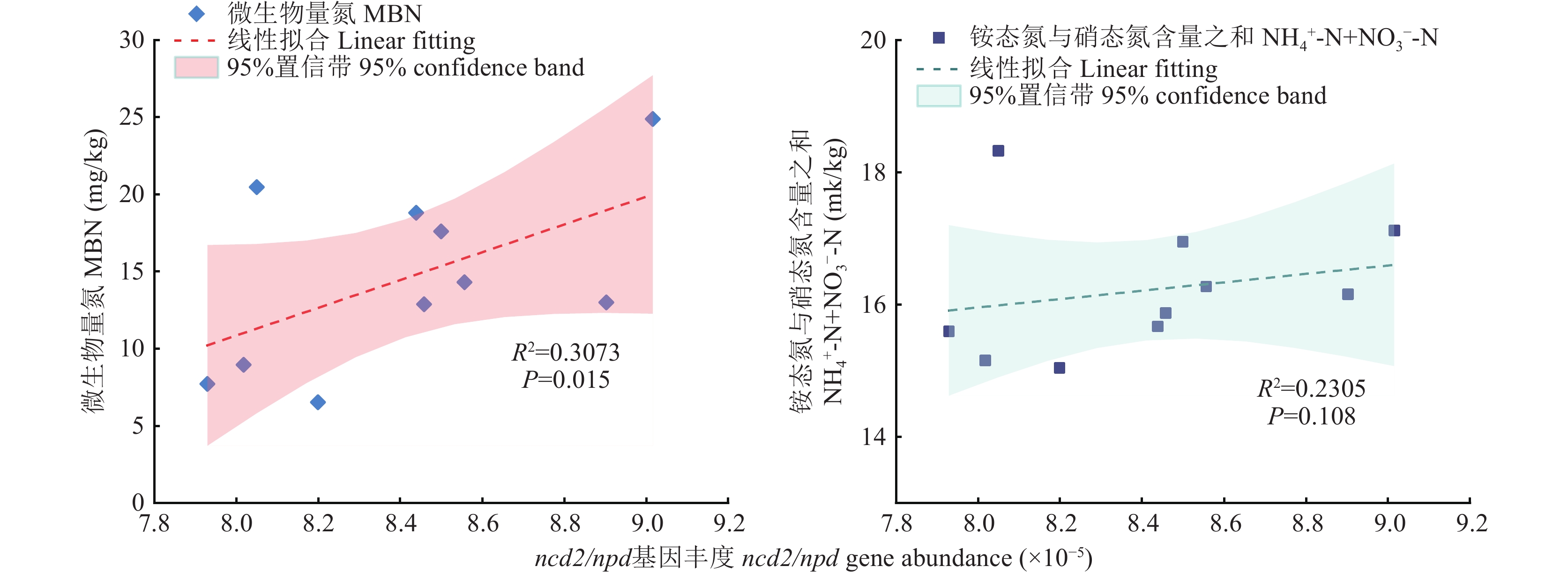
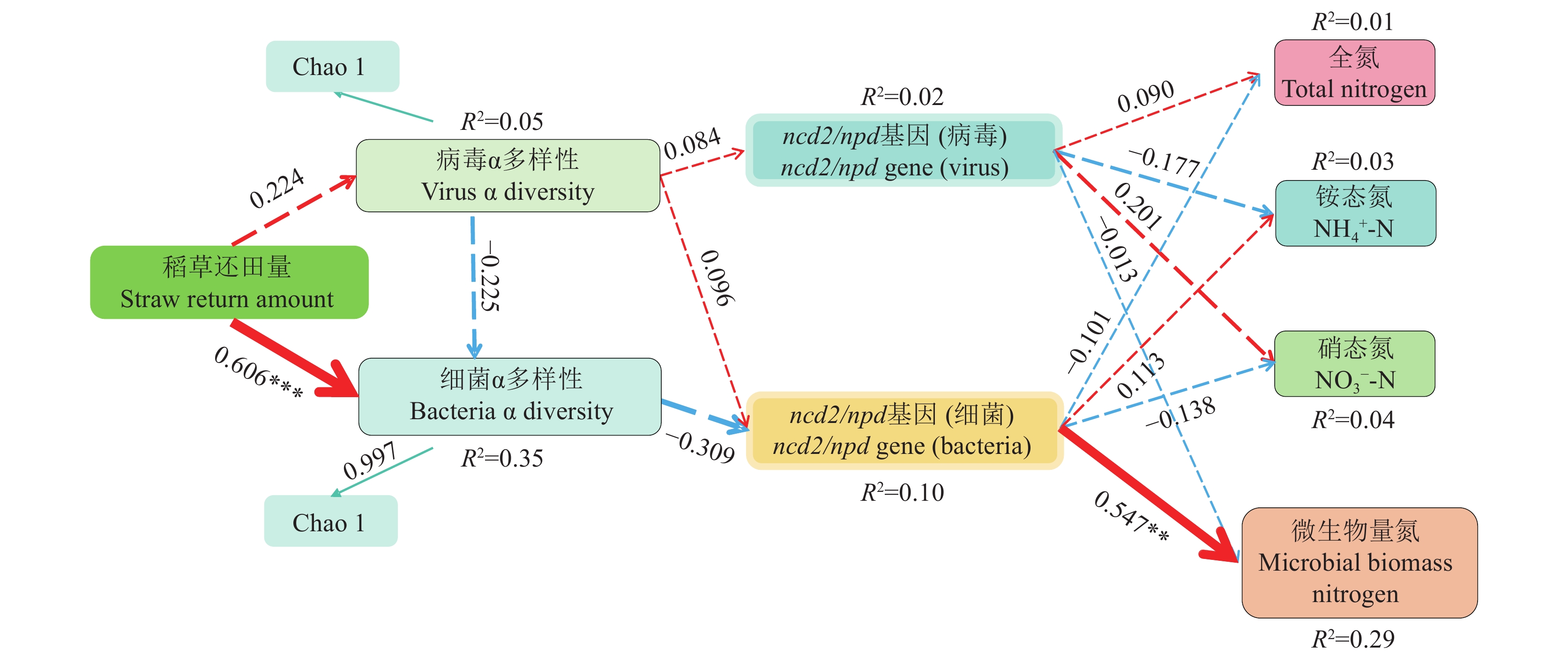
图 5 水稻分蘖期与成熟期土壤细菌和病毒所含的有机氮转化基因丰度注:CK为稻草不还田,S为全部稻草还田(单季稻草还田量6400 kg/hm2),S1、S2、S3处理的稻草还田量为全部稻草还田的1.1、1.2、1.3倍(单季稻草还田量分别为7075、7750、8425 kg/hm2)。Figure 5. Abundance of organic nitrogen transformation genes contained in soil bacteria and viruses at tillering and maturity stages of riceNote: CK is the straw not returned, S is all the straw returned (the amount of straw returned to the field in a single season is 6400 kg/hm2), and the amount of straw returned to the field in S1, S2 and S3 is 1.1, 1.2 and 1.3 times that of all straws returned (the amount of straw returned to the field in a single season are 7075 kg/hm2, 7750 kg/hm2, 8425 kg/hm2, respectively).进一步分析有机氮转化基因ncd2/npd的丰度与土壤氮素指标之间的关系。图6表明,晚稻田土壤细菌携带的有机氮转化基因ncd2/npd的丰度与微生物量氮含量呈显著正相关关系(P=0.015,n=10),与铵态氮、硝态氮总量无显著相关关系(P=0.108,n=10)。为了探究稻草还田量与稻田土壤病毒、细菌群落多样性、ncd2/npd基因丰度和土壤氮素指标之间的关系,构建偏最小二乘路径模型用于解释结果。由图7可知,稻草还田量通过影响土壤病毒和细菌的α多样性,进而间接影响细菌携带的ncd2/npd基因的相对丰度,最终对有机氮转化过程产生重要影响。具体表现为,稻草还田显著影响了稻田土壤细菌的α多样性(R2=0.35,P<0.0007);细菌携带的有机氮转化基因ncd2/npd显著影响了稻田土壤微生物量氮的含量(R2=0.29,P<0.0039)。
![]() 图 7 偏最小二乘路径模型注:箭头旁数字表示路径系数;红色与蓝色箭头分别表示变量之间正负相关关系。模型可靠性的拟合优度为0.34。**—P<0.01; ***—P<0.001。Figure 7. Partial least squares path modelNote: The numbers next to the arrows indicate the path factor; the red and blue arrows indicate positive and negative correlations between variables, respectively. The goodness of fit of the model is 0.34. **—P<0.01; ***—P<0.001.
图 7 偏最小二乘路径模型注:箭头旁数字表示路径系数;红色与蓝色箭头分别表示变量之间正负相关关系。模型可靠性的拟合优度为0.34。**—P<0.01; ***—P<0.001。Figure 7. Partial least squares path modelNote: The numbers next to the arrows indicate the path factor; the red and blue arrows indicate positive and negative correlations between variables, respectively. The goodness of fit of the model is 0.34. **—P<0.01; ***—P<0.001.基于DRAM软件对宏病毒组中AMGs进行鉴定,并结合KEGG数据库进行注释。共鉴定到28条氮循环相关的AMGs (表2)。鉴定到的AMGs被注释为两个KEGG有机氮基因DNMT1/dcm和ilvB/ilvG/ilvI,在有机氮转化过程中扮演着重要的角色。基因DNMT1/dcm和ilvB/ilvG/ilvI均参与有机氮转化的氨基酸代谢过程,前者编码DNA(胞嘧啶-5-)-甲基转移酶,参与半胱氨酸和蛋氨酸代谢过程,是氨基酸代谢过程的重要步骤。后者编码乙酰乳酸合酶Ⅰ/Ⅱ/Ⅲ大亚基参与多个代谢过程,例如:缬氨酸、亮氨酸和异亮氨酸的生物合成,丁酸酯代谢,C5-支链二元酸代谢,泛酸酯和辅酶A的生物合成和氨基酸的生物合成等,其中缬氨酸、亮氨酸和异亮氨酸的生物合成是氨基酸代谢过程的另一重要步骤。
表 2 病毒辅助代谢基因Table 2. Viral auxiliary metabolism genesKO编号 KO number 基因名称 Gene name 基因 Gene 基因详情 Gene description K00558 DNMT1 / dcm M.CK3_contig_11608-cat_2_4
M.S1.1_contig_7575-cat_1_14
M.S1.1_contig_8731-cat_2_2
M.S1.2_contig_12267-cat_2_8
M.S1.2_contig_9944-cat_1_46
M.S2.1_contig_1140-cat_2_16
M.S2.2_contig_897-cat_2_2
M.S3.1_contig_17094-cat_1_43
M.S3.2_contig_28456-cat_2_10
M.S3.3_contig_151241-cat_2_10
M.S3.3_contig_173896-cat_1_4
M.S2_contig_35584-cat_2_18
M.S3_contig_14916-cat_2_3
M.S3_contig_19124-cat_1_13
M.S3_contig_43849-cat_2_9
T.CK2_contig_11671-cat_2_13
T.CK3_contig_9479-cat_1_38
T.S1.1_contig_6392-cat_2_4
T.S1.1_contig_8281-cat_1_92
T.S1.2_contig_2737-cat_1_66
T.S1.2_contig_5126-cat_1_23
T.S2.3_contig_4688-cat_2_10
T.S3.2_contig_16590-cat_1_50
T.S3.3_contig_12427-cat_1_15
T.S3.3_contig_12963-cat_1_33
T.S3.3_contig_8555-cat_2_2DNA(胞嘧啶-5-)-甲基转移酶
DNA (cytosine-5-)-methyltransferaseK01652 ilvB / ilvG / ilvI M.S3.3_contig_182589-cat_2_4
M.S3.3_contig_182589-cat_2_4乙酰乳酸合酶Ⅰ/Ⅱ/Ⅲ大亚基
Acetolactate synthase Ⅰ/Ⅱ/Ⅲ large subunit3. 讨论
3.1 土壤病毒群落结构特征
病毒在土壤环境中数量丰富且种类多样,在稻田土壤生态系统中具有至关重要的作用。土壤病毒群落组成受到较多因素的影响,不同地区、不同土壤类型和不同耕作方式下稻田土壤病毒群落的组成不同[22−23]。无论是水环境还是陆地环境[24],甚至在局部尺度上[25],病毒群落在不同环境中的组成均具有很大差异。本研究中稻田土壤病毒群落组成主要以圆环病毒科(Circoviridae)、微小噬菌体科(Microviridae)和双生病毒科(Geminiviridae)占据优势地位。土壤病毒群落组成会因稻田的地理位置和土壤特性等多种因素影响而不同,刘泽锟等[26]在一项关于全球稻田土壤病毒多样性的研究中发现,稻田土壤病毒大多来自于有尾噬菌体纲(Caudoviricetes)。王亚娇[27]在关于中国三大稻区土壤病毒群落组成的研究中发现,稻田土壤病毒优势群落主要属于长尾噬菌体科(Siphoviridae)、藻类去氧核糖核酸病毒科(Phycodnaviridae)、肌尾噬菌体科(Myoviridae)、疱疹病毒科(Herpesviridae)、短尾噬菌体科(Podoviridae),与本研究呈现的结果明显不同。Han等[28]在关于中国6种典型农业土壤中病毒群落的探究中发现,在吉林的玉米土壤、湖南和江西的水稻土壤中均以圆环病毒科(Circoviridae)为主,山东的玉米土壤、吉林和江苏的水稻土壤中均以微小噬菌体科(Microviridae)丰度占比最高。Gao等[29]研究表明,不同砷(As)污染水平下稻田土壤病毒群落组成主要以长尾噬菌体科(Siphoviridae)、短尾噬菌体科(Podoviridae)、肌尾噬菌体科(Myoviridae)和微小噬菌体科(Microviridae)为主。
3.2 土壤病毒与细菌宿主的联系
病毒自身不具备独立生存的能力,必须依靠寄生宿主来完成生命活动。病毒与宿主之间紧密联系,环境因素的改变可能导致病毒与宿主的相互作用更加复杂。本研究中主要针对氮循环相关的土壤病毒预测宿主,这些宿主主要来自假单胞菌门(Pseudomonadota)、芽孢杆菌门(Bacillota)、放线菌门(Actinomycetota)和拟杆菌门(Bacteroidota)。刘泽锟等[26]研究表明,稻田土壤病毒预测出的宿主来自于11个细菌门,其中变形菌门(Proteobacteria)、芽孢杆菌门(Bacillota)数量最多。Wu等[7]研究表明,珠穆朗玛峰土壤病毒预测的宿主主要来源于8个细菌门,其中变形菌门(Proteobacteria)、芽孢杆菌门(Bacillota)、放线菌门(Actinomycetota)是可能被大多数病毒感染的细菌门。Yu等[30]研究表明,在深海海山沉积物病毒预测的宿主中,变形菌门(Proteobacteria)被预测最多,然后是放线菌门(Actinomycetota)、拟杆菌门(Bacteroidota)、浮霉菌门(Planctomycetes)和Gemmatimonadota门。Emerson等[12]研究表明,在粪肥施用的农业土壤中,预测到的病毒宿主被归类于变形菌门(Proteobacteria)、放线菌门(Actinomycetota)和拟杆菌门(Bacteroidota)等10个细菌门。病毒感染宿主具有特异性,病毒感染造成的选择压力可能引起宿主进化产生抗性,产生抗性的同时可能伴随着自身生长速率降低等后果[31]。在本研究中,稻草还田增加了变形菌门(Proteobacteria)的相对丰度。假单胞菌门(Pseudomonadota)是由变形菌门(Proteobacteria)去除ε-变形菌纲(Epsilonproteobacteria)、δ-变形菌纲(Deltaproteobacteria)和寡弯菌纲(Chloroflexi)后的改称。符合搭乘胜利者“piggyback-the-winner”假说,该假说认为当宿主密度较高时,温和性生活方式更受青睐,病毒通过溶原而不是裂解宿主来利用它们[1]。携带基因ncd2/npd的P2噬菌体科(Peduoviridae),可能通过感染或裂解宿主假单胞菌门(Pseudomonadota)的不动杆菌属(Acinetobacter)将硝基烷烃转化为亚硝酸盐。并且假单胞菌门(Pseudomonadota)作为稻田土壤中被预测到最多的细菌门,在土壤有机氮转化过程中的作用不可被忽视。变形菌门(Proteobacteria)作为深海沉积物中病毒最易感染的第一大宿主,参与硝酸盐还原成氨过程和几丁质降解过程,为其他微生物提供生长所需的碳源和氮源,放线菌门(Actinomycetota)作为深海沉积物中病毒最易感染的第二大宿主,拥有大量的碳水化合物降解基因,参与几丁质、半纤维素和淀粉降解等过程,表明深海沉积物中病毒与变形菌门(Proteobacteria)、放线菌门(Actinomycetota)相互作用,在碳、氮等元素循环过程中发挥关键作用[30−33]。
3.3 土壤病毒与细菌携带的有机氮转化基因与土壤氮素指标的关系及病毒AMGs
土壤病毒基因注释分布在各种功能中,本研究针对KEGG途径筛选氮循环相关的病毒基因,以了解病毒与宿主在元素循环过程中的相互作用以及基因之间的联系。有机氮转化过程相关基因glnA、gltD、gdhA和ncd2/npd均在稻草还田处理中检测到丰度,而在稻草不还田处理中为零,说明稻草还田可以促进稻田土壤病毒携带有机氮转化基因,从而参与土壤氮循环过程。Gazitúa等[34]和Roux等[35]在不同的宏病毒组样本中检测到数量较少的氮循环基因,尽管土壤病毒携带的这些有机氮转化基因并非高置信度,但在土壤氮循环过程中仍具有一定潜力。王美琦等[36]研究表明,稻草还田通过增加土壤中碳含量,导致土壤碳氮比失衡,进而影响土壤微生物对氮素循环相关微生物及基因的调控。因此,即使是稻草还田量的微小差异,也可能通过改变土壤微生物环境,间接影响病毒携带的有机氮转化基因的丰度和活性。基因gdhA和gltD参与L-谷氨酸的脱氨过程,产生2-氧代戊二酸和NH3,此外基因gltD还参与L-谷氨酸的转氨过程,该过程最终产物为L-谷氨酰胺和2-氧代戊二酸。Emerson等[12]研究发现,病毒可能感染厌氧菌科(Anaerolineae)和浮游菌科(Planctomycetaceae)细菌,携带基因gdh参与谷氨酸和谷氨酰胺代谢,并且催化有机氮降解。基因glnA主要参与NH3和L-谷氨酸转化为L-谷氨酰胺的过程。基因ncd2/npd主要参与硝基烷烃转化为亚硝酸盐的过程,亚硝酸盐通过氮循环过程的其他作用进一步转化为被作物可直接吸收利用的氮源,从而参与土壤氮循环过程。基因ncd2/npd是最常见的氮循环相关的AMGs。晚稻田土壤细菌携带的有机氮转化基因ncd2/npd的丰度与微生物量氮含量呈显著正相关。在水稻分蘖期,稻草还田通过调节病毒与细菌的α多样性,降低了细菌携带的有机氮转化基因ncd2/npd的丰度。而稻草还田处理中微生物量氮的含量较低,这可能是稻草还田处理中部分有机氮被转化为无机氮,以供应作物在生长盛期的氮素需求。而水稻成熟期,稻草还田处理中基因ncd2/npd的丰度有所上升,土壤中微生物量氮的含量较分蘖期高,这可能是因为水稻成熟期土壤水分、温度变化以及水稻所需氮素养分较少等因素,使得有机氮向无机氮转化的速率有所减缓。
病毒表达AMGs参与元素生物地球化学循环过程已被广泛认可。以往的研究已经鉴定了许多元素循环相关的AMGs,如参与土壤碳循环的CAZyme和糖苷水解酶基因[10−11],参与磷循环的phoB、dUTPase、MazG、phoH、Thy1、RNR基因和参与氮循环的gdh和ncd2/npd基因[13−14, 37]。这些AMGs在土壤生物地球化学循环中发挥着重要作用。氨基酸是构成土壤有机氮的重要组成部分,对于土壤氮循环过程至关重要。稻草还田促进了稻田土壤病毒表达更多的辅助代谢基因DNMT1/dcm和ilvB/ilvG/ilvI,在氨基酸合成过程发挥关键作用,进而影响稻田土壤氮循环过程。
4. 结论
稻草还田显著影响了稻田土壤病毒类群的相对丰度,促进了病毒携带有机氮转化基因表达更多的AMGs,在土壤氮循环过程中发挥重要作用。ncd2/npd基因丰度与微生物量氮含量呈显著正相关关系(P=0.015,n=10),而与铵态氮、硝态氮总量之间无显著相关关系(P=0.108,n=10)。稻草还田量通过影响土壤病毒和细菌的α多样性,间接影响细菌携带的ncd2/npd基因的相对丰度,最终对有机氮转化过程产生重要影响。
-
图 1 不同稻草还田量处理晚稻分蘖期和成熟期土壤前10位病毒科的相对丰度
注:CK为稻草不还田,S为全部稻草还田(单季稻草还田量6400 kg/hm2),S1、S2、S3处理的稻草还田量为全部稻草还田的1.1、1.2、1.3倍(单季稻草还田量分别为7075、7750、8425 kg/hm2)。柱中不同小写字母表示处理间差异显著(P<0.05).
Figure 1. The relative abundance of top 10 soil virus families in the paddy soil at tillering and maturity stages of late rice receiving different rice straw incorporation rates
Note: CK is the straw not returned, S is the whole straw returned (the amount of straw returned to the field in a single season is 6400 kg/hm2), and the amount of straw returned to the field in S1, S2 and S3 is 1.1, 1.2 and 1.3 times that of all the straw returned (the amount of straw returned to the field in a single season are 7075, 7750 and 8425 kg/hm2, respectively). Different lowercase letters in the bar indicate significant difference among treatments (P<0.05).
图 2 稻草还田下土壤氮循环相关的病毒contigs丰度Bubble图
注:CK为稻草不还田,S为全部稻草还田(单季稻草还田量6400 kg/hm2),S1、S2、S3处理的稻草还田量为全部稻草还田的1.1、1.2、1.3倍(单季稻草还田量分别为7075、7750、8425 kg/hm2)。图中实心圆大小表示稻草还田下土壤氮循环相关病毒contigs的丰度(基于RPKM值)大小,不同颜色代表不同病毒contig。
Figure 2. Bubble diagram of virus contigs abundance associated with soil nitrogen cycling under straw return
Note: CK is the straw not returned, S is all the straw returned (the amount of straw returned to the field in a single season is 6400 kg/hm2), and the amount of straw returned to the field in S1, S2 and S3 is 1.1, 1.2 and 1.3 times that of all straws returned (the amount of straw returned to the field in a single season are 7075, 7750 and 8425 kg/hm2, respectively). The size of the circle in the figure represents the abundance of virus contigs related to soil nitrogen cycling under rice straw return (based on RPKM values), and different colors represent different virus contigs.
图 3 不同处理下水稻分蘖期与成熟期土壤细菌门相对丰度柱状堆叠图
注:CK为稻草不还田,S为全部稻草还田(单季稻草还田量6400 kg/hm2),S1、S2、S3处理的稻草还田量为全部稻草还田的1.1、1.2、1.3倍(单季稻草还田量分别为7075、7750、8425 kg/hm2)。
Figure 3. Histogram of relative abundance of soil bacteria at tillering and maturity stages under different treatments
Note: CK is the straw not returned, S is all the straw returned (the amount of straw returned to the field in a single season is 6400 kg/hm2), and the amount of straw returned to the field in S1, S2 and S3 is 1.1, 1.2 and 1.3 times that of all straws returned (the amount of straw returned to the field in a single season are 7075, 7750 and 8425 kg/hm2, respectively).
图 5 水稻分蘖期与成熟期土壤细菌和病毒所含的有机氮转化基因丰度
注:CK为稻草不还田,S为全部稻草还田(单季稻草还田量6400 kg/hm2),S1、S2、S3处理的稻草还田量为全部稻草还田的1.1、1.2、1.3倍(单季稻草还田量分别为7075、7750、8425 kg/hm2)。
Figure 5. Abundance of organic nitrogen transformation genes contained in soil bacteria and viruses at tillering and maturity stages of rice
Note: CK is the straw not returned, S is all the straw returned (the amount of straw returned to the field in a single season is 6400 kg/hm2), and the amount of straw returned to the field in S1, S2 and S3 is 1.1, 1.2 and 1.3 times that of all straws returned (the amount of straw returned to the field in a single season are 7075 kg/hm2, 7750 kg/hm2, 8425 kg/hm2, respectively).
图 7 偏最小二乘路径模型
注:箭头旁数字表示路径系数;红色与蓝色箭头分别表示变量之间正负相关关系。模型可靠性的拟合优度为0.34。**—P<0.01; ***—P<0.001。
Figure 7. Partial least squares path model
Note: The numbers next to the arrows indicate the path factor; the red and blue arrows indicate positive and negative correlations between variables, respectively. The goodness of fit of the model is 0.34. **—P<0.01; ***—P<0.001.
表 1 各处理早稻和晚稻化肥施用量(kg/hm2)
Table 1 Chemical fertilizer input in each experimental treatment for early and late rice
处理
Treatment总计 Total 早稻 Early rice 晚稻 Late rice N P2O5 K2O N P2O5 K2O N P2O5 K2O CK 310.5 94.5 270.0 155.3 47.3 135.0 155.3 47.3 135.0 S1 310.5 94.5 243.0 155.3 47.3 121.5 155.3 47.3 121.5 S2 310.5 94.5 216.0 155.3 47.3 108.0 155.3 47.3 108.0 S3 310.5 94.5 189.0 155.3 47.3 94.5 155.3 47.3 94.5 S 310.5 94.5 270.0 155.3 47.3 135.0 155.3 47.3 135.0 表 2 病毒辅助代谢基因
Table 2 Viral auxiliary metabolism genes
KO编号 KO number 基因名称 Gene name 基因 Gene 基因详情 Gene description K00558 DNMT1 / dcm M.CK3_contig_11608-cat_2_4
M.S1.1_contig_7575-cat_1_14
M.S1.1_contig_8731-cat_2_2
M.S1.2_contig_12267-cat_2_8
M.S1.2_contig_9944-cat_1_46
M.S2.1_contig_1140-cat_2_16
M.S2.2_contig_897-cat_2_2
M.S3.1_contig_17094-cat_1_43
M.S3.2_contig_28456-cat_2_10
M.S3.3_contig_151241-cat_2_10
M.S3.3_contig_173896-cat_1_4
M.S2_contig_35584-cat_2_18
M.S3_contig_14916-cat_2_3
M.S3_contig_19124-cat_1_13
M.S3_contig_43849-cat_2_9
T.CK2_contig_11671-cat_2_13
T.CK3_contig_9479-cat_1_38
T.S1.1_contig_6392-cat_2_4
T.S1.1_contig_8281-cat_1_92
T.S1.2_contig_2737-cat_1_66
T.S1.2_contig_5126-cat_1_23
T.S2.3_contig_4688-cat_2_10
T.S3.2_contig_16590-cat_1_50
T.S3.3_contig_12427-cat_1_15
T.S3.3_contig_12963-cat_1_33
T.S3.3_contig_8555-cat_2_2DNA(胞嘧啶-5-)-甲基转移酶
DNA (cytosine-5-)-methyltransferaseK01652 ilvB / ilvG / ilvI M.S3.3_contig_182589-cat_2_4
M.S3.3_contig_182589-cat_2_4乙酰乳酸合酶Ⅰ/Ⅱ/Ⅲ大亚基
Acetolactate synthase Ⅰ/Ⅱ/Ⅲ large subunit -
[1] Jansson J K, Wu R N. Soil viral diversity, ecology and climate change[J]. Nature Reviews. Microbiology, 2022, 21(5): 296−311.
[2] 王光华, 刘俊杰, 朱冬, 等. 土壤病毒的研究进展与挑战[J]. 土壤学报, 2020, 57(6): 1319−1332. DOI: 10.11766/trxb202004280203 Wang G H, Liu J J, Zhu D, et al. Review of researches on viruses in soil−advancement and challenges[J]. Acta Pedologica Sinica, 2020, 57(6): 1319−1332. DOI: 10.11766/trxb202004280203
[3] Simon R B J E. Diversity in the soil virosphere: To infinity and beyond?[J]. Trends in Microbiology, 2022, 30(11): 1025−1035. DOI: 10.1016/j.tim.2022.05.003
[4] Graham B E, Camargo P A, Wu R, et al. A global atlas of soil viruses reveals unexplored biodiversity and potential biogeochemical impacts[J]. Nature Microbiology, 2024, 9(7): 1873−1883. DOI: 10.1038/s41564-024-01686-x
[5] Hu L, Hu L, Song C D, et al. Response of soil viral communities to land use changes[J]. Nature Communications, 2022, 13(1): 6027−6040. DOI: 10.1038/s41467-022-33771-2
[6] Christian S, Katerina E, Yuan M T, et al. Spatial turnover of soil viral populations and genotypes overlain by cohesive responses to moisture in grasslands[J]. Proceedings of the National Academy of Sciences of the United States of America, 2022, 119(45): 119−130.
[7] Wu H M, Yan W K, Wu H M, et al. Consecutive monoculture regimes differently affected the diversity of the rhizosphere soil viral community and accumulated soil-borne plant viruses[J]. Agriculture, Ecosystems and Environment, 2022, 337: 1−9.
[8] Liang X L, Radosevich M, DeBruyn J M, et al. Incorporating viruses into soil ecology: A new dimension to understand biogeochemical cycling[J]. Critical Reviews in Environmental Science and Technology, 2024, 54(2): 117−137. DOI: 10.1080/10643389.2023.2223123
[9] 吴汉卿, 阮楚晋, 韩苗, 王钢. 土壤病毒之奥秘: 研究进展、挑战及未来展望[J]. 微生物学报, 2024, 64(6): 1824−1847. Wu H Q, Ruan C J, Han M, Wang G. Mystery of soil viruses: Advances, challenges, and perspectives[J]. Acta Microbiologica Sinica, 2024, 64(6): 1824−1847.
[10] Zhu Y, Zhang Y, Yan S, et al. Viral community structure and functional potential vary with lifestyle and altitude in soils of Mt. Everest[J]. Environment International, 2023, 178: 108055−108065. DOI: 10.1016/j.envint.2023.108055
[11] Jin M, Guo X, Zhang R, et al. Diversities and potential biogeochemical impacts of mangrove soil viruses[J]. Microbiome, 2019, 7(1): 58−73. DOI: 10.1186/s40168-019-0675-9
[12] Emerson J B, Roux S, Brum J R, et al. Host-linked soil viral ecology along a permafrost thaw gradient[J]. Nature Microbiology, 2018, 3(8): 870−880. DOI: 10.1038/s41564-018-0190-y
[13] Huang X, Zhou Z C, Liu H Y, et al. Soil nutrient conditions alter viral lifestyle strategy and potential function in phosphorous and nitrogen metabolisms[J]. Soil Biology and Biochemistry, 2024, 189: 109279−109291. DOI: 10.1016/j.soilbio.2023.109279
[14] Han L L, Yu D T, Bi L, et al. Distribution of soil viruses across China and their potential role in phosphorous metabolism[J]. Environmental Microbiome, 2022, 17(1): 6−17. DOI: 10.1186/s40793-022-00401-9
[15] 郭俊杰, 朱晨, 刘文波, 等. 不同施肥模式对土壤氮循环功能微生物的影响[J]. 植物营养与肥料学报, 2021, 27(5): 751−759. DOI: 10.11674/zwyf.20504 Guo J J, Zhu C, Liu W B, et al. Effects of different fertilization patterns on soil nitrogen cycling functional microorganisms[J]. Journal of Plant Nutrition and Fertilizers, 2021, 27(5): 751−759. DOI: 10.11674/zwyf.20504
[16] 王彬浩, 吴愉萍, 陈彦博, 等. 秸秆还田对土壤微生物功能基因和功能类群的影响[J]. 浙江大学学报(农业与生命科学版), 2024, 50(5): 771−784. Wang B H, Wu Y P, Chen Y B, et al. Effects of straw returning on soil microbial functional genes and functional groups[J]. Journal of Zhejiang University (Agriculture and Life Sciences), 2024, 50(5): 771−784.
[17] 鲍士旦. 土壤农化分析[M]. 北京: 中国农业出版社, 2000. Bao S D. Soil and agricultural chemical analysis[M]. Beijing: China Agriculture Press, 2000.
[18] 杨叶华, 黄兴成, 朱华清, 等. 长期有机与无机肥配施的黄壤稻田土壤细菌群落结构特征[J]. 植物营养与肥料学报, 2022, 28(6): 984−992. DOI: 10.11674/zwyf.2021471 Yang Y H, Huang X C, Zhu H Q, et al. Characteristics of soil bacterial community structure in yellow soil paddy field with long-term combination of organic and inorganic fertilizers[J]. Journal of Plant Nutrition and Fertilizers, 2022, 28(6): 984−992. DOI: 10.11674/zwyf.2021471
[19] 邹湘, 易博, 张奇春, 等. 长期施肥对稻田土壤微生物群落结构及氮循环功能微生物数量的影响[J]. 植物营养与肥料学报, 2020, 26(12): 2158−2167. DOI: 10.11674/zwyf.20418 Zou X, Yi B, Zhang Q C, et al. Effects of long-term fertilization on soil microbial community structure and the number of microorganisms with nitrogen cycling function in paddy field[J]. Journal of Plant Nutrition and Fertilizers, 2020, 26(12): 2158−2167. DOI: 10.11674/zwyf.20418
[20] 唐治喜, 高菊生, 宋阿琳, 等. 用宏基因组学方法研究绿肥对水稻根际微生物磷循环功能基因的影响[J]. 植物营养与肥料学报, 2020, 26(9): 1578−1590. DOI: 10.11674/zwyf.20052 Tang Z X, Gao J S, Song A L, et al. Metagenomics was used to study the effect of green manure on the functional genes of phosphorus cycling in rice rhizosphere microorganisms[J]. Journal of Plant Nutrition and Fertilizers, 2020, 26(9): 1578−1590. DOI: 10.11674/zwyf.20052
[21] 祁慧鹓, 郑晓璇, 孙明明, 等. 土壤宏病毒组的研究方法与进展[J]. 土壤学报, 2021, 58(3): 568−577. DOI: 10.11766/trxb202008210474 Qi H Y, Zheng X X, Sun M M, et al. Review in the soil virus metagenome analytical methods and progress[J]. Acta Pedologica Sinica, 2021, 58(3): 568−577. DOI: 10.11766/trxb202008210474
[22] Yan Y T, Yu D T, Han L L, et al. Diversity and potential interactions of soil viruses and host bacteria under different land use patterns[J]. Forests, 2023, 14(2): 342−356. DOI: 10.3390/f14020342
[23] Wan Y J, Liu Y, Wu Y X, et al. Heterogeneity of soil bacterial and bacteriophage communities in three rice agroecosystems and potential impacts of bacteriophage on nutrient cycling[J]. Environmental Microbiome, 2022, 17(1): 1−17. DOI: 10.1186/s40793-021-00396-9
[24] Paze-Espino D, Eloe-Fadrsh E A, Pavlopoulos G A, et al. Uncovering Earth’s virome[J]. Nature, 2016, 536: 25−30.
[25] Liang X L, Zhang Y Y, Wommack E K, et al. Lysogenic reproductive strategies of viral communities vary with soil depth and are correlated with bacterial diversity[J]. Soil Biology and Biochemistry, 2020, 144: 107767−107778. DOI: 10.1016/j.soilbio.2020.107767
[26] 刘泽锟, 黄士轩, 杨美艳. 稻田土壤中病毒多样性研究[J]. 土壤学报, 2024, 61(6): 1703−1713. DOI: 10.11766/trxb202306130230 Liu Z K, Huang S X, Yang M Y. Virus diversity studies in paddy soils[J]. Acta Pedologica Sinica, 2024, 61(6): 1703−1713. DOI: 10.11766/trxb202306130230
[27] 王亚娇. 我国主要稻区土壤微生物和病毒多样性及冬小麦休耕对真菌群落和病害发生的影响[D]. 北京: 中国农业科学院博士学位论文, 2022. Wang Y J. Analysis of microbial and viral diversity in paddy soil and effects of long-term wheat fallow on soil fungal diversity and fungal diseases[D]. Beijing: PhD Dissertation of Chinese Academy of Agricultural Sciences, 2022.
[28] Han L L, Yu D T, Zhang L M, et al. Genetic and functional diversity of ubiquitous DNA viruses in selected Chinese agricultural soils[J]. Scientific Reports, 2017, 7: 45142−45153. DOI: 10.1038/srep45142
[29] Gao R C, Ma B, Hu M, et al. Ecological drivers and potential functions of viral communities in flooded arsenic-contaminated paddy soils[J]. Science of the Total Environment, 2023, 872: 162289−162297. DOI: 10.1016/j.scitotenv.2023.162289
[30] Yu M S, Zhang M H, Zheng R Y, et al. Diversity and potential host-interactions of viruses inhabiting deep-sea seamount sediments[J]. Nature Communications, 2024, 15(1): 3228−3245. DOI: 10.1038/s41467-024-47600-1
[31] Rivera N F, Ángel M G, Diana L, et al. Bacterial composition and diversity in deep-sea sediments from the Southern Colombian Caribbean Sea[J]. Diversity, 2020, 13(1): 10−23. DOI: 10.3390/d13010010
[32] 张全国. 病毒及其生态功能[J]. 生命科学, 2014, 26(2): 107−111. Zhang Q G. Viruses and their ecological effects[J]. Chinese Bulletin of Life Sciences, 2014, 26(2): 107−111.
[33] Tu Q, He Z, Wu L, et al. Metagenomic reconstruction of nitrogen cycling pathways in a CO2-enriched grassland ecosystem[J]. Soil Biology and Biochemistry, 2017, 106: 99−108. DOI: 10.1016/j.soilbio.2016.12.017
[34] Gazitúa M C, Vik D R, Gregory A C, et al. Potential virus-mediated nitrogen cycling in oxygen-depleted oceanic waters[J]. The ISME Journal, 2021, 15(4): 981−998. DOI: 10.1038/s41396-020-00825-6
[35] Roux S, Hawley A K, Torres B M, et al. Ecology and evolution of viruses infecting uncultivated SUP05 bacteria as revealed by single-cell- and meta-genomics[J]. eLife, 2014, 3: 1−20.
[36] 王美琦, 刘银双, 黄亚丽, 等. 秸秆还田对土壤微生态环境影响的研究进展[J]. 微生物学通报, 2022, 49(2): 807−816. Wang M Q, Liu Y S, Huang Y L, et al. Research progress on effects of straw incorporation on soil micro-ecological environment[J]. Microbiology China, 2022, 49(2): 807−816.
[37] Dyksma S, Lenk S, Sawicka J E, et al. Uncultured gammaproteobacteria and desulfobacteraceae account for major acetate assimilation in a coastal marine sediment[J]. Frontiers in Microbiology, 2018, 9: 3124−3133. DOI: 10.3389/fmicb.2018.03124



 下载:
下载: